| |
| Med Sci (Paris). 2010 December; 26(12): 1035–1037. Published online 2010 December 15. doi: 10.1051/medsci/201026121035.Déplétion des nucléosomes dans les régions promotrices Conséquences sur l’efficacité de l’activation transcriptionnelle Lu Bai1 and Gilles Charvin2,3,4* 1Center for studies in physics and biology, The Rockefeller University, New York, NY 10065, États-Unis 2Laboratoire Joliot-Curie, École normale supérieure, 46, allée d’Italie, 69364 Lyon Cedex 07 3Laboratoire de physique, École normale supérieure, Lyon 4Université de Lyon, France MeSH keywords: Aminopeptidases, physiologie, Animaux, Cycle cellulaire, Protéines de liaison à l'ADN, Dipeptidyl-Peptidases and Tripeptidyl-Peptidases, Drosophila melanogaster, génétique, Cellules eucaryotes, cytologie, Régulation de l'expression des gènes, Humains, Modèles génétiques, Nucléosomes, Régions promotrices (génétique), Saccharomyces cerevisiae, Protéines de Saccharomyces cerevisiae, Protéases à sérine, Facteurs de transcription, Activation de la transcription |
L’activation de la transcription d’un gène nécessite généralement l’accrochage d’activateurs spécifiques dans sa région promotrice. Pour un activateur, trouver les quelques sites d’accrochage dans le génome entier paraît déjà être une tâche immense. Dans les cellules eucaryotes, les activateurs doivent également faire face au fait qu’une large fraction (environ 80 % chez S. cerevisiae) de l’ADN génomique se présente sous la forme de chromatine, structure nucléoprotéique qui consiste en une répétition de nucléosomes, séquences d’ADN d’environ 147 paires de base enroulées autour d’un noyau d’histones. À cause des interactions très fortes entre l’ADN et les histones [
1], il est généralement admis que les nucléosomes représentent a priori une barrière pour l’accès des activateurs transcriptionnels à leurs sites d’accrochage respectifs. Ainsi, de nombreux promoteurs chez la levure, la drosophile et l’homme contiennent des séquences d’ADN dépourvues de nucléosomes (nucleosome depleted regions ou NDR), ce qui permet d’éviter la compétition entre activateur et histones vis-à-vis de l’accrochage sur l’ADN [
2–
6]. Cependant, plusieurs études ont montré que les NDR ne sont pas strictement nécessaires à l’accrochage d’un activateur. Ainsi, Gal4, qui est l’activateur des gènes GAL chez S. cerevisiae, peut reconnaître ses sites d’accrochage même s’ils sont masqués par des nucléosomes, et ainsi activer correctement la transcription [
7–
10]. En conséquence, quelle est donc la fonction des NDR au niveau des promoteurs, sachant que la déplétion de nucléosomes représente un coût énergétique important ? Pourquoi les NDR sont-il si abondants et très conservés dans les promoteurs eucaryotes ? |
Présence de nucléosomes et activation de la transcription Dans une étude récente [
11], nous avons apporté un éclairage nouveau sur ces questions. Dans la plupart des études précédentes, les données transcriptionnelles étaient exprimées comme la moyenne de mesures faites dans des populations de cellules. Au contraire, nous avons travaillé sur des cellules individuelles de levures et mesuré in vivo, dans des expériences de vidéomicroscopie de fluorescence, la transcription de gènes spécifiques en fonction du temps [
12–
14]. Deux promoteurs de gènes régulés au cours du cycle cellulaire, CLN2 (une cycline active lors de la transition G1/S) et HO
1
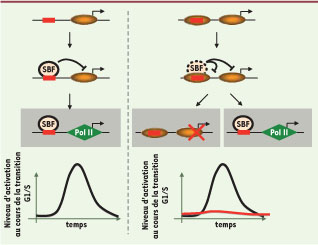
, ont été sélectionnés pour étudier l’influence des NDR sur l’activation transcriptionnelle. Différentes combinaisons des domaines, parfois mutés, de ces promoteurs ont été clonées dans des plasmides pour diriger l’expression du gène codant la GFP (green fluorescent protein) et intégrées dans la levure. Cette stratégie, qui permet de suivre la dynamique du signal de fluorescence de cellules individuelles au cours du temps, nous a permis de quantifier la variabilité d’une cellule à une autre de l’activation de ces gènes et de mettre en évidence des effets de mémoire, propriétés qui sont difficiles, sinon impossibles, à observer avec d’autres techniques. La transcription de CLN2 et HO est sous le contrôle d’un même activateur appelé SBF (Swi4/Swi6 cell cycle box binding factor) qui se fixe sur plusieurs sites dans des NDR de leurs régions promotrices. Dans une souche sauvage, il en résulte une activation transcriptionnelle très efficace et reproductible lors de la transition G1/S du cycle cellulaire. A contrario, si l’on place ces sites de fixation dans une région où ils sont masqués par un nucléosome, la transcription devient très erratique, suivant une logique de « tout ou rien ». En suivant les divisions successives d’une même cellule, une activation presque équivalente à celle de la souche sauvage peut être observée pour certains cycles cellulaires, alors que pour d’autres cycles, aucune activité n’est détectable, comme le schématise la Figure 1. En d’autres termes, la présence d’un nucléosome au niveau du site de fixation diminue la probabilité d’activer la transcription à chaque cycle mais ne change pas la force du promoteur si celui-ci est activé. Le caractère très variable de la transcription mesurée dans ce cas est cohérent avec la corrélation observée à l’échelle du génome entre la densité en nucléosomes sur les sites de fixation et la variabilité (« bruit ») transcriptionnelle [
15-
17].  | Figure 1
Schéma de la séquence d’activation transcriptionnelle par SBF, avec des sites de fixations localisés dans une NDR (à gauche) ou dans les nucléosomes (à droite). La flèche représente le site de démarrage de la transcription, les ellipses brunes les nucléosomes, et les rectangles rouges les sites de fixation de SBF. SBF s’accroche sur les sites de liaison localisés dans les NDR et assure une transcription efficace à chaque cycle cellulaire (à gauche). Au contraire, la localisation des sites de fixation dans les nucléosomes entraîne un dysfonctionnement partiel de l’activation du promoteur au cours de certains cycles cellulaires (à droite). Courbes en bas : dynamique d’activation du promoteur. À gauche, la présence du site de liaison dans une NDR rend l’activation du promoteur efficace et reproductible à chaque transition G1/S du cycle cellulaire pour toutes les cellules (la courbe noire représente le signal observé à l’aide d’un rapporteur GFP). À droite, l’activation de la transcription lors de la transition G1/S est de type « toutou-rien » (courbe rouge ou noire), entraînant une variabilité importante entre les cellules. |
|
Influence des séquences promotrices dépourvues en nucléosomes sur la vie cellulaire Une autre observation intéressante est que cette activation de type « tout ou rien » présente un effet de mémoire. La probabilité d’observer l’activation des promoteurs lors d’un cycle cellulaire est plus élevée que la moyenne si l’activation a eu lieu au cycle précédent, et la durée caractéristique de mémoire est de l’ordre d’un cycle. Le mécanisme responsable de cet effet de mémoire demeure obscur, mais pourrait être lié à un phénomène d’héritabilité épigénétique de la configuration des nucléosomes. Quel est donc l’avantage procuré par les NDR dans les régions promotrices ? Quelques éléments de réponse peuvent être tirés de notre étude ainsi que d’autres travaux empruntés à la littérature. En premier lieu, l’activation à partir d’un site de fixation nucléosomal peut requérir une concentration plus élevée d’activateurs et dépendre davantage des complexes de remodelage de la chromatine [7–11]. Le coût lié à ces deux processus pourrait être bien supérieur à celui nécessaire à la déplétion de quelques nucléosomes. Ensuite, la compétition de l’accrochage entre l’activateur et le nucléosome pourrait retarder la réponse transcriptionnelle, une situation qui a été déjà été mise en évidence dans le cas de l’activation par Gal4 [10]. Enfin, comme mentionné plus haut pour les gènes régulés au cours du cycle cellulaire, un site d’accrochage masqué par des nucléosomes fait courir le risque d’une variabilité d’expression très accrue, ce qui pourrait avoir un effet délétère sur la robustesse des fonctions cellulaires impliquées. À l’appui de cette idée, nous avons en effet montré que remplacer le promoteur de CLN2, gène essentiel de la transition G1/S, par un variant sans NDR, entraîne une diminution de l’adaptation des cellules. |
Les auteurs déclarent n’avoir aucun confit d’intérêts concernant les données publiées dans cet article. |
Footnotes |
1. Luger K, Mäder AW, Richmond RK, et al. Crystal structure of the nucleosome core particle at 2.8 A resolution. Nature 1997; 389 : 251-60. 2. Lee W, Tillo D, Bray N, et al. A high-resolution atlas of nucleosome occupancy in yeast. Nat Genet 2007; 39 : 1235-44. 3. Mavrich TN, Ioshikhes IP, Venters BJ, et al. A barrier nucleosome model for statistical positioning of nucleosomes throughout the yeast genome. Genome Res 2008; 18 : 1073-83. 4. Mavrich TN, Jiang C, Ioshikhes IP, et al. Nucleosome organization in the Drosophila genome. Nature 2008; 453 : 358-62. 5. Schones DE, Cui K, Cuddapah S, et al. Dynamic regulation of nucleosome positioning in the human genome. Cell 2008; 132 : 887-98. 6. Yuan GC, Liu YJ, Dion MF, et al. Genome-scale identification of nucleosome positions in S. cerevisiae. Science 2005; 309 : 626-30. 7. Burns LG, Peterson CL. The yeast SWI-SNF complex facilitates binding of a transcriptional activator to nucleosomal sites in vivo. Mol Cell Biol 1997; 17 : 4811-9. 8. Morse RH. Nucleosome disruption by transcription factor binding in yeast. Science 1993; 262 : 1563-6. 9. Xu M, Simpson RT, Kladde MP. Gal4p-mediated chromatin remodeling depends on binding site position in nucleosomes but does not require DNA replication. Mol Cell Biol 1998; 18 : 1201-12. 10. Floer M, Wang X, Prabhu V, et al. A RSC/nucleosome complex determines chromatin architecture and facilitates activator binding. Cell 2010; 141 : 407-18. 11. Bai L, Charvin G, Siggia ED, Cross FR. Nucleosomedepleted regions in cell-cycle-regulated promoters ensure reliable gene expression in every cell cycle. Dev Cell 2010; 18 : 544-55. 12. Bean JM, Siggia ED, Cross FR. Coherence and timing of cell cycle start examined at single-cell resolution. Mol Cell 2006; 21 : 3-14. 13. Charvin G, Cross FR, Siggia ED. A microfluidic device for temporally controlled gene expression and longterm fluorescent imaging in unperturbed dividing yeast cells. PLoS One 2008; 3 : e1468. 14. Skotheim JM, Di Talia S, Siggia ED, Cross FR. Positive feedback of G1 cyclins ensures coherent cell cycle entry. Nature 2008; 454 : 291-6. 15. Choi JK, KimYJ. Intrinsic variability of gene expression encoded in nucleosome positioning sequences. Nat Genet 2009; 41 : 498-503. 16. Field Y, Kaplan N, Fondufe-Mittendorf Y, et al. Distinct modes of regulation by chromatin encoded through nucleosome positioning signals. PLoS Comput Biol 2008; 4 : e1000216. 17. Tirosh I, Barkai N. Two strategies for gene regulation by promoter nucleosomes. Genome Res 2008; 18 : 1084-91. |