| |
| Med Sci (Paris). 2010 June; 26(6-7): 641–646. Published online 2010 June 15. doi: 10.1051/medsci/2010266-7641.Idiosyncratiques et « omiques » peuvent-ils rimer ? Céline Narjoz,1,2* Philippe Beaune,1,2 and Isabelle de Waziers1 1Université Paris-Descartes et Inserm U775, 45, rue des Saints-Pères, 75006 Paris, France 2APHP, Hôpital européen Georges Pompidou |
Idiosyncratique signifie « spécifique à un individu ». On considère généralement que les toxicités idiosyncratiques sont des effets secondaires rares qui peuvent apparaître à dose thérapeutique et dont le mécanisme toxique est souvent mal connu. Trois types de réactions idiosyncratiques sont identifiés : (1) les réactions métaboliques ; (2) celles relayées par le système immunitaire (réactions auto-immunes, réactions d’hypersensibilité ou allergies médicamenteuses); (3) celles potentialisées par l’inflammation. Cependant, la définition des réactions idiosyncratiques reste controversée [
1] puisque les toxicologues excluent de la définition les réactions qui impliquent un effet pharmacologique connu du médicament, tandis que les allergologues excluent les réactions relayées par le système immunitaire, mais non celles qui résultent des propriétés pharmacologiques du médicament. Pour lever toute ambiguïté et proposer une définition simple et claire, nous avons pris en compte, dans cette revue, l’ensemble des réactions liées à des facteurs individuels, qu’elles soient métaboliques ou relayées par le système immunitaire. La prévention des toxicités idiosyncratiques constitue un enjeu médical puisque celles-ci correspondent à environ 6 à 10 % des effets secondaires des médicaments et qu’elles peuvent être responsables d’hépatites fulminantes, de toxidermies, de pneumopathies ou d’atteintes hématologiques, etc. Leur détection représente également un enjeu économique puisque ces toxicités sont mal détectées lors du développement du médicament et qu’elles conduisent, le plus souvent, à une restriction d’utilisation du médicament ou à son retrait du marché [1]. Ces réactions idiosyncratiques sont généralement imprévisibles. Les modèles animaux standard utilisés en pharmacotoxicologie ne permettent pas en général de prédire de tels effets. Par ailleurs, la taille des populations étudiées dans les essais cliniques est trop faible pour détecter des évènements survenant à de faibles fréquences. |
Les mécanismes physiopathologiques des réactions idiosyncratiques L’effet indésirable d’un médicament peut être dû à l’effet délétère sur une cible du médicament, à la génération d’un métabolite ou à la réaction de l’organisme comme une stimulation du système immunitaire. On peut distinguer principalement deux types d’idio syncrasie toxique : Les réactions idiosyncratiques métaboliques dues à des polymorphismes génétiques ou à des interactions métaboliques conduisant à la formation anormale de métabolites [
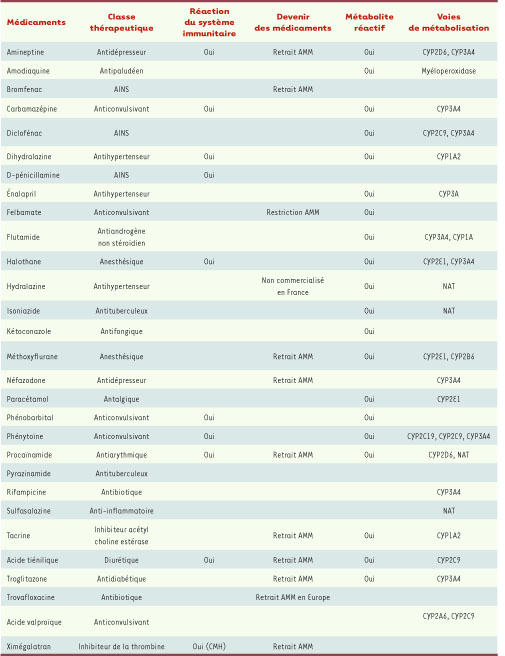
2] ou à l’accumulation du produit parent. La plupart des enzymes impliquées dans le métabolisme des médicaments sont connues pour être polymorphes (par exemple les cytochromes P450 [CYP], etc.)1. Les effets indésirables rares, détectés après la mise sur le marché d’un médicament, peuvent ainsi effectivement résulter d’une susceptibilité métabolique d’origine génétique (exemples dans le Tableau I). 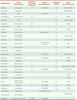 | Tableau I
Exemples de médicaments associés à des réactions idiosyncratiques. AINS : anti-inflammatoire non stéroïdien; AMM : autorisation de mise sur le marché; CYP : cytochrome P450; CMH : complexe majeur d’histocompatibilité. |
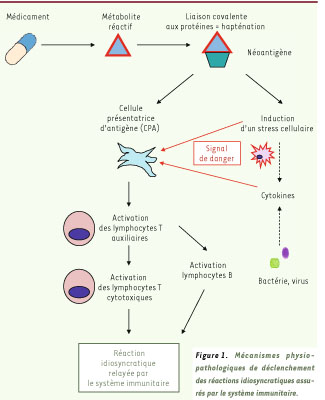
Les réactions idiosyncratiques relayées par le système immunitaire (Figure 1). Plusieurs hypothèses ont été émises pour expliquer le déclenchement de ces réponses immunitaires, mais aucun mécanisme n’a clairement été démontré. La métabolisation des médicaments induisant des réactions idiosyncratiques passe, le plus souvent, par la formation d’un métabolite réactif (haptène) qui se fixe sur l’enzyme qui l’a généré (adduit, néoantigène). Cet adduit sera alors considéré comme étranger à l’organisme et déclenchera une réponse immunitaire pouvant être dirigée contre le néoantigène mais parfois aussi contre l’enzyme native [
3,
4]. À cette théorie de l’haptène, P. Matzinger associe « l’hypothèse de danger » : elle suggère que le système immunitaire ne protège pas l’organisme contre une entité étrangère mais plutôt contre une entité dangereuse pour lui [
5]. Ainsi, l’activation du système immunitaire ne peut pas être uniquement déclenchée par la formation du néoantigène mais nécessite que celui-ci soit associé avec un signal de danger comme une infection ou un stress cellulaire. Un tel signal de danger pourrait également être induit par le médicament ou un de ses métabolites (par exemple nécrose ou apoptose cellulaire). Une troisième hypothèse, celle de l’interaction pharmacologique, suppose que certains médicaments pourraient activer directement le système immunitaire, sans métabolisation ni formation d’adduit protéique, en interagissant directement avec les molécules du complexe majeur d’histocompatibilité (CMH) [
6]. Cependant, le fait que ce type d’interaction pharmacologique puisse déclencher une réaction immunitaire n’a jamais été démontré. 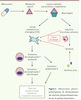 | Figure 1
Mécanismes physiopathologiques de déclenchement des réactions idiosyncratiques assurés par le système immunitaire.
|
Dans le cas des réactions immunoallergiques, les mécanismes, bien que variés, appartiennent à l’un des quatre types de réactions immunologiques classifiés par Gells et Coombs [
7] et font intervenir des effecteurs de l’immunité spécifique : soit des anticorps de type IgE (réactions immédiates); soit des lymphocytes T (réactions retardées) qui vont entraîner des lésions cutanées, des bronchospasmes et, dans les cas les plus graves, des chocs anaphylactiques. |
Sous ce terme sont regroupées des méthodes permettant d’analyser, sans a priori, l’ensemble des variations d’un système biologique. Ces technologies permettent l’étude des polymorphismes génétiques (génomique), l’expression des ARN messagers (transcriptomique), des protéines (protéomique), ou des métabolites produits (métabolomique) [
8]. Dans la suite de cette revue sera présentée une synthèse des premières utilisations des approches « omiques », essentiellement génomique et transcriptomique, pour la compréhension et la prédiction des réactions idiosyncratiques aux médicaments. Génomique et réactions idiosyncratiques Les SNP (single nucleotide polymorphism) constituent la forme la plus abondante (90 %) des variations génétiques du génome humain. Les puces à ADN permettent la caractérisation d’une partie ou de l’ensemble des SNP d’un individu. Récemment, leurs utilisations ont permis d’identifier des polymorphismes génétiques responsables d’une sensibilité particulière. Nous prendrons pour exemple les analyses réalisées au cours des réactions idiosyncratiques métaboliques et immunologiques dues à trois médicaments, la warfarine, l’abacavir et le ximélagatran. La warfarine est un anticoagulant oral largement utilisé dans le traitement des maladies thromboemboliques. Sa fenêtre thérapeutique est étroite. Il faut donc définir la dose à l’équilibre pour chaque patient afin d’obtenir une bonne efficacité thérapeutique avec un risque hémorragique minimum. Après des recherches à l’aide des approches gène-candidat, les polymorphismes des gènes codant pour le cytochrome P4502C9 (CYP2C9), la vitamine K époxyde réductase (VKORC1) [
29] et le cytochrome P4504F2 (CYP4F2) ont été significativement associés à la variabilité de réponse à la warfarine par des approches de génomique (genome-wide) [
9–
11]. Ainsi, les génotypes de CYP2C9, de VKORC1 et de CYP4F2, l’âge et le poids permettent d’expliquer 60,5 % de la variabilité interindividuelle de la dose à l’équilibre de la warfarine [
12]. Ces génotypes constituent donc des biomarqueurs utilisables par les cliniciens pour limiter les effets indésirables des anticoagulants anti-vitamine K. L’abacavir est un inhibiteur nucléosidique de la transcriptase inverse utilisé dans le traitement des patients infectés par le VIH (virus de l’immunodéficience humaine). Cependant, on observe une hypersensibilité idiosyncratique chez environ 5 % des patients traités, ce qui peut conduire, dans quelques cas, au décès. Des approches génomiques ont montré que l’allèle HLAB*5701 est associé à l’hypersensibilité à l’abacavir [
13–
15], et une étude prospective a prouvé l’utilité du génotypage de l’allèle HLA-B*5701 avant l’administration d’abacavir pour supprimer le risque de ces réactions [
16]. Enfin, le ximélagatran a été utilisé dans la prévention et le traitement de maladies thrombo-emboliques. Une étude génomique rétrospective cas-contrôle a été entreprise pour identifier les SNP qui pourraient être associés à l’élévation de l’alanine aminotransférase (ALAT) sérique, observée chez 6 à 13 % des patients traités sur une longue durée par ce médicament. Une très forte association entre l’élévation d’ALAT et les allèles HLA DRB*107 et DQA1*02 a été observée [
17]. Ces approches génomiques ont permis d’identifier les patients qui présentent un risque de développer des effets indésirables grâce à de nouveaux biomarqueurs, sans toujours apporter une explication au phénomène. Transcriptomique et réactions idiosyncratiques Les industries pharmaceutiques montrent un intérêt particulier vis-àvis des technologies « omiques » susceptibles d’identifier plus précocement un profil transcriptomique, protéomique ou métabonomique 2 spécifique d’une toxicité. L’identification d’une signature moléculaire caractéristique d’une toxicité permettrait d’identifier des biomarqueurs pertinents [
18,
19]. Seules quelques études transcriptomiques ont été publiées en ce qui concerne les composés impliqués dans des réactions idiosyncratiques. Nous présentons des études impliquant d’une part la D-pénicillamine, la carbamazépine, la phénytoïne et l’acide tiénilique qui provoquent des réactions idiosyncratiques relayées par le système immunitaire, et d’autre part la trovafloxacine et la troglitazone qui provoquent des hépatotoxicités idiosyncratiques potentialisées par un phénomène inflammatoire (d’origine virale, bactérienne, ou dû à une maladie inflammatoire). La D-pénicillamine est un antii nflammatoire qui provoque, chez certains patients, des réactions autoimmunes variées (syndrome lupique, myasthénies). Une signature transcriptomique, composée de trois gènes, KID-1 (kidney ischemia developpemen t), SGK (serum glucocorticoid-regulated kinase) et CINC (cytokine-induced neutrophil chemoattractant), a été identifiée à partir des profils d’expression d’ARN de foies de rats traités ou non par la Dpénicillamine. L’étude a démontré, par la suite, que ces trois gènes pouvaient servir de marqueurs sériques prédictifs du développement d’une réaction autoimmune chez les animaux traités [
20]. Les autres études ont mis en évidence des processus biologiques impliqués dans les phénomènes de toxicité idiosyncratique plutôt que des marqueurs prédictifs. Des études menées avec les anticonvulsivants (carbamazépine et phénytoïne) et avec l’acide tiénilique (AT) ont été réalisées dans le but de trouver un signal de danger précoce. Les animaux ont été sacrifiés dans les 6 heures qui ont suivi l’administration du traitement sans savoir si une réaction auto-immune se développait. Pour la carbamazépine et la phénytoïne, les gènes du stress oxydant pourraient être à l’origine du DRESS syndrome (drug reaction with eosinophilia and systemic symptoms) que ces médicaments peuvent provoquer [
21]. L’AT est un diurétique retiré du marché au début des années 1990 en raison des hépatites idiosyncratiques qu’il provoquait. Une étude transcriptomique réalisée à partir de foies de rats sacrifiés 6 heures et 24 heures après un traitement par l’AT a démontré que l’expression de gènes impliqués dans le stress oxydant, l’inflammation, et la régénération hépatique est déjà modifiée à 6 heures. Cette étude suggère que le stress oxydant, mais aussi un stress du réticulum endoplasmique, pourraient être à l’origine des signaux de danger produits par la cellule hépatique pour déclencher la réponse immunitaire. En revanche, 24 heures après le traitement, les gènes régulés sont plutôt ceux impliqués dans la régénération cellulaire, le cycle cellulaire et la réponse au stress. Ainsi, la cellule a mis en place des mécanismes de défense, de réparation et d’adaptation au stress [
22]. La trovafloxacine (TVX) est un antibiotique retiré du marché parce qu’il provoquait des hépatotoxicités idiosyncratiques. La lévofloxacine (LVX) est un antibiotique de la même classe qui ne provoque pas ce type de réaction. Chez le rat, une inflammation induite par des doses non toxiques de LPS (lipopolysaccharide bactériens, endotoxine) administrées en association avec des doses non toxiques de TVX provoque une hépatotoxicité comparable à celle observée chez l’homme. Afin d’identifier les mécanismes à l’origine des réactions différentes, une étude transcriptomique a comparé l’expression des gènes hépatiques de rats traités soit par LPS + TVX soit par LPS + LVX [
23]. La principale différence observée est une augmentation de l’expression de gènes codant pour des chimiokines (CINC-1, MIP-2 : macrophage-inflammatory protein-2) et des cytokines (GM-CSF : granulocyte macrophage colony-stimulating factor, IL(interleukine)-6) uniquement chez les rats LPS + TVX. Ces augmentations entraîneraient une accumulation des neutrophiles responsables de l’hépatotoxicité. Avec une approche similaire, les mêmes effets ont été observés chez des souris traitées par LPS + TVX [
24], et il a été montré que l’IL-18 et l’IFN-γ (interféron-γ) jouaient un rôle crucial dans l’hépatotoxicité [
25]. Cette approche identifie donc des pistes mécanistiques et des biomarqueurs de la toxicité chez l’homme. Par ailleurs, un modèle in vitro a été validé pour la troglitazone (TGZ), un agoniste de PPARγ (peroxisome proliferator-activated receptor) utilisé dans le traitement du diabète de type 2 qui induit des hépatotoxicités idiosyncratiques. Lors de leur préparation, les hépatocytes isolés sont soumis à une réponse proinflammatoire qui pourrait mimer le traitement par LPS des modèles in vivo. Des profils transcriptomiques différents ont été identifiés lors de l’addition aux cultures primaires d’hépatocytes humains de TGZ (composé toxique) et de rosiglitazone et pioglitazone (composés non toxiques) [
26]. Une étude récente a consisté à stimuler des cellules hépatiques (hépatocytes primaires humains, hépatocytes de rats, lignée d’hépatome humain HepG2) par un cocktail cytokinique (IFN γ, TNF, IL-1α, IL-6). Une cytotoxicité a été ensuite observée lors d’un traitement par des médicaments induisant des réactions idiosyncratiques (ranitidine, TVX, nimésulide), mais non lors d’un traitement par des médicaments non hépatotoxiques (famotidine, LVX, aspirine) [
27]. Les résultats obtenus dans ces différentes études transcriptomiques soulignent, entre autres, la participation de l’inflammation (cytokines, chimiokines) dans les réactions idiosyncratiques. Ainsi, un épisode inflammatoire suffisamment intense chez un patient pourrait abaisser le seuil de toxicité de certains médicaments [
28]. La réponse inflammatoire, qui constitue l’élément essentiel de la dynamique de l’immunité innée, serait en étroite connexion avec la réponse immunitaire (immunité acquise) et jouerait un rôle potentialisateur majeur dans les premières étapes de la réponse immunitaire. |
Le nombre d’études utilisant les technologies « omiques » est encore limité, mais les résultats déjà obtenus sont prometteurs. Le développement des puces SNP a permis l’identification, en génomique, de susceptibilités individuelles d’origine génétique comme en témoignent les différents exemples présentés. Des biomarqueurs prédictifs utilisables en clinique et recommandés par les autorités (CYP2C9, VKORC1 et warfarine; HLA B*5701 et abacavir) ont ainsi pu être identifiés. Une autre approche réside dans l’identification de profils transcriptomique, protéomique ou métabolique spécifiques des réactions idiosyncratiques. Les études essentiellement transcriptomiques menées jusqu’à présent ont mis en évidence des processus biologiques impliqués dans les mécanismes pathologiques des toxicités idiosyncratiques. Ainsi, des gènes impliqués dans le stress cellulaire, le stress oxydant ou l’inflammation sont probablement induits par la production de métabolites réactifs. Les différentes études présentées ci-dessus suggèrent, tout au moins pour certains médicaments, que les études transcriptomiques pourraient prédire les réactions idiosyncratiques dans les modèles utilisant soit des rats cotraités par LPS et le médicament à tester, soit des cellules hépatiques en culture. La diversité des mécanismes de toxicité à l’origine de réactions idiosyncratiques n’a pas permis la détermination d’une signature spécifique de ces effets. En revanche, l’utilisation des techniques « omiques » dans différents modèles in vivo et in vitro se révèle être d’un grand intérêt dans la compréhension des mécanismes de toxicité, dans l’identification de biomarqueurs prédictifs de la toxicité du médicament et, grâce à la génomique, de la susceptibilité individuelle. |
Les auteurs déclarent n’avoir aucun conflit d’intérêts concernant les données publiées dans cet article. |
Footnotes |
1. Uetrecht J. Idiosyncratic drug reactions: current understanding. Annu Rev Pharmacol Toxicol ; 47 : 513-39. 2. Weber WW. The molecular basis of hereditary acetylation polymorphisms. DrugMetab Dispos 1986 ; 14 : 377-81. 3. Bourdi M, Larrey D, Nataf J, et al. Anti-liver endoplasmic reticulum autoantibodies are directed against human cytochrome P-450IA2. A specific marker of dihydralazine-induced hepatitis. J Clin Invest 1990 ; 85 : 1967-73. 4. Park BK, Pirmohamed M, Kitteringham NR. Role of drug disposition in drug hypersensitivity: a chemical, molecular, and clinical perspective. Chem Res Toxicol 1998 ; 11 : 969-88. 5. Gallucci S, Lolkema M, Matzinger P. Natural adjuvants: endogenous activators of dendritic cells. Nat Med 1999 ; 5 : 1249-55. 6. Pichler WJ. Pharmacological interaction of drugs with antigen-specific immune receptors: the p-i concept. Curr Opin Allergy Clin Immunol 2002 ; 2 : 301-5. 7. Demoly P, Hillaire-Buys D, Raison-Peyron N, et al. Identifier et comprendre les allergies médicamenteuses. Med sci (Paris) 2003; 19 : 327-36. 8. Harrill AH, Rusyn I. Systems biology and functional genomics approaches for the identification of cellular responses to drug toxicity. Expert Opin Drug Metab Toxicol 2008; 4 : 1379-89. 9. Wadelius M, Chen LY, Lindh JD, et al. The largest prospective warfarin-treated cohort supports genetic forecasting. Blood 2009 ; 113 : 784-92. 10. Caldwell MD, Awad T, Johnson JA, et al. CYP4F2 genetic variant alters required warfarin dose. Blood ; 111 : 4106-12. 11. Takeuchi F, McGinnis R, Bourgeois S, et al. A genome-wide association study confirms VKORC1, CYP2C9, and CYP4F2 as principal genetic determinants of warfarin dose. PLoS Genet 2009 ; 5 : e1000433. 12. Borgiani P, Ciccacci C, Forte V, et al. CYP4F2 genetic variant (rs2108622) significantly contributes to warfarin dosing variability in the Italian population. Pharmacogenomics 2009 ; 10 : 261-6. 13. Hetherington S, McGuirk S, Powell G, et al. Hypersensitivity reactions during therapy with the nucleoside reverse transcriptase inhibitor abacavir. Clin Ther 2001 ; 23 : 1603-14. 14. Hughes AR, Spreen WR, Mosteller M, et al. Pharmacogenetics of hypersensitivity to abacavir: from PGx hypothesis to confirmation to clinical utility. Pharmacogenomics J 2008 ; 8 : 365-74. 15. Nelson MR, Bacanu SA, Mosteller M, et al. Genome-wide approaches to identify pharmacogenetic contributions to adverse drug reactions. Pharmacogenomics J 2009 ; 9 : 23-33. 16. Hughes S, Hughes A, Brothers C, et al. PREDICT-1 (CNA106030): the first powered, prospective trial of pharmacogenetic screening to reduce drug adverse events. Pharm Stat 2008 ; 7 : 121-9. 17. Kindmark A, Jawaid A, Harbron CG, et al. Genome-wide pharmacogenetic investigation of a hepatic adverse event without clinical signs of immunopathology suggests an underlying immune pathogenesis. Pharmacogenomics J 2008 ; 8 : 186-95. 18. Thome-Kromer B, Bonk I, Klatt M, et al. Toward the identification of liver toxicity markers: a proteome study in human cell culture and rats. Proteomics 2003 ; 3 : 1835-62. 19. Bandara LR, Kelly MD, Lock EA, Kennedy S. A correlation between a proteomic evaluation and conventional measurements in the assessment of renal proximal tubular toxicity. Toxicol Sci 2003 ; 73 : 195-206. 20. Séguin B, Boutros PC, Li X, Okey AB, Uetrecht JP. Gene expression profiling in a model of D-penicillamine-induced autoimmunity in the Brown Norway rat: predictive value of early signs of danger. Chem Res Toxicol 2005 ; 18 : 1 193-202. 21. Lu W, Li X, Uetrecht JP. Changes in gene expression induced by carbamazepine and phenytoin: testing the danger hypothesis. J Immunotoxicol 2008 ; 5 : 107-13. 22. Pacitto SR, Uetrecht JP, Boutros PC, Popovic M. Changes in gene expression induced by tienilic Acid and sulfamethoxazole: testing the danger hypothesis. J Immunotoxicol 2007 ; 4 : 253-66. 23. Waring JF, Liguori MJ, Luyendyk JP, et al. Microarray analysis of lipopolysaccharide potentiation of trovafloxacin-induced liver injury in rats suggests a role for proinflammatory chemokines and neutrophils. J Pharmacol Exp Ther 2006 ; 316 : 1080-7. 24. Shaw PJ, Hopfensperger MJ, Ganey PE, Roth RA. Lipopolysaccharide and trovafloxacin coexposure in mice causes idiosyncrasy-like liver injury dependent on tumor necrosis factor-alpha. Toxicol Sci 2007 ; 100 : 259-66. 25. Shaw PJ, Ditewig AC, Waring JF, et al. Coexposure of mice to trovafloxacin and lipopolysaccharide, a model of idiosyncratic hepatotoxicity, results in a unique gene expression profile and interferon gamma-dependent liver injury. Toxicol Sci 2009 ; 107 : 270-80. 26. Kier LD, Neft R, Tang L, et al. Applications of microarrays with toxicologically relevant genes (tox genes) for the evaluation of chemical toxicants in Sprague Dawley rats in vivo and human hepatocytes in vitro. Mutat Res 2004 ; 549 : 101-13. 27. Cosgrove BD, King BM, Hasan MA, et al. Synergistic drug-cytokine induction of hepatocellular death as an in vitro approach for the study of inflammation-associated idiosyncratic drug hepatotoxicity. Toxicol Appl Pharmacol 2009 ; 237 : 317-30. 28. Luyendyk JP, Maddox JF, Cosma GN, et al. Ranitidine treatment during a modest inflammatory response precipitates idiosyncrasy-like liver injury in rats. J Pharmacol Exp Ther 2003 ; 307 : 9-16. 29. Le Bonniec B. La cible de la warfarine identifiée. Med Sci (Paris) 2004 ; 20 : 512-4. |