| |
| Med Sci (Paris). 2010 June; 26(6-7): 564–566. Published online 2010 June 15. doi: 10.1051/medsci/2010266-7564.
ANRIL ou l’étrange histoire d’un grand ARN non codant Éric Pasmant,* Ingrid Laurendeau, Audrey Sabbagh, Béatrice Parfait, Michel Vidaud, Dominique Vidaud, and Ivan Bièche UMR745 Inserm, Université Paris Descartes, Faculté des sciences pharmaceutiques et biologiques, 4, avenue de l’Observatoire, 75006 Paris, France Service de biochimie et génétique moléculaire, Hôpital Beaujon, AP-HP, 100, boulevard du Général Leclerc, 92110 Clichy, France MeSH keywords: Maladies cardiovasculaires, génétique, Gènes suppresseurs de tumeur, Homéostasie, Humains, Tumeurs, ARN non traduit, physiologie, Protéine p14(ARF) suppresseur de tumeur |
Cluster p16/CDKN2A-p15/CDKN2B-p14/ARF et prédisposition aux cancers La prédisposition héréditaire au mélanome cutané malin est en partie liée à des mutations constitutionnelles du gène p16/CDKN2A (cyclin-dependent kinase inhibitor 2A), également appelé INK4a (inhibitor of cyclin-dependent kinase 4A). Ce gène code pour une protéine inhibitrice des CDK4 (cyclin-dependent kinase 4) et CDK6 qui régulent le cycle cellulaire. Un syndrome héréditaire associant des prédispositions au mélanome et à des tumeurs du système nerveux a été décrit dans de rares familles, définissant le syndrome mélanome-astrocytome (OMIM 155755). Nous avons eu l’occasion d’étudier une famille présentant un tel syndrome et d’identifier chez les membres atteints une large délétion constitutionnelle incluant l’ensemble des gènes p16/CDKN2A, p15/CDKN2B et p14/ARF (alternate reading frame, phase de lecture alternative du locus CDKN2A) de ce locus en 9p21.3, suggérant un « syndrome des gènes contigus » [
1]. |
ANRIL, un nouveau venu au locus p16/CDKN2A-p15/CDKN2B-p14/ARF
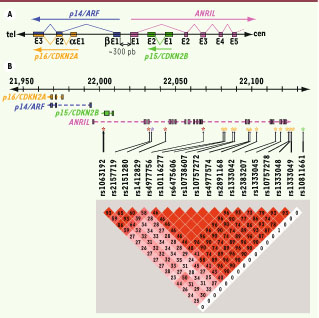
Cette étude nous a également permis d’identifier au sein de cette délétion constitutionnelle un grand ARN non codant que nous avons nommé ANRIL pour antisens noncoding RNA in the INK4 locus (OMIM 613149). Le gène ANRIL, constitué de 19 exons répartis sur 126 kb, est transcrit en un ARNm de 3 834 pb dans le sens opposé à celui des trois autres gènes du cluster (orientation anti-sens). ANRIL chevauche les deux exons de p15/CDKN2B au niveau de son intron 1 (Figure 1A). La proximité (~300 pb) des sites d’initiation de la transcription d’ANRIL et de p14/ARF ainsi que la corrélation positive de leurs taux d’expression dans des tissus humains normaux et tumoraux suggéraient une régulation transcriptionnelle coordonnée de ces deux gènes (Figure 1A). La récente mise en évidence de la fixation de la protéine régulatrice CTCF (CCCTC binding factor) au niveau des séquences promotrices de ces deux gènes a permis d’expliquer la corégulation de p14/ARF et d’ANRIL, pourtant orientés en sens opposé [
2]. CTCF est capable de se fixer sur des sites isolateurs de la chromatine1 et de moduler l’activité transcriptionnelle des régions adjacentes.  | Figure 1
Cluster des gènes p16/CDKN2A-p15/CDKN2B-p14/ARF-ANRIL. A. Organisation transcriptionnelle des gènes p16/CDKN2A, p15/CDKN2B, p14/ARF et ANRIL au locus 9p21.3. B. Localisation de 18 SNP du locus 9p21.3 utilisés dans les études de GWAS (génome humain hg19/ GRCh37). Les SNP en déséquilibre de liaison (DL) sont associés en blocs. Ces groupes distincts sont retrouvés comme marqueurs de susceptibilité à diverses maladies : gliome (astérisques rouges ; rs1063192, rs2157719, rs1412829 et rs4977756), carcinome basocellulaire (astérisque violet ; rs2151280), risque cardiovasculaire (nombreux SNP en fort déséquilibre de liaison dans la région 3’ d’ANRIL et marqués par un astérisque orange) et diabète de type 2 (astérisque vert; rs10811661). Les profils de DL entre les SNP du locus ont été évalués dans l’échantillon européen de HapMap. Le DL entre chaque paire de SNP, mesuré par l’indice r2, varie entre 0 et 100 % (100 % représentant un DL complet entre deux SNP). Plus la valeur r2 est élevée, plus la couleur des carrés est foncée. Il est à noter que le SNP rs10811661 associé avec le diabète de type 2 est totalement indépendant des autres SNP du locus (r2 < 1 %). |
|
Les études d’association génétique génome entier révèlent un lien entre ANRIL et diverses pathologies De manière très surprenante, et alors que le rôle d’ANRIL restait à élucider, des études d’association génétique génome entier ou GWAS (genome wide association study) portant sur diverses maladies multigéniques ont montré que les SNP (single nucleotide polymorphisms) les plus significativement associés à l’infarctus du myocarde, aux anévrismes intracrâniens mais aussi au diabète de type 2 étaient situés au niveau du gène ANRIL (Figure 1B) [
3,
4,
5]. Le lien entre ces maladies et le gène ANRIL fut confirmé dans un second temps par des études d’expression qui montrèrent une corrélation entre l’augmentation du risque cardiovasculaire et le taux de transcrits d’ANRIL dans les lymphocytes du sang périphérique [
6]. Ces études d’expression ont permis de confirmer la corégulation transcriptionnelle de p16/CDKN2A, p15/CDKN2B, p14/ARF et ANRIL, mais également d’appréhender la complexité de la régulation transcriptionnelle d’ANRIL avec l’identification d’au moins huit variants d’épissage [
7]. Parmi les quatre gènes du cluster, le taux d’expression d’ANRIL montrait la plus forte association aux événements cardiovasculaires [
8]. |
La question du lien physiopathologique entre ANRIL et le risque cardiovasculaire restait entière lorsque de nouvelles études d’association ont identifié ANRIL comme locus de susceptibilité à certains cancers incluant gliomes et carcinomes basocellulaires (Figure 1B) [
9]. Ces résultats étaient donc en accord avec l’étude princeps qui avait conduit à l’identification d’une délétion d’ANRIL au sein d’une famille associant une prédisposition aux mélanomes mais également aux tumeurs du système nerveux. La susceptibilité aux différentes maladies retrouvée par GWAS pourrait être conférée par des SNP distincts au niveau du gène ANRIL (Figure 1B) [
10]. Une possible explication du lien entre ANRIL et ces pathologies si diverses est venue d’une étude ayant trait au déclin des fonctions autoréplicatives des cellules souches au cours du vieillissement [
11]. N. Sharpless et R. DePinho ont en effet suggéré que la sénescence réplicative2 serait un mécanisme majeur à l’origine des pathologies liées au vieillissement, dont notamment l’athérosclérose, le diabète de type 2 et les cancers. Ainsi, l’implication d’ANRIL (du fait de sa position proche du locus p16/CDKN2A-p15/CDKN2B-p14/ARF et des résultats de l’étude GWAS) dans le vieillissement cellulaire pourrait apporter une séduisante hypothèse unificatrice expliquant l’association de divers facteurs de risques à cet ARN non codant. Des études fonctionnelles in vitro (lignées cellulaires) ou in vivo (modèles animaux) sont maintenant nécessaires pour prouver les rôles respectifs d’ANRIL dans la régulation du locus p16/CDKN2A-p15/CDKN2B-p14/ARF et dans les différents processus physiopathologiques que lui ont assigné les études GWAS. |
Les grands ARN non codants L’identification d’ANRIL souligne également le rôle régulateur sans doute sousestimé des grands ARN non codants alors que se développe intensément depuis quelques années l’étude d’une autre classe d’ARN non codants : les microARN [
12]. Les nouvelles techniques d’analyse génomique permettront sans doute d’établir un répertoire de ces grands (> 200 nt) ARN non codants (dont le nombre est estimé à plusieurs milliers) et de mieux comprendre leurs mécanismes d’action [
13]. De plus, il est troublant de constater que de nombreuses études GWAS révèlent des pics d’association dans des régions non codantes du génome, appelées « déserts de gènes », dont certains auteurs font l’hypothèse qu’ils pourraient abriter de grands ARN non codants [
14]. |
Les auteurs déclarent n’avoir aucun conflit d’intérêts concernant les données publiées dans cet article.
|
Footnotes |
1. Pasmant E, Laurendeau I, Héron D, et al. Characterization of a germ-line deletion, including the entire INK4/ARF locus, in a melanoma-neural system tumor family: identification of ANRIL, an antisense noncoding RNA whose expression coclusters with ARF. Cancer Res 2007; 67 : 3963-9. 2. Rodriguez C, Borgel J, Court F, et al. CTCF is a DNA methylation-sensitive positive regulator of the INK/ ARF locus. Biochem Biophys Res Commun 2010; 392 : 129-34. 3. McPherson R, Pertsemlidis A, Kavaslar N, et al. A common allele on chromosome 9 associated with coronary heart disease. Science 2007; 316 : 1488-91. 4. Helgadottir A, Thorleifsson G, Magnusson KP, et al. The same sequence variant on 9p21 associates with myocardial infarction, abdominal aortic aneurysm and intracranial aneurysm. Nat Genet 2008; 40 : 217-24. 5. Zeggini E, Weedon MN, Lindgren CM, et al. Replication of genome-wide association signals in UK samples reveals risk loci for type 2 diabetes. Science 2007; 316 : 1336-41. 6. Liu Y, Sanoff HK, Cho H, et al. INK4/ARF transcript expression is associated with chromosome 9p21 variants linked to atherosclerosis. PLoS One 2009; 4 : e5027. 7. Folkersen L, Kyriakou T, Goel A, et al. Relationship between CAD risk genotype in the chromosome 9p21 locus and gene expression. Identification of eight new ANRIL splice variants. PLoS One 2009; 4 : e7677. 8. Holdt LM, Beutner F, Scholz M, et al. ANRIL Expression is associated with atherosclerosis risk at chromosome 9p21. Arterioscler Thromb Vasc Biol 2010; 30 : 620-7. 9. Shete S, Hosking FJ, Robertson LB, et al. Genome-wide association study identifies five susceptibility loci for glioma. Nat Genet 2009; 41 : 899-904. 10. Broadbent HM, Peden JF, Lorkowski S, et al. Susceptibility to coronary artery disease and diabetes is encoded by distinct, tightly linked SNPs in the ANRIL locus on chromosome 9p. Hum Mol Genet 2008;17 : 806-14. 11. Sharpless NE, DePinho RA. How stem cells age and why this makes us grow old. Nat Rev Mol Cell Biol 2007;8 : 703-13. 12. Finoux AL, Chartrand P. Micro-ARN : oncogènes et suppresseurs de tumeurs. Med Sci (Paris) 2008; 24 : 1049-54. 13. Mercer TR, Dinger ME, Mattick JS. Long non-coding RNAs : insights into functions. Nat Rev Genet 2009; 10 : 155-9 14. Mattick JS. The genetic signatures of noncoding RNAs. PLoS Genet 2009; 5 : e1000459. 15. Fourel G, Gilson E. Empreinte parentale : insulateurs sur commande. Med Sci (Paris) 2001; 17 : 86-9. |