| |
| Med Sci (Paris). 2010 March; 26(3): 259–266. Published online 2010 March 15. doi: 10.1051/medsci/2010263259.« Attrape-moi si tu peux » Surveillance de l’intégrité structurale et fonctionnelle des ARN cellulaires eucaryotes Nathalie Leporé1 and Denis L.J. Lafontaine1,2* 1Fonds de la recherche scientifique (FRS-FNRS), Métabolisme de l’ARN, Institut de biologie et de médecine moléculaires (IBMM), Université Libre de Bruxelles (ULB), avenue des Professeurs Jeener et Brachet, 12, B-6041 Gosselies, Belgique 2Center for Microscopy and Molecular Imaging (CMMI), Académie Wallonie-Bruxelles, Charleroi-Gosselies, Belgique |
L’acide ribonucléique ou ARN est au cœur de l’expression des gènes et à la base de tous les processus biologiques. La cellule eucaryote synthétise ainsi des milliers de molécules d’ARN, appartenant à plusieurs dizaines de classes distinctes, chacune porteuse de fonctions et de structures spécialisées. Parmi leurs nombreuses propriétés, les ARN ont deux caractéristiques majeures : (1) ils ne sont jamais synthétisés avec leurs extrémités matures et comprennent parfois des séquences internes non codantes (ce qui implique de nombreuses étapes de maturation et offre l’opportunité de réguler leur synthèse à un niveau post-transcriptionnel) et (2) ils sont associés à des protéines avec lesquelles ils fonctionnent de façon très étroite, sous la forme de particules ribonucléoprotéiques, dites RNP. Générer des ARN matures fonctionnels à partir de précurseurs nécessite donc de très nombreuses étapes de maturation, dont la plupart débutent de façon cotranscriptionnelle. Ainsi, on distingue des étapes de clivage, de modification covalente, d’assemblage, de transport, etc. Chacune de ces étapes est une source potentielle d’erreurs. Accumuler des RNP défectueuses est à éviter car cela s’avérerait délétère pour la cellule. Par exemple des ARNm avec des erreurs ou des ribosomes infidèles généreraient des protéines avec des fautes ou tronquées. La cellule a donc sélectionné au cours de l’évolution des systèmes de surveillance ou de contrôle de qualité très performants, et partiellement redondants, dans le but de rapidement reconnaître et dégrader les RNP défectueuses. Plusieurs mécanismes ont ainsi été mis à jour et plusieurs facteurs trans de cette surveillance ont été identifiés. Le plus souvent, il s’agit d’acteurs également connus pour leurs activités dans la synthèse des extrémités matures des ARN et la dégradation. Les mécanismes de reconnaissance des RNP défectueuses, en revanche, restent quasi entièrement à déterminer. Dans cet article, nous passons en revue les acteurs principaux et les mécanismes de contrôle de qualité récemment mis à jour pour les trois classes majeures d’ARN cellulaires : les ARNm, les ARNt et les ARNr. |
Qu’est-ce que la surveillance ARN ? La surveillance est l’ensemble des mécanismes de contrôle de qualité qui ont été sélectionnés au cours de l’évolution pour permettre à la cellule d’éliminer rapidement ses ARN aberrants et surnuméraires. Deux types d’erreurs peuvent être introduites : des altérations en cis dans la séquence de l’ARN, liées à des dysfonctionnements de l’ARN polymérase ou à des stress génotoxiques (tels que l’exposition aux rayonnements UV, l’oxydation, l’alkylation, etc.) et des erreurs en trans, liées à un défaut d’assemblage, à la perte d’un facteur de maturation ou d’une protéine constitutive de la RNP. La surveillance implique deux étapes : l’identification du substrat aberrant, c’est-à-dire sa « stigmatisation » (par exemple par la polyadénylation de l’extrémité 3’-terminale de son constituant ARN ou l’ubiquitinylation de certaines protéines qui lui sont associées, voir ci-dessous) et sa dégradation. La dégradation implique des endoRNases et/ou des exoRNases (RNase pour ribonucléase) et peut être réalisée après l’adressage de la RNP défectueuse vers un sous-compartiment cellulaire spécialisé, tels que les corps de maturation cytoplasmiques (P-bodies) ou la vacuole/lysosome, enrichis respectivement en facteurs de maturation de l’ARN et en hydrolases non spécifiques. La surveillance permet également à la cellule de se débarrasser de RNP en fin de vie ou dont l’abondance est devenue inappropriée à la suite de l’adaptation aux conditions environnementales toujours changeantes (par exemple suite à un stress nutritionnel). |
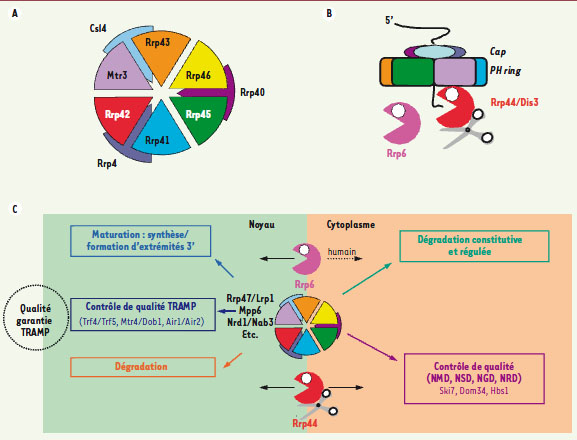
Les acteurs de la surveillance L’exosome à ARN et ses cofacteurs L’exosome à ARN est un complexe multiprotéique conservé au cours de l’évolution dont l’activité principale d’exoRNase 3’->5’ est impliquée dans la dégradation, la synthèse d’extrémités 3’-matures et la surveillance de la majorité des classes d’ARN cellulaires, ainsi que de certains ARN viraux [
1–
3]. Chez les eucaryotes, l’exosome est constitué d’un cœur de neuf sous-unités et d’une ou deux RNases associées (Rrp44/Dis3 et Rrp6) (Figure 1). Six des neuf sous-unités du cœur de l’exosome forment un anneau structural (qui constitue une plateforme de recrutement pour des RNases associées et des cofacteurs qui leur confèrent une spécificité) et sont homologues aux RNases bactériennes phosphorolytiques : les RNase PH et PNPase, et à l’exosome d’Archaea. Ces sous-unités ont toutefois perdu leur activité catalytique de dégradation de l’ARN chez l’homme pour une raison qui reste à élucider. Les RNases phosphorolytiques utilisent du phosphate inorganique comme cofacteur et produisent des nucléotides diphosphates. Les trois autres sous-unités du cœur de l’exosome sont des protéines de liaison à l’ARN de type S1 ou KH qui forment un « chapeau » qui connecte les sous-unités de l’anneau entre elles. Rrp44/Dis3 est homologue aux RNases II/R bactériennes et possède à la fois une activité exoRNase 3’->5’ et endoRNase [
4,
5]. L’activité endoRNase de Rrp44/Dis3 réside dans un domaine protéique dit de type PIN 1, et permet vraisemblablement de faciliter la progression de l’exosome sur des substrats hautement structurés. L’exosome fonctionne de concert avec de nombreux cofacteurs qui fournissent la spécificité de substrat et modulent sa fonction [ 1,
6] (Figure 1). Dans le noyau, l’exosome fonctionne avec Rrp6 (homologue aux RNases bactériennes T/D), une seconde sous-unité catalytique exoRNase 3’->5’ du complexe. Rrp44/Dis3 et Rrp6 sont des enzymes hydrolytiques qui, de façon remarquable, ont des propriétés biochimiques complémentaires. Les RNases hydrolytiques utilisent l’eau comme cofacteur et produisent des nucléotides monophosphates. Rrp44/Dis3 et Rrp6 fonctionnent différemment : la première pénètre son substrat et le digère de part en part, alors que Rrp6 entre et sort de son substrat à de multiples reprises. Rrp44/Dis3 dégrade aussi bien les ARN présentant, ou non, de nombreuses structures secondaires, par exemple, du type épingles à cheveux ou tige-boucle, mais est assez inefficace pour dégrader des ARN polyadénylés ou riches en séquences AU (établi in vitro). Rrp6, quant à elle, a une préférence marquée pour les substrats non structurés, les séquences polyadénylées ou riches en AU [
7]. De nombreux autres cofacteurs de l’exosome ont été caractérisés aussi bien dans le noyau que dans le cytoplasme. Dans le noyau, ceux-ci comprennent : une protéine de liaison à l’ARN double brin (Rrp47/Lrp1), une protéine de liaison à des séquences riches en pyrimidine (Mpp6), des protéines de liaison à des séquences spécifiques d’ARN (Nrd1 et Nab3) et le complexe de polyadénylation et de « discrimination de la norme » TRAMP (voir ci-dessous). Dans le cytoplasme, la protéine Ski7 (une GTPase homologue à certains facteurs traductionnels, voir légende de la Figure 2) constitue un cofacteur clef de recrutement de l’exosome.  | Figure 1.
Structure et fonction de l’exosome à ARN eucaryote. A-B. Représentation graphique de l’organisation structurale de l’exosome. A. Vue transversale. Chacune des sous-unités de la structure cœur de l’exosome est nommée. Les sous-unités du « chapeau » sont Csl4, Rrp40 et Rrp4 ; les autres sous-unités forment l’anneau. B. Vue de profil. Les deux activités RNases associées avec l’exosome (Rrp44/Dis3 et Rrp6) sont représentées. La position de Rrp6 au sein du complexe est actuellement inconnue. Les sous-unités du « chapeau » (cap) et de l’anneau (PH-ring) sont représentées. Le parcours de l’ARN à travers la cavité délimitée par l’anneau central est symbolisé. C. Les multiples fonctions de l’exosome et ses cofacteurs. Les différentes fonctions de l’exosome dans la synthèse, la dégradation et la surveillance des ARN et leur localisation sont résumées. La sous-unité exoRNase Rrp6 distingue les formes nucléaire (présence) et cytoplasmique (absence) de l’exosome chez la levure. Chez l’homme, Rrp6 serait présente dans le noyau et le cytoplasme. TRAMP : complexe de polyadénylation nucléaire (voir description dans le texte) ; NMD : nonsense-mediated decay détruit les ARNm contenant un codon stop dans un contexte peu favorable à la terminaison de la traduction ; NSD : nonstop mRNA decay élimine les ARNm dépourvus de codon stop ; NGD : no-go decay dégrade les ARNm qui induisent des pauses traductionnelles ; NRD : nonfunctional rRNA decay. |
 | Figure 2.
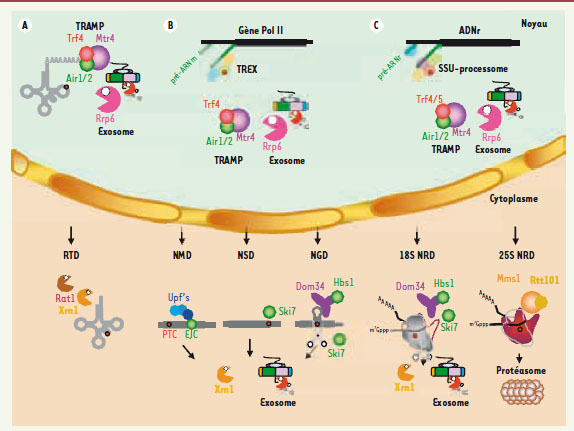
Les voies majeures de la surveillance ARN. Chacune des altérations en cis- (dans la séquence de l’ARN) ou en trans (liées à l’absence de liaison, ou à la perte, d’un facteur d’assemblage ou d’une protéine constitutive de la RNP) est représentée par un point rouge. A. ARNt noyau : les précurseurs d’ARNt hypomodifiés sont polyadénylés à leur extrémité 3’ par le complexe TRAMP4. Le complexe TRAMP et la queue de poly(A) agissent comme un signal de recrutement et de stimulation de l’exosome qui va mener à une digestion exoribonucléolytique 3’->5’ du substrat.Cytoplasme : les ARNt hypomodifiés sont dégradés par la voie du Rapid tRNA decay ou RTD qui implique leur digestion 5’->3’ par les exoRNases Xrn1 et Rat1. B. ARNm noyau : l’assemblage cotranscriptionnel d’une mRNP est symbolisé (gène en noir ; transcrit naissant en vert). Les défauts d’assemblage entraînent soit la séquestration des pré-mRNP dans des foci spatialement liés aux sites de transcription, soit la polyadénylation par TRAMP4 de leurs constituants ARN et leur dégradation rapide par l’exosome. TREX : transport-export.Cytoplasme : un ARNm est schématisé avec soit un stop prématuré (annoté PTC) dans la phase ouverte de lecture (cas du NMD), soit pas de stop (cas du NSD), soit une tige-boucle, symbolisant un ralentissement du ribosome dans sa progression (cas du NGD). Lors du NMD, un complexe de surveillance constitué des protéines Upf initie la dégradation menée aussi bien par Xrn1, suite au décoiffage (mais ne nécessitant pas de déadénylation préalable), que par l’exosome, suite à une déadénylation accélérée. Chez les mammifères, les protéines Upf sont assistées du complexe EJC pour exon junction complex, une signature de l’épissage, qui servirait de plateforme d’assemblage aux protéines Upf’s. C’est en partie la distance entre le PTC et l’EJC (mammifères) ou entre le PTC et la queue de poly(A) liée par la protéine Pab1 (levure), qui jouerait un rôle déterminant dans la reconnaissance du codon stop prématuré. Notons que chez les insectes, le NMD nécessite un clivage endonucléolytique initiateur, ce qui le rapproche d’un point de vue du mécanisme du NGD (voir ci-dessous). Dans le NSD, c’est Ski7, une protéine homologue respectivement aux facteurs d’élongation et de terminaison de la traduction eEF1A et eRF3 qui, agissant comme un véritable mime moléculaire, reconnaîtrait le ribosome bloqué sur l’ARNm (dont le site A est vide) et recruterait l’exosome. Il n’y a pas de déadénylation préliminaire dans le NSD. Xrn1 et la dégradation 3’->5’ contribuent au NSD. Chez les procaryotes, les polypeptides synthétisés à partir d’ARNm dépourvus de codon stop sont étiquetés pour la dégradation par l’ajout d’une séquence lue sur un ARNtm [47]. Dans le NGD, les protéines partenaires Dom34 et Hbs1 sont recrutées sur un messager induisant un ralentissement dans la progression du ribosome, ce qui enclenche au moins un clivage endonucléolytique par une protéine qui demeure inconnue [48] ; les fragments générés sont dégradés par Xrn1 et l’exosome. Dom34 et Hbs1 sont homologues respectivement aux facteurs de terminaison de la traduction eRF1 et eRF3 et, dans ce cas, le modèle de reconnaissance du site A vide du ribosome serait également basé sur le principe du mime moléculaire. C. ARNr noyau : les défauts d’assemblage du préribosome entraînent la polyadénylation de leurs ARN par les complexes TRAMP5 (ARN destiné à la petite sous-unité) ou TRAMP4 (grande sous-unité) suivie de leur dégradation par l’exosome. Le SSU-processome qui est un complexe macromoléculaire indispensable à la formation de la petite sous-unité du ribosome est représenté. Il résulte de l’assemblage hiérarchisé de plusieurs modules protéiques autonomes organisés autour de l’ARNsno U3 qui, lui, interagit par appariement Watson-Crick avec l’extrémité 5’ de l’ARN naissant. Cytoplasme : les petites sous-unités défectueuses du ribosome (suite à l’introduction par l’expérimentateur d’altérations en cis- dans des positions ultra-conservées du site de décodage) sont dégradées par le mécanisme de 18S NRD, qui rappelle à beaucoup d’égards le mécanisme de NGD. Les grandes sous-unités non fonctionnelles (résultant de l’introduction du même type de mutation dans le centre peptidyl transférase) sont également reconnues et dégradées. Le processus dénommé 25S NRD est toutefois distinct d’un point de vue du mécanisme mis en jeu. Dans ce cas, des composants protéiques associés avec le ribosome, dont la nature moléculaire reste à préciser, sont ubiquitinylés par le complexe Mms1-Rtt101 et les ribosomes modifiés dirigés pour la dégradation vers le protéasome. |
De façon notable, il existe un certain degré de redondance au sein des activités RNases de l’exosome et dans la spécificité de ses cofacteurs : on parle de « spécificités chevauchantes ». Une telle stratégie confère une robustesse élevée aux nombreuses réactions médiées par l’exosome. Le complexe TRAMP Un des cofacteurs importants de l’exosome dans le cadre de la surveillance est le complexe de polyadénylation nucléaire TRAMP (Trf4/Air2/Mtr4p polyadenylation complex). Il s’agit d’un complexe hétérotrimérique qui résulte de l’association d’une poly(A) polymérase (soit Trf4, soit Trf5) avec une protéine putative de liaison à l’ARN à domaine zinc-knuckle
2, (soit Air1, soit Air2) et une hélicase à ARN à domaine DEVH 3 (Mtr4, également connue sous le nom de Dob1) [
8–
10]. Puisque la poly(A) polymérase et la protéine de liaison à l’ARN sont toutes deux encodées par deux gènes paralogues 4, il existe plusieurs combinaisons possibles dans la formation des complexes TRAMP et c’est la polymérase qui donne le nom aux complexes (TRAMP4 si c’est Trf4 qui est utilisée ; TRAMP5 dans le cas de Trf5). Trf4 et Trf5 ont une localisation sous-cellulaire différente (Trf5 est nucléolaire ; Trf4 est nucléaire), ainsi qu’une abondance relative et des effets sur la croissance distincts qui expliquent sans doute leur préférence pour certains substrats (voir ci-dessous). Les autres facteurs Hormis l’exosome, la cellule dispose de plusieurs activités exoRNases 5’->3’ et endoRNases qui participent à des degrés divers à la surveillance. Pour cette discussion, nous nous limiterons à la description des deux exoRNAses 5’->3’ : Rat1 et Xrn1. Dans le noyau, c’est principalement l’exoRNase Rat1 et son cofacteur Rai1 qui sont actifs dans la surveillance alors que dans le cytoplasme, c’est plutôt Xrn1. La structure tridimensionnelle du complexe Rat1/Rai1, récemment établie à l’échelle atomique [
11], a révélé que non seulement Rai1 stabilise Rat1 mais que par ailleurs, par son activité de pyrophosphatase (Rai1 s’est alors révélée être homologue à la protéine bactérienne RppH), cette protéine augmente l’affinité de Rat1 envers ses substrats en convertissant les extrémités 5’-triphosphates des ARN en 5’-monophosphates. Bien que Rat1 et Xrn1 aient une distribution cellulaire majoritairement nucléaire et cytoplasmique, respectivement, elles peuvent souvent se substituer l’une à l’autre. Ainsi, dans le cas de Xrn1, il a été démontré, à de très nombreuses reprises, qu’elle contribue à des étapes de maturation nucléolaire et nucléaire des précurseurs d’ARN ribosomiques. La fonction de « torpille à polymérases », qui éjecte véritablement les ARN polymérases I et II lors de la terminaison de la transcription semblerait, par contre, spécifique à Rat1 [
12,
13]. Il est intéressant de mentionner la découverte récente chez la levure d’une nouvelle exoRNAse 5’->3’ [
14]. Il s’agit de la protéine Rrp17 qui est conservée chez les mammifères et qui joue un rôle partiellement redondant à celui de Rat1 dans la biogenèse des ribosomes. L’implication éventuelle de Rrp17 dans le métabolisme d’autres classes d’ARN et la surveillance reste à établir. |
Les ARNt constituent une des classes d’ARN les plus abondamment modifiées de la cellule, ce qui a des répercussions importantes sur leur structure et leur fonction. Ainsi, la voie majeure de la surveillance nucléaire, la voie dite TRAMP-exosome, a initialement été mise en évidence sur un précurseur d’ARNt (ARNtMET initiateur) défectueux structuralement en raison de l’absence d’une seule de ses modifications [10,
15,
16] (Figure 2A). Dans cette voie, l’addition de courtes queues de poly(A) et/ou la liaison du complexe TRAMP à l’ARN condamnent véritablement les molécules aberrantes à la dégradation, à la fois en discriminant les ARN normaux des ARN anormaux et en stimulant l’activité de l’exosome à ARN [
2]. Il s’est alors rapidement avéré que le complexe TRAMP joue un rôle assez universel dans ce que nous appelons la discrimination de la norme puisqu’il est susceptible d’envoyer vers la voie de dégradation les précurseurs de la majorité des classes d’ARN testés à ce jour, à savoir : hormis les précurseurs d’ARNt que nous venons d’aborder, ceux des ARNm et des ARNr (voir ci-dessous) (Figures 2B et C), des ARNsn (impliqués dans l’épissage des précurseurs d’ARNm), des ARNsno (impliqués dans la maturation et le clivage des précurseurs d’ARNr), des transcrits cryptiques instables Pol II (les CUT), des introns épissés d’ARNm, etc. Dans le cytoplasme, les ARNt hypomodifiés sont également rapidement éliminés par un processus dénommé RTD pour Rapid tRNA Decay. Ici, la dégradation débute par l’extrémité 5’ des ARNt par les exoRNases Rat1 et Xrn1 [
17] (Figure 2A). |
Dans le noyau, les ARNm naissants sont assemblés cotranscriptionnellement avec de multiples protéines qui vont véritablement sceller leur destin et refléter leur histoire. Ainsi, les pré-ARNm s’associent avec le complexe TREX (transport-export) qui couple leur synthèse, leur maturation et leur export [
18]. Des défauts de maturation ou d’assemblage des pré-ARNm entraînent leur rétention et leur accumulation dans des foci nucléaires, à proximité des sites de transcription, ou leur dégradation rapide par l’exosome et son coactivateur TRAMP [
19,
20]. Par ailleurs, plusieurs protéines associées au pore nucléaire ont été impliquées dans la rétention nucléaire de précurseurs d’ARNm, notamment de ceux qui n’ont pas été correctement épissés [
21,
22]. Enfin, l’existence d’un tel contrôle de qualité, dit périnucléaire, des pré-ARNm est renforcée par la découverte récente de la présence transitoire au pore d’une endoRNase à domaine PIN (la protéine Swt1), qui y cliverait les ARN défectueux, ainsi que par l’observation de l’association persistante aux pores de nombreux gènes dans des mutants du complexe TREX [
23,
24]. Il est important de noter à ce stade que la courte queue de poly(A) ajoutée par TRAMP à l’extrémité 3’ des précurseurs aberrants d’ARN joue le rôle d’une étiquette déstabilisatrice, alors que la longue queue canonique des ARNm est, elle, importante pour l’export, la traduction et la stabilité du messager. Chez les bactéries, la polyadénylation joue également un rôle déstabilisateur pour les ARN. Pour cette raison, il a été proposé que la polyadénylation réalisée par TRAMP soit d’origine ancestrale. Lorsqu’ils sont activement traduits, les ARNm sont circularisés par des interactions entre des protéines qui se lient de part et d’autre de la molécule ; à savoir sur la coiffe monométhylée 5’-terminale et la queue de poly(A). La dégradation d’un ARNm implique majoritairement un raccourcissement de la queue de poly(A) (par des complexes de déadénylation tels que Ccr4-Not et Pan2/Pan3) jusqu’à ce que celle-ci atteigne une taille critique qui ne permette plus la circularisation exposant dès lors la coiffe à la dégradation (par le complexe de décoiffage Dcp1/Dcp2). Le corps du messager est alors rapidement dégradé dans une direction 5’->3’ par Xrn1. Une alternative, qui représente une voie mineure, est que la déadénylation est suivie d’une dégradation 3’->5’ par l’exosome. Dans le cytoplasme, trois grands mécanismes de surveillance des ARNm ont été décrits : le NMD (nonsense-mediated decay), qui détruit les ARNm contenant un codon stop dans un contexte peu favorable à la terminaison de la traduction ; le NSD (nonstop mRNA decay), qui élimine les ARNm dépourvus de codon stop et le NGD (no-go decay) qui dégrade les ARNm qui induisent des pauses traductionnelles [
25–
27]. Ces mécanismes ont fait l’objet d’une revue récente dans Médecine/Sciences [
28] et ne sont donc que brièvement évoqués dans la légende de la Figure 2. Dans certains cas, la dégradation des ARNm est spatialement et fonctionnellement liée à des organites cellulaires spécialisés : les corps de maturation cytoplasmiques (P-bodies). Il s’agit de granules cytoplasmiques à ARN et à protéines, conservés à travers l’évolution et constitués d’ARNm traductionnellement silencieux, en complexe avec un ensemble de répresseurs traductionnels, la machinerie de décoiffage et Xrn1 [
29]. |
Le ribosome est une RNP de grande taille constituée de deux sous-unités porteuses de fonctions spécialisées dans la traduction : la petite sous-unité 40S, qui est responsable du décodage du messager, et la grande sous-unité 60S, qui prend en charge la formation du lien peptidique ; gardons à l’esprit que le ribosome est un ribozyme [
30]. Chaque ribosome est constitué d’une petite centaine de pièces, soit 4 ARNr et environ 80 protéines ribosomiques, et il ne faut pas moins de 200 facteurs de synthèse de nature protéique et autant de ARNsno pour les assembler en sous-unités fonctionnelles. Fabriquer les sous-unités ribosomiques implique de nombreuses étapes dont la plupart sont initiées dans le nucléole [
31]. Ainsi, on dénombre des réactions de synthèse des composants ARN et protéiques, de clivages et de modifications covalentes (méthylation de sucres et de bases, pseudo-uridylation) des ARN précurseurs, de modifications des protéines (phosphorylation, sumoylation, ubiquitinylation, etc.), ainsi que le remodelage constant de la structure tridimensionnelle des particules préribosomiques en cours de maturation et des étapes de transport (intranucléaire et nucléocytoplasmique) [
32,
33]. Le remodelage de la structure des préribosomes et les échanges dynamiques de facteurs de synthèse en leur sein impliquent de nombreuses hélicases à ARN, des ATPases de type AAA et des GTPases [
34,
35]. Chacune de ces nombreuses étapes est soumise à un taux d’erreurs avec pour conséquence la production de ribosomes imparfaits, potentiellement infidèles dans le processus de traduction. Par ailleurs, au vu du coût énergétique gargantuesque qu’implique la production de ribosomes, il est indispensable dans certaines conditions, telles que des carences nutritionnelles, non seulement d’interrompre leur synthèse mais également d’entreprendre activement leur dégradation afin de recycler des « blocs de construction » indispensables pour faire face aux conditions environnementales devenues défavorables. Cinq voies de dégradation des ribosomes À ce jour, cinq voies de dégradation des ribosomes ont été décrites [
36] ; chacune est synthétiquement énoncée ici : (1) la voie TRAMP-exosome [
37–
39], (2) le 18S NRD ( nonfunctional rRNA decay) [
40,
41], (3) le 25S NRD [
42], (4) la ribophagie [
43] et (5) la Piecemeal microautophagy du noyau ou PMN [
44]. Nous avons déjà abordé la voie de la surveillance impliquant TRAMP et l’exosome. C’est la seule voie nucléaire de dégradation de préribosomes décrite à ce jour, les quatre autres étant cytoplasmiques. Les voies TRAMP-exosome et nonfunctional rRNA decay Dans le noyau, tout défaut dans l’assemblage de la machinerie de clivage des précurseurs d’ARN ribosomiques se solde par la polyadénylation de l’extrémité 3’-terminale des ARN par TRAMP, suivie d’une dégradation 3’->5’, médiée par l’exosome. Ici les deux complexes TRAMP4 et TRAMP5 sont à l’œuvre. TRAMP5 préfère les ARNr précurseurs de la petite sous-unité, alors que TRAMP4 agit plutôt sur ceux destinés à former la grande sous-unité. La surveillance nucléaire 3’->5’ entre à de multiples positions sur les précurseurs d’ARNr. Toutefois, au vu de leur grande taille et de leur degré élevé de compaction, il y a fort à parier qu’une dégradation 5’->3’ opère également. Des exoRNases candidates ont été décrites (voir ci-dessus : Xrn1, Rat1 et Rrp17). Dans le 18S NRD, des sous-unités ribosomiques 40S porteuses d’erreurs en cis dans le centre de décodage, et donc non fonctionnelles, sont reconnues par les protéines Dom34 et Hbs1. L’ARNr 18S (constituant de la sous-unité 40S mature) défectueux est vraisemblablement clivé endonucléolytiquement, par une activité qui reste à définir, et les fragments générés sont digérés par Xrn1 et l’exosome (Figure 2C). Le mécanisme du 18S NRD n’est pas sans rappeler celui du NGD puisque dans les deux cas, c’est une petite sous-unité en cours de lecture du messager qui est reconnue et que les facteurs trans impliqués à ce jour sont les protéines Dom34 et Hbs1 (voir la légende de la Figure 2). Les protéines Dom34 et Hbs1 sont respectivement homologues aux facteurs de terminaison de la traduction eRF1 et eRF3. eRF1 adopte une structure tridimensionnelle qui mime la forme et la distribution de charges d’un ARNt ; cette configuration est largement conservée dans Dom34. Les grandes sous-unités ribosomiques porteuses de mutations dans le centre peptidyl transférase sont elles aussi reconnues par la surveillance, mais dans ce cas, le mécanisme est totalement différent puisqu’il implique l’ubiquitinylation de certains de ses composants protéiques, par le complexe Mms1-Rtt101 suivi d’un adressage au protéasome (Figure 2C). Macro-autophagie et micro-autophagie Finalement, en condition de carences nutritionnelles, les ribosomes surnuméraires sont adressés vers le lysosome (vacuole chez la levure et les plantes) pour y être digérés. Deux voies ont ici été découvertes qui dépendent soit du processus de macro-autophagie soit de celui de micro-autophagie. La différence entre la macro-autophagie et la micro-autophagie réside dans le mécanisme de capture du matériel à dégrader. Dans la macro-autophagie, le matériel cytoplasmique à dégrader est séquestré par la formation d’une vésicule (autophagosome) qui vient se fusionner à la vacuole. Dans la micro-autophagie, le matériel à digérer est directement capturé par une invagination de la membrane vacuolaire. Globalement, l’autophagie est un processus aspécifique, n’importe quelles molécules ou organelles présentes à l’endroit de la séquestration étant amenées à la vacuole ; toutefois des situations de macro-autophagie et de micro-autophagie spécifiques ont été décrites. Ainsi, la macro-autophagie spécifique du ribosome mature est dénommée la ribophagie, et implique, dans le cas de la grande sous-unité, la déubiquitinylation de composants ribosomiques, par le complexe Bre5-Ubp3 [ 43]. Dans le cas des petites sous-unités, qui sont également soumises à la ribophagie, l’effecteur reste à identifier. Il est à noter qu’à ce jour, aussi bien dans le cas du 25S NRD que dans celui de la ribophagie des sous-unités 60S, les composants ribosomiques qui sont respectivement ubiquitinylés et déubiquitinylés restent à découvrir. Enfin, le dernier mécanisme de dégradation des ribosomes, la piecemeal microautophagy du noyau, implique la formation d’une jonction spécialisée (dénommée la jonction vacuole-noyau ou NVJ) directement entre la membrane de la vacuole et celles du noyau qui mène à la dégradation aspécifique de préribosomes par le transfert direct de matériel nucléolaire vers la vacuole. |
Quels sont les mécanismes de reconnaissance de la norme ? Nos connaissances actuelles pointent essentiellement sur deux mécanismes possibles. Dans le noyau, c’est la dynamique d’assemblage, c’est-à-dire la cinétique, qui serait contrôlée. Ainsi, chaque réaction disposerait d’une fenêtre d’opportunité très précise pour se réaliser ; fenêtre au-delà de laquelle la particule en cours de maturation serait détournée vers une voie de dégradation (notamment suite à la polyadénylation des ARN par TRAMP). Le mode précis de reconnaissance des substrats à dégrader par TRAMP est actuellement inconnu. Dans le cytoplasme, c’est plutôt la fonction des particules RNP qui serait vérifiée ; ceci est bien illustré par les mécanismes du NGD et du NRD dans lesquels c’est le ralentissement de la progression du ribosome sur le messager qui enclenche la surveillance (Figure 2). |
Pourquoi a-t-on besoin de la surveillance ? La dégradation des ARN cellulaires a deux fonctions : elle permet d’éliminer ce qui est non fonctionnel, à la suite d’un défaut de synthèse ou d’une altération, et ce qui est devenu inutile ou surnuméraire, à la suite de conditions de croissance altérées ou devenues restrictives. Plusieurs observations illustrent que la surveillance est indispensable au maintien de l’homéostasie cellulaire. Ainsi, il est possible, en inactivant la surveillance nucléaire, de forcer l’accumulation de précurseurs de ribosomes partiellement mal assemblés. Toutefois des cellules porteuses de tels ribosomes ne sont pas viables, ce qui suggère qu’ils auraient bel et bien dû être éliminés car ils ne sont pas fonctionnels [37]. Chez la levure toujours, des cellules défectueuses pour la ribophagie ont une durée de vie réduite et sont incapables de faire face à des stress nutritionnels [43]. Enfin, chez l’humain, l’accumulation de ribosomes altérés par oxydation a été corrélée avec des maladies dégénératives sévères telles que les maladies d’Alzheimer et de Parkinson, ainsi que dans la formation de la plaque athérosclérotique, indiquant qu’une surveillance défectueuse pourrait jouer un rôle prépondérant dans l’étiologie, et peut-être même dans la progression, de certaines maladies [
45,
46]. |
Dans ces quelques pages, nous espérons avoir donné un aperçu global des mécanismes de la surveillance des ARN. Les principes fondateurs de cette surveillance sont, d’une part, qu’elle est active à toutes les étapes de vie d’un ARN, et ce depuis son site de synthèse jusqu’à son site de fonction, et d’autre part qu’elle se fonde sur le recrutement par des cofacteurs spécifiques de facteurs généraux (dont l’exosome à ARN est l’exemple canonique). Les enjeux de la recherche sur la surveillance des ARN résident dans la compréhension détaillée de l’expression régulée de nos gènes. Un des défis pour le futur est d’établir précisément les mécanismes moléculaires de discrimination de la norme et d’établir à quel point ils sont spécifiques d’un substrat. |
Les auteurs déclarent n’avoir aucun conflit d’intérêts concernant les données publiées dans cet article.
|
Footnotes |
1. Lykke-Andersen S, Brodersen DE, Jensen TH. Origins and activities of the eukaryotic exosome. J Cell Sci 2009; 122 : 1487–94. 2. Andersen KR, Jensen TH, Brodersen DE. Take the A tail--quality control of ribosomal and transfer RNA. Biochim Biophys Acta 2008; 1779 : 532–7. 3. Schmid M, Jensen TH. The exosome: a multipurpose RNA-decay machine. Trends Biochem Sci 2008; 33 : 501–10. 4. Schaeffer D, Tsanova B, Barbas A, et al. The exosome contains domains with specific endoribonuclease, exoribonuclease and cytoplasmic mRNA decay activities. Nat Struct Mol Biol 2009; 16 : 56–62. 5. Lebreton A, Tomecki R, Dziembowski A, Seraphin B. Endonucleolytic RNA cleavage by a eukaryotic exosome. Nature 2008; 456 : 993–6. 6. Houseley J, LaCava J, Tollervey D. RNA-quality control by the exosome. Nat Rev Mol Cell Biol 2006; 7 : 529–39. 7. Liu Q, Greimann JC, Lima CD. Reconstitution, activities, and structure of the eukaryotic RNA exosome. Cell 2006; 127 : 1223–37. 8. LaCava J, Houseley J, Saveanu C, et al. RNA degradation by the exosome is promoted by a nuclear polyadenylation complex. Cell 2005; 121 : 713–24. 9. Wyers F, Rougemaille M, Badis G, et al. Cryptic pol II transcripts are degraded by a nuclear quality control pathway involving a new poly(A) polymerase. Cell 2005; 121 : 725–37. 10. Vanacova S, Wolf J, Martin G, et al. A new yeast poly(A) polymerase complex involved in RNA quality control. PLoS Biol 2005; 3 : e189. 11. Xiang S, Cooper-Morgan A, Jiao X, et al. Structure and function of the 5’-->3’ exoribonuclease Rat1 and its activating partner Rai1. Nature 2009; 458 : 784–8. 12. El Hage A, Koper M, Kufel J, Tollervey D. Efficient termination of transcription by RNA polymerase I requires the 5’ exonuclease Rat1 in yeast. Genes Dev 2008; 22 : 1069–81. 13. Kawauchi J, Mischo H, Braglia P, et al. Budding yeast RNA polymerases I and II employ parallel mechanisms of transcriptional termination. Genes Dev 2008; 22 : 1082–92. 14. Oeffinger M, Zenklusen D, Ferguson A, et al. Rrp17p is a eukaryotic exonuclease required for 5’ end processing of Pre-60S ribosomal RNA. Mol Cell 2009; 36 : 768–81. 15. Kadaba S, Krueger A, Trice T, et al. Nuclear surveillance and degradation of hypomodified initiator tRNAMet in S. cerevisiae. Genes Dev 2004; 18 : 1227–40. 16. Schneider C, Anderson JT, Tollervey D. The exosome subunit Rrp44 plays a direct role in RNA substrate recognition. Mol Cell 2007; 27 : 324–31. 17. Chernyakov I, Whipple JM, Kotelawala L, et al. Degradation of several hypomodified mature tRNA species in Saccharomyces cerevisiae is mediated by Met22 and the 5’-3’ exonucleases Rat1 and Xrn1. Genes Dev 2008; 22 : 1369–80. 18. Strasser K, Masuda S, Mason P, et al. TREX is a conserved complex coupling transcription with messenger RNA export. Nature 2002; 417 : 304–8. 19. Rougemaille M, Gudipati RK, Olesen JR, et al. Dissecting mechanisms of nuclear mRNA surveillance in THO/sub2 complex mutants. EMBO J 2007; 26 : 2317–26. 20. Thomsen R, Libri D, Boulay J, et al. Localization of nuclear retained mRNAs in Saccharomyces cerevisiae. RNA 2003; 9 : 1049–57. 21. Galy V, Gadal O, Fromont-Racine M, et al. Nuclear retention of unspliced mRNAs in yeast is mediated by perinuclear Mlp1. Cell 2004; 116 : 63–73. 22. Vinciguerra P, Iglesias N, Camblong J, et al. Perinuclear Mlp proteins downregulate gene expression in response to a defect in mRNA export. EMBO J 2005; 24 : 813–23. 23. Skruzny M, Schneider C, Racz A, et al. An endoribonuclease functionally linked to perinuclear mRNP quality control associates with the nuclear pore complexes. PLoS Biol 2009; 7 : e8. 24. Rougemaille M, Dieppois G, Kisseleva-Romanova E, et al. THO/Sub2p functions to coordinate 3’-end processing with gene-nuclear pore association. Cell 2008; 135 : 308–21. 25. Isken O, Maquat LE. Quality control of eukaryotic mRNA: safeguarding cells from abnormal mRNA function. Genes Dev 2007; 21 : 1833–56. 26. Doma MK, Parker R. RNA quality control in eukaryotes. Cell 2007; 131 : 660–8. 27. Houseley J, Tollervey D. The many pathways of RNA degradation. Cell 2009; 136 : 763–76. 28. Camier S, Séraphin B. Détruisez ce message (ARN) après l’avoir lu ! Med Sci (Paris) 2007; 23 : 850–6. 29. Balagopal V, Parker R. Polysomes, P bodies and stress granules: states and fates of eukaryotic mRNAs. Curr Opin Cell Biol 2009; 21 : 403–8. 30. Lafontaine DLJ, Tollervey D. The function and synthesis of ribosomes. Nat Rev Mol Cell Biol 2001; 2 : 514–20. 31. Hernandez-Verdun D, Louvet E. Le nucléole : structure, fonctions et maladies associées. Med Sci (Paris) 2004; 20 : 37–44. 32. Fromont-Racine M, Senger B, Saveanu C, Fasiolo F. Ribosome assembly in eukaryotes. Gene 2003; 313 : 17–42. 33. Henras AK, Soudet J, Gerus M, et al. The post-transcriptional steps of eukaryotic ribosome biogenesis. Cell Mol Life Sci 2008; 65 : 2334–59. 34. Strunk BS, Karbstein K. Powering through ribosome assembly. RNA 2009; 15 : 2083–104. 35. Guenther UP, Jankowsky E. Helicase multitasking in ribosome assembly. Mol Cell 2009; 36 : 537–8. 36. Lafontaine DLJ. A garbage can for ribosomes: how eukaryotes degrade their ribosomes ? Trends Biochem Sci 2010, Epub ahead of print PMID: 20097077. 37. Wery M, Ruidant S, Schillewaert S, et al. The nuclear poly(A) polymerase and Exosome cofactor Trf5 is recruited cotranscriptionally to nucleolar surveillance. RNA 2009; 15 : 406–19. 38. Dez C, Dlakic M, Tollervey D. Roles of the HEAT repeat proteins Utp10 and Utp20 in 40S ribosome maturation. RNA 2007; 13 : 1516–27. 39. Dez C, Houseley J, Tollervey D. Surveillance of nuclear-restricted pre-ribosomes within a subnucleolar region of Saccharomyces cerevisiae. EMBO J 2006; 25 : 1534–46. 40. LaRiviere FJ, Cole SE, Ferullo DJ, Moore MJ. A late-acting quality control process for mature eukaryotic rRNAs. Mol Cell 2006; 24 : 619–26. 41. Cole SE, LaRiviere FJ, Merrikh CN, Moore MJ. A convergence of rRNA and mRNA quality control pathways revealed by mechanistic analysis of nonfunctional rRNA decay. Mol Cell 2009; 34 : 440–50. 42. Fujii K, Kitabatake M, Sakata T, et al. A role for ubiquitin in the clearance of nonfunctional rRNAs. Genes Dev 2009; 23 : 963–74. 43. Kraft C, Deplazes A, Sohrmann M, Peter M. Mature ribosomes are selectively degraded upon starvation by an autophagy pathway requiring the Ubp3p/Bre5p ubiquitin protease. Nat Cell Biol 2008; 10 : 602–10. 44. Roberts P, Moshitch-Moshkovitz S, Kvam E, et al. Piecemeal microautophagy of nucleus in Saccharomyces cerevisiae. Mol Biol Cell 2003; 14 : 129–41. 45. Nunomura A, Hofer T, Moreira PI, et al. RNA oxidation in Alzheimer disease and related neurodegenerative disorders. Acta Neuropathol 2009; 118 : 151–66. 46. Martinet W, De Meyer GR, Herman AG, Kockx MM. RNA damage in human atherosclerosis: pathophysiological significance and implications for gene expression studies. RNA Biol 2005; 2 : 4–7. 47. Keiler KC. Biology of trans-translation. Annu Rev Microbiol 2008; 62 : 133–51. 48. Passos DO, Doma MK, Shoemaker CJ, et al. Analysis of Dom34 and its function in no-go decay. Mol Biol Cell 2009; 20 : 3025–32. |