| |
| Med Sci (Paris). 2009 May; 25(5): 439–441. Published online 2009 May 15. doi: 10.1051/medsci/2009255439.Le microenvironnement tumoral Un nouveau paramètre pronostique dans le cancer du sein Greg Finak,1 Julie Laferrièe,2* Michael Hallett,1,2 and Morag Park2,3 1McGill Center for Bioinformatics, Universitè McGill, Montrèal, Quèbec, Canada 2Rosalind and Morris Goodman Cancer Centre, Universitè McGill, 1160, avenue des Pins ouest, Montrèal, Quèbec H3A 1A3, Canada 3Dèpartement d’oncologie, Universitè McGill, Montrèal, Quèbec, Canada MeSH keywords: Tumeurs du sein, Carcinome canalaire du sein, Femelle, Analyse de profil d'expression de gènes, Humains, Protéines tumorales, Réaction de polymérisation en chaîne, Valeur prédictive des tests, Pronostic, Cellules stromales, Marqueurs biologiques tumoraux |
Le cancer du sein, particulièrement le carcinome canalaire infiltrant, est une maladie très hétérogène médicalement et histologiquement. Les études génomiques de tumeurs du sein ont permis de déceler de nouveaux sous-types moléculaires, notamment de type basal, exprimant HER2 (epidermal growth factor type II), ou luminal (sous-types A et B), qui se caractérisent par des profils d’expression génique uniques, ainsi que par des aberrations chromosomiques particulières [
1,
2,
14,
15]. De multiples données tendent à indiquer que ces différences moléculaires influencent la réponse aux traitements, la progression tumorale, ainsi que le pronostic [
3,
4]. Un degré de complexité supplémentaire est apporté par l’intervention du microenvironnement tumoral qui interagit avec les cellules cancéreuses et modifie plusieurs aspects du développement tumoral, notamment la croissance, l’angiogenèse, l’invasion, la formation de métastases, ainsi que l’échappement immunitaire [
5–
7]. Toutefois, peu d’études génomiques à grande échelle à partir d’échantillons cliniques de cancer du sein ont évalué l’impact du microenvironnement tumoral sur le pronostic [
8–
10]. |
Identification d’une signature moléculaire prédictive spécifique des modifications du stroma tumoral Nous avons entrepris l’une des premières études de ce genre [
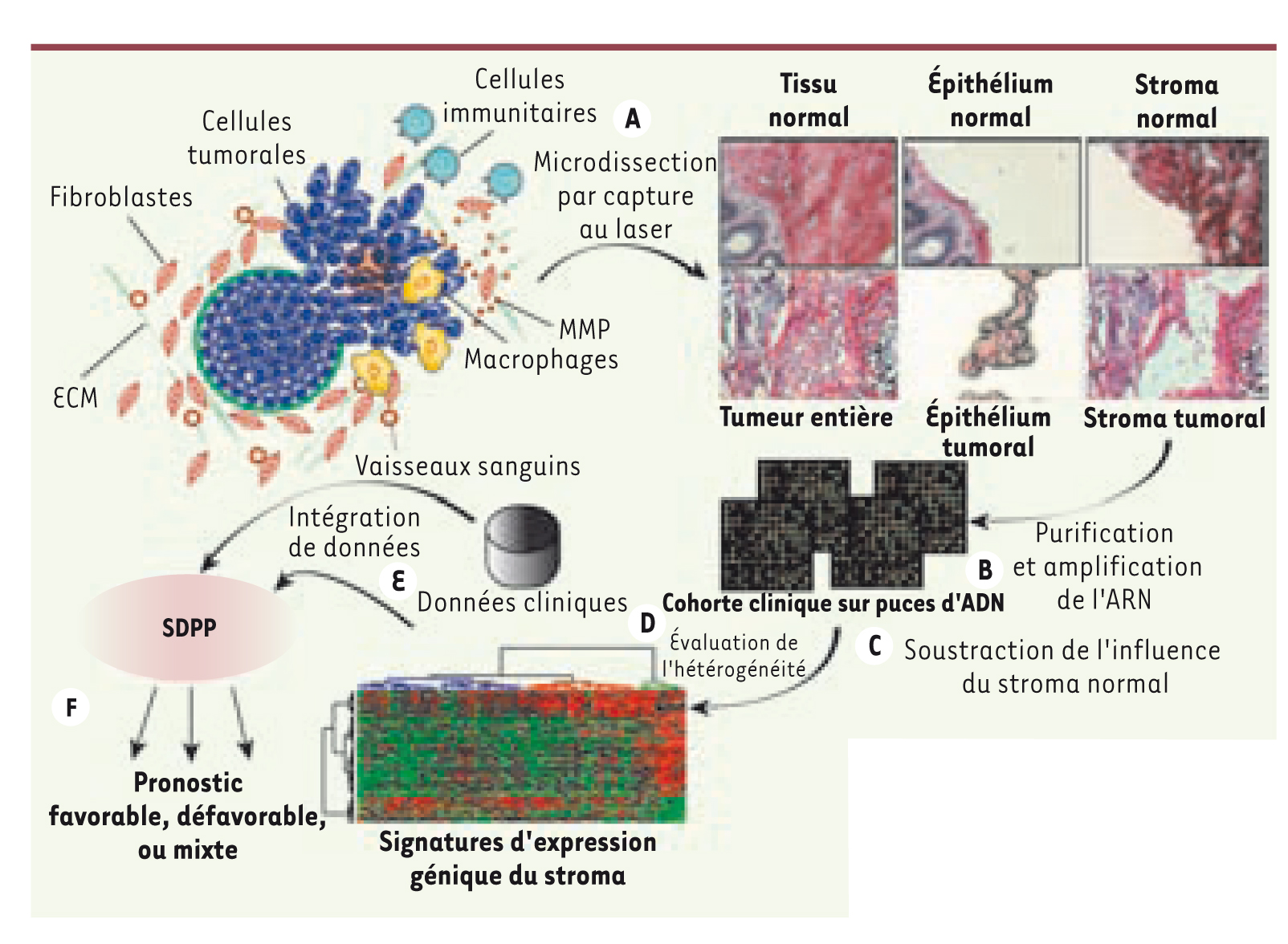
11]. À cette fin, nous avons appliqué à 53 tumeurs du sein primaires (carcinome canalaire infiltrant) une approche de microdissection par capture au laser afin d’isoler successivement, chez une même patiente, les cellules stromales qui entourent la tumeur, ainsi que des cellules du tissu normal (Figure 1A) [
12]. Nous avons utilisé les données d’expression génique obtenues à partir de ces échantillons analysés par puces à ADN (Figure 1B) afin de déceler des changements spécifiques au stroma des tumeurs par opposition au stroma normal (Figure 1C). En étudiant la correspondance entre le pronostic clinique et les signatures d’expression génique constituées de centaines de gènes reflétant les modifications du stroma tumoral (Figure 1D) [11], nous avons produit une courte liste de 26 gènes qui peut être adaptée en un test clinique (Figure 1E). Ce groupe de 26 gènes, que nous avons nommé stroma-derived prognostic predictor (SDPP) ou test prédictif pronostique établi à partir du stroma, s’est avéré indépendant non seulement des facteurs pronostiques cliniques courants mais aussi des autres tests prédictifs existants fondés sur des analyses d’expression génique de tumeurs entières. Le SDPP identifie des individus dont la tumeur est classée comme de mauvais pronostic d’après les données de multiples bases de données d’expression génique provenant de tumeurs entières, et dont la tumeur appartient à divers sous-types histopathologiques de cancer du sein, et en particulier ceux sans envahissement ganglionnaire. De plus, cette signature SDPP semble beaucoup plus puissante et fiable que les autres facteurs prédictifs précédemment établis et rendus publics. C’est particulièrement vrai dans le cas des tumeurs surexprimant HER2 (human epidermal growth factor receptor 2) : chez ces patientes, la signature SDPP offre jusqu’à six fois plus de chances que la signature de 70 gènes décrite précédemment [3] de prédire une évolution défavorable. Les 26 gènes constituant le SDPP sont le reflet des multiples processus survenant dans le microenvironnement, qu’il s’agisse de réactions immunitaires, de réponses à l’hypoxie ou du processus d’angiogenèse. Parmi les gènes associés à un pronostic favorable, on compte ceux qui codent pour des marqueurs des cellules T CD8 ou des cellules NK (natural killer) ; des gènes de réponse à l’hypoxie et des gènes pro-angiogéniques sont au contraire associés à un pronostic défavorable. Ces résultats suggèrent que l’immunosurveillance est un paramètre important du pronostic. D’ailleurs, un modèle suggère que les macrophages associés à la tumeur, qui sont recrutés dans les régions hypoxiques des tumeurs dont la croissance est rapide, peuvent avoir des fonctions immunosuppressives, proangiogéniques ainsi que des fonctions de remodelage de la matrice extracellulaire, favorisant ainsi l’invasion des cellules tumorales et la progression cancéreuse [
13]. En revanche, les individus ayant déclenché une réponse immunitaire anti-tumorale semblent bénéficier d’un bon pronostic.  | Figure 1.
Étapes menant à l’instauration d’un test de prédiction pronostique basé sur les caractéristiques du microenvironnement tumoral. A. Le stroma tumoral et le stroma normal sont isolés à l’aide de la technologie de microdissection par capture au laser. B. L’ARN est isolé et amplifié pour être analysé par la technique des puces à ADN. C. À partir de la base de données de la cohorte clinique, l’influence du stroma normal est supprimée. D. L’hétérogénéité des profils d’expression génique est évaluée et des signatures sont identifiées. E. Les données sont intégrées avec les informations cliniques. F. Le prédicteur tripartite dérivé du stroma est identifié et validé. MMP : métalloprotéinases de la matrice ; ECM : extracellular matrix ; SDPP : stroma-derived prognostic predictor.
|
|
Paramètres techniques critiques Cette capacité qu’offre un test fondé uniquement sur la biologie du microenvironnement tumoral de prévoir des résultats cliniques dans différents types de cancer du sein ne fait que mettre en exergue l’importance, sous-estimée jusqu’alors, du rôle de ce microenvironnement dans la progression tumorale. Les 26 gènes représentatifs des propriétés du stroma surpassent à eux seuls les facteurs prédictifs moléculaires existants sélectionnés à partir de plus grands ensembles de gènes identifiés par l’analyse de tumeurs entières. Puisque l’utilisation du SDPP, en combinaison avec les facteurs prédictifs existants, améliore la prédiction de l’évolution tumorale, il paraît donc important d’intégrer ces aspects microenvironnementaux dans l’évaluation pronostique d’une tumeur. Plusieurs points techniques importants expliquent la puissance de la signature SDPP que nous avons décrite : pour sélectionner les caractéristiques voulues, l’ensemble des gènes a été ordonné selon le pouvoir prédictif de chaque gène indépendamment des facteurs pronostiques cliniques et pathologiques connus (Figure 1D). Une telle classification a favorisé l’inclusion de gènes liés à des processus biologiques potentiellement originaux influençant le pronostic par rapport aux gènes associés aux signatures génétiques connues, par exemple celles des récepteur hormonaux. Un deuxième aspect important concerne la classification tripartite du SDPP. Le SDPP subdivise les tumeurs en fonction du pronostic, favorable, défavorable ou mixte. Cette troisième catégorie regroupe les tumeurs que le SDPP ne peut pas ranger avec certitude dans l’une des deux autres catégories, faute d’information suffisante pour établir une prédiction avec un degré de confiance suffisamment élevé. En général, une telle approche permet des prédictions plus précises ; à défaut, elle donne une représentation honnête des cas où les méthodes automatisées de prévision échouent. |
Vers un test prédictif utilisable en routine clinique ? Actuellement, la grande variabilité interlaboratoire et les coûts inhérents à la mise en place des plateformes technologiques comptent parmi les principaux obstacles qui freinent l’utilisation clinique des facteurs prédictifs fondés sur des résultats d’expression génique. Cette situation compromet l’application clinique des données des puces à ADN ; il s’y ajoute la taille importante de la plupart des « signatures » moléculaires proposées pour évaluer le pronostic, qui ne facilite pas leur détection par les platesformes diagnostiques communément utilisées. En revanche, la taille relativement petite de la signature SDPP rend ce test plus propice à un usage clinique et les 26 gènes peuvent facilement être adaptés à une analyse diagnostique standard par l’intermédiaire d’une approche de PCR multiplexe.
La classification des cancers du sein en sous-types moléculaires établie par Sørlie et al., est maintenant assez bien reconnue [1]. Les futures études devraient examiner les rapports entre le microenvironnement tumoral et les sous-types moléculaires de cancer du sein. Une meilleure compréhension des interactions tumeur-stroma permettrait de proposer de nouvelles stratégies thérapeutiques tenant compte à la fois des modifications du stroma et de l’épithélium tumoral. Par exemple, une telle approche pourrait permettre de mieux identifier les patients susceptibles de bénéficier des thérapies anti-angiogéniques en combinaison avec des traitements anti-tumoraux ; on se rapprocherait ainsi de l’idéal de la personnalisation des traitements. |
1. Sorlie T, Tibshirani R, Parker J, et al. Repeated observation of breast tumor subtypes in independent gene expression data sets. Proc Natl Acad Sci USA 2003; 100 : 8418–23. 2. Chin K, DeVries S, Fridlyand J, et al. Genomic and transcriptional aberrations linked to breast cancer pathophysiologies. Cancer Cell 2006; 10 : 529–41. 3. Van’t Veer LJ, Dai H, van de Vijver MJ, et al. Gene expression profiling predicts clinical outcome of breast cancer. Nature 2002; 415 : 530–6. 4. Van de Vijver MJ, He YD, van’t Veer LJ, et al. A gene-expression signature as a predictor of survival in breast cancer. N Engl J Med 2002; 347 : 1999–2009. 5. Condeelis J, Pollard JW. Macrophages: obligate partners for tumor cell migration, invasion, and metastasis. Cell 2006; 124 : 263–6. 6. Smyth MJ, Dunn GP, Schreiber RD. Cancer immunosurveillance and immunoediting: the roles of immunity in suppressing tumor development and shaping tumor immunogenicity. Adv Immunol 2006; 90 : 1–50. 7. Radisky DC, Bissell MJ. Cancer. Respect thy neighbor! Science 2004; 303 : 775–7. 8. Chang HY, Sneddon JB, Alizadeh AA, et al. Gene expression signature of fibroblast serum response predicts human cancer progression: Similarities between tumors and wounds. PLoS Biol 2004; 2 : E7. 9. West RB, Nuyten DSA, Subramanian S, et al. Determination of stromal signatures in breast carcinoma. PLoS Biol 2005; 3 : e187. 10. Allinen M, Beroukhim R, Cai L, et al. Molecular characterization of the tumor microenvironment in breast cancer. Cancer Cell 2004; 6 : 17–32. 11. Finak G, Bertos N, Pepin F, et al. Stromal gene expression predicts clinical outcome in breast cancer. Nat Med 2008; 14 : 518–27. 12. Finak G, Sadekova S, Pepin F, et al. Gene expression signatures of morphologically normal breast tissue identify basal-like tumors. Breast Cancer Res 2006; 8 : R58. 13. Lewis CE, Pollard JW. Distinct role of macrophages in different tumor microenvironments. Cancer Res 2006; 66 : 605–12. 14. Bertucci F, Finetti P, Cervera N, Birnbaum D. Classification pronostique du cancer du sein et profils d’expression génique sur puces à ADN. Med Sci (Paris) 2008; 24 : 599–606. 15. Charafe-Jauffret E, Chaffanet M, Bertucci F, et al. Les cancers du sein : vers un modèle cellulaire et moléculaire intégré. Med Sci (Paris) 2007; 23 : 626–32. |