| |
| Med Sci (Paris). 2009 April; 25(4): 383–390. Published online 2009 April 15. doi: 10.1051/medsci/2009254383.Les deux visages de l’autophagie dans le système nerveux Julien Puyal,1* Vanessa Ginet,1,2 Anne Vaslin,1 Anita C. Truttmann,2 and Peter G.H. Clarke1* 1Département de Biologie Cellulaire et de Morphologie (DBCM), Université de Lausanne, Rue du Bugnon 9, CH-1005 Lausanne, Suisse 2Département de Pédiatrie et de Chirurgie Pédiatrique, Centre Hospitalier Universitaire et Université de Lausanne, 1011 Lausanne, Suisse |
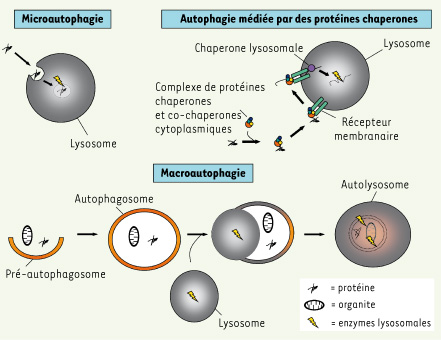
L’autophagie est un processus d’auto-digestion qui consiste en une dégradation de composants intracellulaires par le lysosome. Il existe trois différents types d’autophagie [
1] : la microautophagie, l’autophagie médiée par des protéines chaperones et la forme principale, la macroautophagie (Figure 1). Dans cette revue nous nous focaliserons sur la macroautophagie (que nous désignerons par la suite par autophagie), en nous concentrant sur ses rôles dans les neurones. Bien que le terme « autophagie » soit apparu il y a un peu plus d’une quarantaine d’années, ce n’est que très récemment, grâce notamment à l’apport des outils génétiques, que ce processus a fait l’objet d’un nombre croissant d’études. L’autophagie joue un rôle physiologique important dans la croissance cellulaire, la différenciation et surtout dans le maintien de l’homéostasie qui nécessite l’élimination et le remplacement continuel des protéines et des organites non fonctionnels. Ce turn-over est accompli par différentes voies dont les principales sont la voie du protéasome et celle de l’autophagie. Alors que le système ubiquitine-protéasome représente la voie la plus importante de dégradation des protéines à courte durée de vie [
2], l’autophagie est le principal mécanisme impliqué dans le catabolisme des protéines à longue durée de vie et des organites (mitochondries, réticulum endoplasmique, peroxisomes…). D’autre part, l’autophagie peut représenter une réponse de survie dans certaines conditions de stress en assurant l’élimination des métabolites toxiques, des pathogènes intracellulaires et des organites ou des protéines endommagés. Son activation permet également une production énergétique suffisante au maintien des fonctions vitales lors de conditions pauvres en nutriments. Cependant, dans certaines conditions, une digestion anormale de constituants essentiels de la cellule par autophagie pourrait accompagner la mort cellulaire. Ce rôle paradoxal de l’autophagie, présent dans de nombreux types cellulaires, est évident dans les neurones. 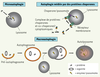 | Figure 1.
Types d’autophagie dans la cellule. Il existe 3 formes d’autophagie. La microautophagie est un processus impliquant la séquestration d’une partie du cytoplasme par la membrane du lysosome directement. L’autophagie médiée par des protéines chaperones concerne spécifiquement les protéines qui portent une séquence précise de 5 acides aminés (KFERQ). Ces protéines sont prises en charge par un complexe formé par les protéines chaperones (hsc70) et co-chaperones (hip, hop, hsp40, hsp90 et bag1) qui les dirigent jusqu’au lysosome où un récepteur associé à la membrane lysosomale (LAMP-2A) les reconnaît et permet leur translocation dans la lumière du lysosome. Enfin, la macroautophagie, forme la moins sélective d’autophagie, dans laquelle une portion entière de cytoplasme contenant les organites ou les protéines à dégrader est séquestrée dans une vésicule formée de plusieurs membranes nommée autophagosome. L’origine de ces membranes n’est pas élucidée mais on parle d’un processus d’élongation d’une structure membranaire nommée « pré-autophagosome » (ou phagophore). L’autophagosome va ensuite fusionner avec un lysosome pour former un « autolysosome » dans lequel les enzymes lysosomales pourront digérer les différents constituants. Ainsi la macroautophagie génère des nucléotides, des acides aminés et des acides gras libres qui pourront être réutilisés pour la synthèse de nouvelles macromolécules et d’énergie (ATP). |
|
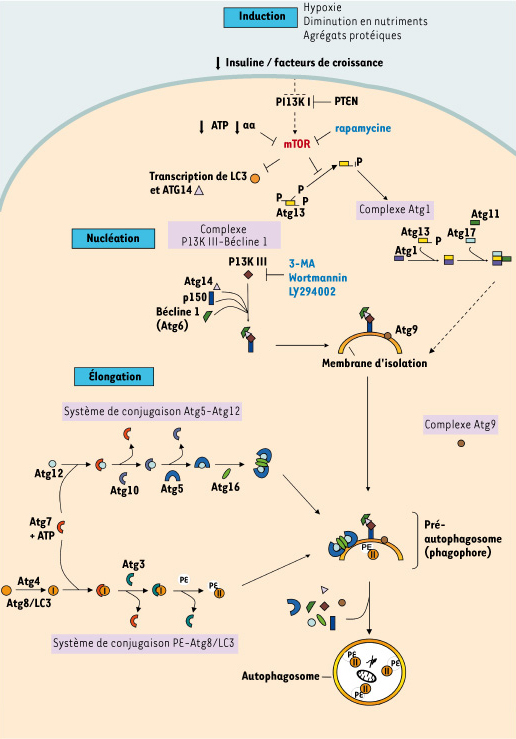
Mécanismes moléculaires de la (macro)autophagie Bien que l’autophagie ait tout d’abord été décrite à l’aide de critères morphologiques dans des cellules de mammifères, ses mécanismes moléculaires ont été découverts et disséqués chez la levure. Aujourd’hui, plus de 30 gènes Atg (pour autophagy-related genes) ont été mis en évidence chez la levure, dont plus de la moitié sont impliqués dans l’autophagie, et sont fortement conservés de la levure aux cellules de mammifères. Étant un processus physiologique essentiel, l’autophagie est très précisément régulée, maintenue à un niveau basal en conditions normales et induite lorsqu’elle est nécessaire. La protéine kinase mTor (mammalian target of rapamycin) est l’acteur central de la régulation et de l’induction de l’autophagie [
3,
4] (Figure 2), bien que, récemment, une induction indépendante de mTor ait été également décrite [
5]. En effet, mTor, lorsqu’elle est active, inhibe la voie de l’autophagie. La diminution en nutriments est un inducteur important de l’autophagie. Dans une telle condition, mTor est inactivée, ce qui permet la levée de son inhibition sur différentes cascades de signalisation menant soit à la modification de l’activité des protéines Atg, soit au changement de leur niveau d’expression.  | Figure 2.
Mécanismes moléculaires impliqués dans la formation de l’autophagosome et dans la régulation de l’autophagie. La protéine kinase mTor, inhibiteur de l’autophagie, est l’acteur central du contrôle de l’induction de l’autophagie. La diminution en nutriments (manque en acides aminés (aa) et en énergie [ATP]) et par conséquent la diminution en facteurs de croissance ou en insuline, sont des inducteurs importants de l’autophagie. En effet, dans de telles conditions, mTor est inhibée suite notamment à l’activité de la phosphatase PTEN qui inhibe la phosphoinositol 3 kinase de classe I. Ainsi Atg13 est partiellement déphosphorylée (mTor contrôlant son état de phosphorylation) ce qui permet sa liaison avec Atg1. Ce complexe se lie ensuite à Atg11 et Atg17 induisant ainsi une augmentation de l’autophagie. En parallèle, l’inactivation de mTor permet également l’activation de la PP2A (protein phosphatase 2A) et la translocation de Gln3 (glutaminase) dans le noyau où il active la transcription de gènes autophagiques tels que LC3 et Atg14. Suite à cette phase d’induction les étapes de nucléation vésiculaire (dans laquelle le complexe Bécline 1/PI3KIII joue un rôle indispensable) et d’élongation (assurée par l’action concomitante des deux systèmes de conjugaisons Atg12-Atg5 et PE-Atg8/LC3) sont activées. Tout au long du processus d’élongation, le complexe Atg9 (Atg9, Atg2 et Atg18) joue un rôle essentiel dans le recrutement de ces différentes protéines. Ainsi le pré-autophagosome (ou phagophore) s’allonge, entoure les constituants à dégrader, puis se referme pour former un autophagosome doté le plus souvent d’une double membrane. LC3-II est la seule protéine Atg connue à ce jour présente au stade d’autophagosome, toutes les autres étant recyclées (grâce à l’action du complexe Atg9). La rapamycine est un activateur de l’autophagie (inhibe mTor) alors que la 3-MA (3-méthyladénine), la wortmannine et le LY294002 sont des inhibiteurs de la voie de la PI3K-III et par conséquent de l’autophagie. |
La biogenèse de l’autophagosome commence par une phase de nucléation qui consiste en la formation d’une portion membranaire comprenant toutes les protéines nécessaires à la création d’un autophagosome et dont l’origine est encore débattue. Cette membrane isolant ces constituants forme ensuite un pré-autophagosome (ou phagophore) en réponse à une phase d’élongation, puis d’incurvation. Enfin la fusion bout à bout de la membrane séquestre les constituants du cytoplasme à dégrader dans une vésicule à double membrane, l’autophagosome. Ce processus de biogenèse de l’autophagosome dépend de l’interaction de différents complexes dans lesquels chaque protéine Atg a un ou plusieurs rôles à jouer [
6–
8]. Les complexes les plus importants • Le complexe protéique Atg1 Lorsque mTor est inactivée, Atg13 est partiellement phosphorylée et peut ainsi se lier à Atg1 (une sérine-thréonine protéine kinase). Le complexe interagit ensuite avec Atg11 et Atg17, ce qui induit l’activité d’Atg1 et ainsi l’augmentation de l’autophagie. Le complexe Atg1 est donc impliqué dans l’étape d’initiation et de nucléation (par son action dans le recrutement de différentes protéines Atg au niveau de la membrane d’isolation) mais également dans la dynamique et l’élongation de la membrane du pré-autophagosome (car l’activité kinase d’Atg1 contrôle la dissociation des protéines Atg). • Le complexe Bécline1/PI3K III (phosphoinositol 3 kinase de classe III) L’activation de la PI3K III joue un rôle indispensable dans l’étape initiale de nucléation de la membrane d’isolation. L’activation de la PI3K III dépend de la formation d’un complexe multiprotéique composé notamment par bécline 1 (orthologue d’Atg6 chez les mammifères), Atg14 et la kinase p150. Ce complexe bécline 1-PI3K III participe à la fois à l’induction de l’autophagie, à la formation de l’autophagosome (par son rôle dans le recrutement de protéines importantes tels que les complexes Atg12-Atg5 et PE-Atg8/LC3) et à l’incurvation du pré-autophagosome. • Le système de conjugaison Atg12/Atg5 Atg12 et Atg5 forment un complexe auquel se lie Atg16 via Atg5. Par oligomérisation, Atg16 forme des complexes plus grands essentiels au processus d’élongation. La présence de ce complexe est également indispensable au recrutement du complexe PE-Atg8/LC3. • Le système de conjugaison PE-Atg8/LC3 LC3 (microtubule-associated protein 1 light chain 3) est un orthologue d’Atg8 chez les mammifères. LC3 subit une suite de modifications post-transcriptionnelles aboutissant à la liaison d’un phosphatidyl-éthanolamine (PE), phospholipide membranaire abondant lui permettant de s’incorporer dans la membrane du pré-autophagosome. Cette forme post-transcriptionnelle, LC3-II, semble jouer une fonction structurale permettant l’élongation et la formation de l’autophagosome mature. • Le complexe Atg9 Atg9 est une protéine transmembranaire localisée notamment dans la membrane du pré-autophagosome qui est essentielle au recrutement des complexes Atg12-Atg5 et PE-Atg8/LC3. Une fonction d’Atg9 dans le recrutement de lipides membranaires nécessaire à l’étape d’élongation est soupçonnée. Avant la formation complète de l’autophagosome, seule la fraction de LC3-II intravacuolaire reste associée à l’autophagosome, les autres protéines impliquées sont recyclées pour une utilisation ultérieure [
9,
10]. LC3-II représente ainsi un marqueur de choix pour révéler la présence d’autophagosomes [
11]. Une fois la formation de l’autophagosome terminée, la fusion avec le lysosome peut se produire. Chez les mammifères, l’étape de fusion est complexe et requiert des étapes de maturation précédant le transport de l’autophagosome vers le lysosome. Peu de choses sont connues sur ces étapes. Cependant, les microtubules, éléments du cytosquelette, semblent jouer un rôle dans cette maturation [
12]. Parmi les protéines nécessaires à la fusion, on retrouve des protéines impliquées dans d’autres types de transport vésiculaire comme certaines GTPases (Rab22, Rab24, Rab7), des protéines SNARE (SNAP and NSF attachment receptors) et la protéine NSF (N-ethylmaleimide sensitive fusion protein) [
13]. Une fois la fusion accomplie, l’environnement acide du lysosome permet la dégradation de l’autophagosome par les enzymes lysosomales produisant ainsi des macromolécules recyclables pouvant être utilisées pour la synthèse de protéines et la production d’énergie. |
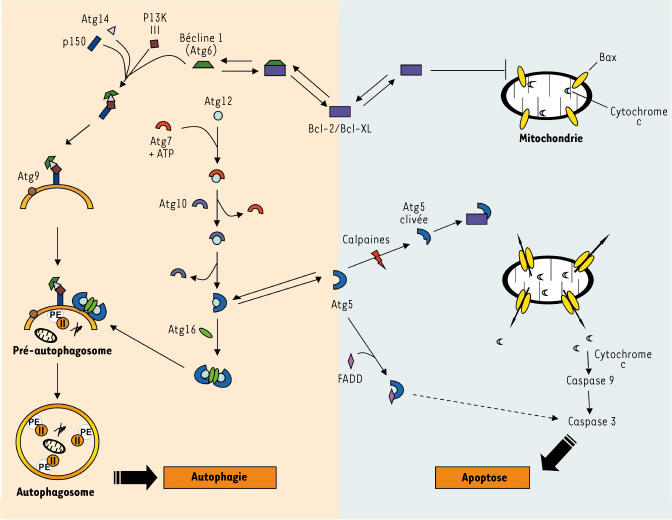
Mort cellulaire autophagique Il existe trois principaux types de mort cellulaire : le type 1 ou apoptose, le type 2 ou mort autophagique et le type 3 qui comprend plusieurs sous-types dont le mieux connu est la nécrose [
14]. L’étude morphologique des cellules lors du développement du cerveau a été à la base de la description de la mort cellulaire autophagique [14,
15] qui a été caractérisée par une présence importante d’autophagosomes, une disparition progressive des organites et une préservation de l’intégrité du cytosquelette et du noyau. Ainsi le terme « mort cellulaire autophagique » décrit une forme de mort cellulaire programmée, différente sur le plan morphologique de l’apoptose, au cours de laquelle l’activité autophagique contribue à la mort de la cellule, soit par son caractère excessif, soit par la destruction sélective d’organites indispensables au bon fonctionnement de la cellule. La compréhension du rôle de l’autophagie dans la mort cellulaire est complexe car une cellule peut présenter une forme mixte de mort avec des caractéristiques à la fois d’apoptose et d’autophagie. La question est donc de déterminer pour chaque condition si l’autophagie est présente afin de contrecarrer l’apoptose (en dégradant les mitochondries déficientes par exemple), dans ce cas l’autophagie est un mécanisme de survie, ou si l’autophagie fait partie du processus de mort cellulaire, soit comme voie de mort parallèle et différente de l’apoptose, soit comme partie intégrante du programme apoptotique. La complexité vient notamment du fait que ces deux voies sont régulées par des médiateurs communs et que l’activation de l’une peut modifier l’activité de l’autre [
16] (Figure 3). La démonstration que l’autophagie peut être directement responsable de la mort cellulaire provient d’études récentes utilisant des cellules incapables d’exécuter l’apoptose. En effet, des fibroblastes génétiquement modifiés pour ne pas exprimer les protéines pro-apoptiques Bax et Bak présentent une mort cellulaire dépendante des gènes autophagiques atg5 et bécline
1 lorsqu’ils sont exposés à des inducteurs d’apoptose [
17]. De même, l’inhibition pharmacologique des caspases, effecteurs de l’apoptose, entraîne une mort cellulaire programmée dans des cultures cellulaires qui peut être bloquée par l’inhibition des gènes atg7 et bécline 1. Ainsi, des cellules peuvent mourir de manière totalement dépendante de l’autophagie [
18]. 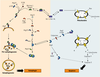 | Figure 3.
Relations entre l’autophagie et l’apoptose. L’autophagie et l’apoptose partagent des régulateurs communs. Bcl-2/Bcl-XL régulent à la fois l’apoptose et l’autophagie. Bcl-2/Bcl-XL (molécules anti-apoptotiques) inhibent l’apoptose par le blocage de l’activation de la voie intrinsèque de l’apoptose en interagissant au niveau de la mitochondrie avec les molécules pro-apoptotiques (comme Bax par exemple). Bcl-2/Bcl-XL peuvent également interagir avec le domaine BH3 de Bécline 1 et ainsi inhiber l’autophagie [
41,
42]. Dans certaines conditions, Atg5 peut intervenir dans la cascade apoptotique. Atg5 peut être clivée par l’action des calpaines [
43]. Cette forme clivée va être transloquée au niveau de la mitochondrie et interagir avec Bcl -XL pour lever son inhibition sur Bax. L’activation de Bax permet la perméabilisation de la membrane mitochondriale et ainsi la translocation du cytochrome c dans le cytoplasme et l’initiation de l’apoptose par l’activation de la caspase 9, puis de la caspase 3. De plus, Atg5 peut également interagir avec FADD ( Fas-associated protein with death domain) et stimuler la mort cellulaire caspase dépendante [
44]. |
|
Paradoxe de la macroautophagie dans le système nerveux Les neurones du cerveau sont des cellules privilégiées, hautement protégées contre certains stimulus induisant fortement l’autophagie dans d’autres organes (déplétion en nutriments par exemple) grâce à des mécanismes d’adaptation uniques et aux cellules gliales. De plus, en conditions normales, l’efficacité du processus autophagique est telle qu’il est difficile de détecter la présence d’autophagie (autophagosomes) dans des neurones sains [
19]. Autophagie et développement du système nerveux De nombreuses études ont d’abord rapporté la présence morphologique d’autophagie au cours de l’embryogenèse [ 14, 15] et suggéré un rôle de l’autophagie dans la mort cellulaire programmée associée à la morphogenèse. D’une façon générale, la mort cellulaire autophagique se produit lorsqu’un ensemble de cellules doit mourir localement alors que l’apoptose est plutôt impliquée dans l’élimination de cellules isolées. Ainsi, la mort neuronale lors de la métamorphose implique fortement l’autophagie. Par exemple les neurones de Rohon-Beard localisés dans la moelle épinière de la larve de grenouille disparaissent par mort cellulaire autophagique [14]. La maturité neuronale est un autre facteur pouvant influencer le type de mort cellulaire. En effet, il a été démontré que les motoneurones immatures chez la larve de grenouille meurent préférentiellement par apoptose alors que les motoneurones matures disparaissent par mort autophagique [14]. La mort neuronale autophagique a aussi été mise en évidence dans des neurones privés de soutien trophique rétrograde lors du développement [
20]. Récemment, un autre rôle de l’autophagie consistant en sa participation à l’élimination des cellules mortes par apoptose lors du développement a été mis en évidence. En effet, l’autophagie permettrait à ces cellules de produire l’énergie nécessaire à la production du signal leur permettant d’être phagocytées [
21]. Un exemple pour le système nerveux est le développement embryonnaire de la rétine de poulet, utilisé comme modèle de développement neuronal associant prolifération cellulaire, différenciation neuronale et mort cellulaire. Alors que les cellules ganglionnaires rétiniennes sont les premières à se différencier, des processus de mort cellulaire programmée sont induits au centre de la rétine. L’inhibition de l’autophagie par la 3-méthyladénine (3-MA) engendre l’accumulation de cellules apoptotiques liée à une réduction du niveau d’ATP [
22], démontrant ainsi le rôle indispensable de l’autophagie dans l’élimination des cadavres cellulaires. Cependant, la compréhension de la fonction spécifique de l’autophagie dans le développement du système nerveux provient, peu à peu, d’études génétiques ciblant les gènes de l’autophagie. Les embryons de souris possédant une délétion du gène codant pour Bécline 1 meurent tôt au cours de l’embryogenèse en raison d’un important retard développemental [
23]. De même, des souris n’exprimant pas Ambra1 (activating molecule in Beclin 1 regulated autophagy), une protéine favorisant l’activité du complexe Bécline 1/PI3K-III, meurent à un stade embryonnaire précoce et présentent différentes anomalies du tube neural associées à une prolifération incontrôlée des cellules nerveuses, à une accumulation de protéines ubiquitinylées et à une apoptose excessive [
24]. Les embryons n’exprimant pas Atg5 ou Atg7 survivent quant à eux à l’embryogenèse sans présenter de défaut histologique mais, lorsque l’apport nutritionnel placentaire s’interrompt à la naissance, des dysfonctionnements énergétiques sévères apparaissent et mènent à la mort en période post-natale [
25,
26]. Ces derniers travaux portent sur l’inactivation de l’autophagie dans toutes les cellules de l’organisme et affectent donc le développement et le fonctionnement d’autres organes vitaux que le cerveau, tels que le foie ou le cœur. Une meilleure compréhension du rôle de l’autophagie, spécifiquement dans le développement du système nerveux, vient de l’étude de souris chez lesqelles la délétion d’un gène autophagique est ciblée uniquement dans les neurones. Ainsi de telles souris Atg5−/−
ou Atg7−/−
développent des problèmes moteurs sévères liés à une neurodégénérescence (voir plus loin) et meurent prématurément dans les 2 mois suivant la naissance [
27,
28]. L’analyse histologique révèle une importante perte de neurones dans l’hippocampe et les cortex cérébral et cérébelleux. Ces dernières études démontrent donc le rôle essentiel de l’autophagie constitutive lors du développement du système nerveux. Nombreux sont les aspects qu’il reste encore à élucider sur le rôle développemental de l’autophagie, comme notamment ses liens avec les processus de différenciation et de prolifération. Maladies neurodégénératives : stimuler l’autophagie pour limiter la neurodégénérescence ? Une caractéristique commune de nombreuses maladies neurodégénératives est l’accumulation de protéines mutées ou toxiques et leur agrégation. Ces maladies sont désignées sous le terme de « protéinopathies » et comprennent notamment la maladie de Parkinson (agrégats d’α-synucléine), la maladie de Huntington (agrégats de polyglutamine), la maladie d’Alzheimer (agrégats du peptide β-amyloïde issus d’un mauvais clivage de la protéine APP ( Amyloid protein precursor), les taupathies (agrégats de la protéine tau hyperphosphorylée) ou encore les encéphalopathies spongiformes bovines. Dans toutes ces différentes maladies, la présence excessive d’autophagosomes dans les neurones en dégénérescence a été mise en évidence [
29]. Bien que l’autophagie puisse être impliquée dans la neurodégénérescence, son activation semblerait être d’abord un mécanisme protecteur visant à dégrader les protéines mutées ou toxiques. En effet, et comme cela a déjà été précédemment discuté dans ces colonnes [
30], l’augmentation de l’autophagie pourrait compenser une déficience de l’activité du protéasome. De même, une déficience de l’autophagie peut mener à une formation d’agrégats comme cela a été démontré chez les souris dont seuls les neurones n’expriment pas Atg5 ou Atg7 et qui développent rapidement des symptômes de maladies neurodégénératives avec la présence d’inclusions [27, 28]. L’utilisation d’agents pharmacologiques modifiant l’activité autophagique tend à confirmer le rôle protecteur de cette dernière dans les maladies neurodégénératives associées à des protéinopathies. En effet, des inhibiteurs d’autophagie, comme la 3-MA ou la bafilomycine A1, diminuent la dégradation des agrégats alors que des inducteurs d’autophagie comme la rapamycine augmentent leur élimination et diminuent ainsi leur toxicité [
31,
32]. Un très bon exemple est celui de la maladie d’Huntington qui est caractérisée par la production anormale de polymères polyQ due à la mutation du gène codant pour la protéine huntingtine, et leur accumulation sous forme d’agrégats insolubles. Le traitement à la rapamycine atténue la toxicité des polyQ non seulement dans des modèles de cultures cellulaires, mais aussi dans des modèles animaux où ce traitement permet de prévenir la neurodégénérescence et d’augmenter les performances comportementales [
33]. L’accumulation de vacuoles autophagiques observée dans les protéinopathies peut donc refléter une stimulation du flux autophagique (représentant tout le processus autophagique de la génération de l’autophagosome à la fusion/dégradation par le lysosome) mais également une déficience dans la maturation ou la dégradation des vacuoles autophagiques, par les lysosomes. Ainsi, dans le cas de la maladie d’Alzheimer, une déficience dans la fusion avec le lysosome semblerait être à l’origine de l’accumulation des vacuoles autophagiques et par conséquent du défaut de l’élimination de β-amyloïde [
34]. La stimulation de l’autophagie semblerait donc être une stratégie neuroprotectrice prometteuse pour les maladies neurodégénératives associées à des protéinopathies. Excitotoxicité et ischémie cérébrale : inhiber l’autophagie comme stratégie neuroprotectrice ? L’excitotoxicité, phénomène induit par une suractivation des récepteurs de neurotransmetteurs excitateurs (glutamate), est un mécanisme associé à la mort neuronale dans de nombreuses pathologies du système nerveux. Une induction de l’autophagie a été mise en évidence dans différents modèles d’excitotoxicité comme, par exemple, en réponse à une application d’une forte dose d’agonistes glutamatergiques (NMDA) sur des tranches organotypiques d’hippocampe [
35] ou après l’injection directe de kaïnate (KA) in vivo dans l’hippocampe de souris [
36]. La souris Lurcher, modèle génétique d’excitotoxicité dû à une mutation entraînant l’expression d’un récepteur du glutamate actif de manière constitutive, développe une dégénérescence des cellules de Purkinje associée à une présence excessive d’autophagosomes [
37]. Dans ces modèles d’excitotoxicité, le rôle de cette augmentation de l’autophagie dans la mort neuronale reste encore à définir. Cependant, une très récente étude dans un modèle d’excitotoxicité par injection de KA dans le striatum de rat a permis de démontrer une neuroprotection par la 3-MA suggérant ainsi un rôle de l’autophagie dans la médiation de la mort neuronale [
38]. D’autres arguments faisant intervenir l’autophagie dans la médiation de la mort neuronale proviennent d’études sur différents modèles d’ischémie cérébrale (pathologie dans laquelle l’excitotoxicité est très impliquée). En effet, même si le rôle de l’apoptose (comme mort cellulaire retardée) est démontré dans l’ischémie cérébrale, les stratégies utilisant des inhibiteurs de caspases se sont avérées peu ou pas efficaces dans différents modèles animaux. Ces résultats suggèrent ainsi qu’inhiber trop bas dans la cascade de signalisation apoptotique n’est pas suffisant et/ou que d’autres mécanismes peuvent être impliqués dans la médiation de la mort neuronale. Ainsi, de très récents travaux ont permis de mettre en évidence l’induction d’une forte activité autophagique, sur les plans morphologiques et biochimiques, suite à l’ischémie cérébrale [
39]. De plus, dans un modèle d’asphyxie néonatale qui combine hypoxie et ischémie, des souris génétiquement modifiées et dont les neurones n’expriment pas Atg7, présentent une résistance quasi-totale à l’hypoxie-ischémie au niveau des neurones pyramidaux de l’hippocampe par rapport à des souris sauvages [
40]. Ainsi, cette inhibition génétique (très spécifique) de l’autophagie a permis de démontrer pour la première fois que l’autophagie peut être impliquée dans la médiation de la mort neuronale suite à l’ischémie cérébrale et ouvre désormais la voie à de nouvelles stratégies thérapeutiques basées sur son inhibition. |
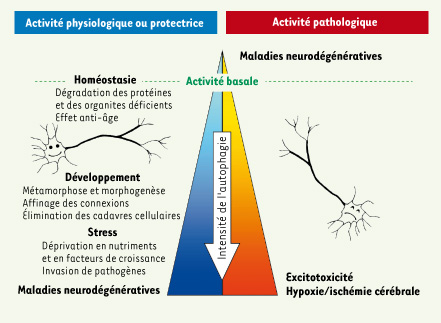
L’autophagie est un mécanisme physiologique indispensable au bon développement et au bien-être des neurones par ses différents rôles dans le contrôle de la qualité des protéines et des organites (Figure 4). Le rôle de son induction en conditions de stress est cependant beaucoup plus complexe car elle peut être impliquée aussi bien dans la survie (maladies neurodégénératives) que dans la mort neuronale (ischémie cérébrale). Ainsi chaque pathologie demande une étude spécifique du rôle joué par l’autophagie. Une meilleure compréhension des mécanismes moléculaires de l’autophagie ainsi que l’élucidation des relations entre autophagie et les différentes voies de mort cellulaire devraient permettre de savoir comment manipuler l’autophagie (stimulation ou inhibition) afin de développer de nouvelles stratégies neuroprotectrices.  | Figure 4.
Rôles de l’autophagie dans le système nerveux. L’autophagie basale ou constitutive est un processus physiologique essentiel au bien-être du neurone en participant au maintien de l’homéostasie par notamment la dégradation des protéines et organites non fonctionnels. Une diminution de l’activité autophagique basale peut être impliquée dans le développement de maladies neurodégénératives. Son induction peut avoir des rôles paradoxaux, selon la nature des stimulus et l’intensité de son activité. L’autophagie peut être impliquée dans des fonctions soit de survie (déprivation en nutriments, stress oxydatif), de mort cellulaire physiologique (lors du développement) ou pathologique (suite à l’ischémie cérébrale). |
|
Le travail de notre laboratoire sur l’autophagie est soutenu par le subside 3100A0-113925 du Fonds national suisse, les subsides de la Fondation motrice et de la Fondation Théodore Ott.
|
1. Cuervo AM. Autophagy : many paths to the same end. Mol Cell Biochem 2004; 263 : 55–72. 2. Andermarcher E, Bossis G, Farras R, et al. La degradation protéasomique : de l’adressage des protéines aux nouvelles perspectives thérapeutiques. Med Sci (Paris) 2005; 21 : 141–9. 3. Carrera AC. TOR signaling in mammals. J Cell Sci 2004; 15 ; 117: 4615–6. 4. Pattingre S, Espert L, Biard-Piechaczyk M, et al. Regulation of macroautophagy by mTOR and Beclin 1 complexes. Biochimie 2008; 90 : 313–23. 5. Sarkar S, Ravikumar B, Floto RA, et al. Rapamycin and mTOR-independent autophagy inducers ameliorate toxicity of polyglutamine-expanded huntingtin and related proteinopathies. Cell Death Differ 2009; 16 : 46–56. 6. Codogno P. Les gènes ATG et la macro-autophagie. Med Sci (Paris) 2004; 20 : 734–6. 7. Levine B, Klionsky DJ. Development by self-digestion : molecular mechanisms and biological functions of autophagy. Dev Cell 2004; 6 : 463–77. 8. Yorimitsu T, Klionsky DJ. Autophagy : molecular machinery for self-eating. Cell Death Differ 2005; 12 (suppl 2) : 1542–52. 9. Reggiori F, Tucker KA, Stromhaug PE, et al. The Atg1-Atg13 complex regulates Atg9 and Atg23 retrieval transport from the pre-autophagosomal structure. Dev Cell 2004; 6 : 79–90. 10. Legakis JE, Yen WL, Klionsky DJ. A cycling protein complex required for selective autophagy. Autophagy 2007; 3 : 422–32. 11. Kabeya Y, Mizushima N, Ueno T, et al. LC3, a mammalian homologue of yeast Apg8p, is localized in autophagosome membranes after processing. EMBO J 2000; 19 : 5720–8. 12. Aplin A, Jasionowski T, Tuttle DL, et al. Cytoskeletal elements are required for the formation and maturation of autophagic vacuoles. J Cell Physiol 1992; 152 : 458–66. 13. Wang CW, Klionsky DJ. The molecular mechanism of autophagy. Mol Med 2003; 9 : 65–76. 14. Clarke PGH. Developmental cell death : morphological diversity and multiple mechanisms. Anat Embryol (Berl) 1990; 181 : 195–213. 15. Schweichel JU, Merker HJ. The morphology of various types of cell death in prenatal tissues. Teratology 1973; 7 : 253–66. 16. Maiuri MC, Zalckvar E, Kimchi A, et al. Self-eating and self-killing : crosstalk between autophagy and apoptosis. Nat Rev Mol Cell Biol 2007; 8 : 741–52. 17. Shimizu S, Kanaseki T, Mizushima N, et al. A role of Bcl-2 family of proteins in non-apoptotic programmed cell death dependent on autophagy genes. Nat Cell Biol 2004; 12 : 1221–8. 18. Yu L, Alva A, Su H, et al. Regulation of an ATG7-beclin 1 program of autophagic cell death by caspase-8. Science 2004; 304 : 1500–2. 19. Nixon RA, Wegiel J, Kumar A, et al. Extensive involvement of autophagy in Alzheimer disease : an immuno-electron microscopy study. J Neuropathol Exp Neurol 2005; 64 : 113–22. 20. Hornung JP, Koppel H, Clarke PG. Endocytosis and autophagy in dying neurons : an ultrastructural study in chick embryos. J Comp Neurol 1989; 283 : 425–37. 21. Qu X, Zou Z, Sun Q, et al. Autophagy gene-dependent clearance of apoptotic cells during embryonic development. Cell 2007; 128 : 931–46. 22. Mellén MA, de la Rosa EJ, Boya P. The autophagic machinery is necessary for removal of cell corpses from the developing retinal neuroepithelium. Cell Death Differ 2008; 15 : 1279–90. 23. Yue Z, Jin S, Yang C, Levine AJ, et al. Beclin 1, an autophagy gene essential for early embryonic development, is a haploinsufficient tumor suppressor. Proc Natl Acad Sci USA 2003; 100 : 15077–82. 24. Fimia GM, Stoykova A, Romagnoli A, et al. Ambra1 regulates autophagy and development of the nervous system. Nature 2007; 447 : 1121–5. 25. Kuma A, Hatano M, Matsui M, et al. The role of autophagy during the early neonatal starvation period. Nature 2004; 432 : 1032–6. 26. Komatsu M, Waguri S, Ueno T, et al. Impairment of starvation-induced and constitutive autophagy in Atg7-deficient mice. J Cell Biol 2005; 169 : 425–34. 27. Komatsu M, Waguri S, Chiba T, et al. Loss of autophagy in the central nervous system causes neurodegeneration in mice. Nature 2006; 441 : 880–4. 28. Hara T, Nakamura K, Matsui M, et al. Suppression of basal autophagy in neural cells causes neurodegenerative disease in mice. Nature 2006; 441 : 885–9. 29. Rubinsztein DC, DiFiglia M, Heintz N, et al. Autophagy and its possible roles in nervous system diseases, damage and repair. Autophagy 2005; 1 : 11–22. 30. Puyal J, Ginet V, Vaslin A, et al. L’autophagie remplaçant de luxe du protéasome. Med Sci (Paris) 2008; 24 : 19–21. 31. Ravikumar B, Duden R, Rubinsztein DC. Aggregate-prone proteins with polyglutamine and polyalanine expansions are degraded by autophagy. Hum Mol Genet 2002; 11 : 1107–17. 32. Webb JL, Ravikumar B, Atkins J, et al. Alpha-Synuclein is degraded by both autophagy and the proteasome. J Biol Chem 2003; 278 : 25009–13. 33. Ravikumar B, Vacher C, Berger Z, et al. Inhibition of mTOR induces autophagy and reduces toxicity of polyglutamine expansions in fly and mouse models of Huntington disease. Nat Genet 2004; 36 : 585–95. 34. Nixon RA. Autophagy, amyloidogenesis and Alzheimer disease. J Cell Sci 2007; 120 : 4081–91. 35. Borsello T, Croquelois K, Hornung JP, et al. N-methyl-d-aspartate-triggered neuronal death in organotypic hippocampal cultures is endocytic, autophagic and mediated by the c-Jun N-terminal kinase pathway. Eur J Neurosci 2003; 18 : 473–85. 36. Shacka JJ, Lu J, Xie ZL, et al. Kainic acid induces early and transient autophagic stress in mouse hippocampus. Neurosci Lett 2007; 414 : 57–60. 37. Yue Z, Horton A, Bravin M, et al. A novel protein complex linking the delta 2 glutamate receptor and autophagy : implications for neurodegeneration in lurcher mice. Neuron 2002; 35 : 921–33. 38. Wang Y, Han R, Liang ZQ, et al. An autophagic mechanism is involved in apoptotic death of rat striatal neurons induced by the non-N-methyl-D-aspartate receptor agonist kainic acid. Autophagy 2008; 16 : 214–26. 39. Uchiyama Y, Koike M, Shibata M. Autophagic neuron death in neonatal brain ischemia/hypoxia. Autophagy 2008; 4 : 404–8. 40. Koike M, Shibata M, Tadakoshi M, et al. Inhibition of autophagy prevents hippocampal pyramidal neuron death after hypoxic-ischemic injury. Am J Pathol 2008; 172 : 454–69. 41. Pattingre S, Tassa A, Qu X, et al. Bcl-2 antiapoptotic proteins inhibit Beclin 1-dependent autophagy. Cell 2005; 122 : 927–39. 42. Maiuri MC, Le Toumelin G, Criollo A, et al. Functional and physical interaction between Bcl-X(L) and a BH3-like domain in Beclin-1. EMBO J 2007; 26 : 2527–39. 43. Yousefi S, Perozzo R, Schmid I, et al. Calpain-mediated cleavage of Atg5 switches autophagy to apoptosis. Nat Cell Biol 2006; 8 : 1124–32. 44. Pyo JO, Jang MH, Kwon YK, et al. Essential roles of Atg5 and FADD in autophagic cell death : dissection of autophagic cell death into vacuole formation and cell death. J Biol Chem 2005; 280 : 20722–9. |