| |
| Med Sci (Paris). 2009 February; 25(2): 121–122. Published online 2009 February 15. doi: 10.1051/medsci/2009252121.L’acétylation des histones Un nouveau maillon de la chaîne d’assemblage du nucléosome Hugo Wurtele,1 Qin Li,3 Hui Zhou,3 Zhiguo Zhang,3 and Alain Verreault1,2* 1Institut de recherche en immunologie et cancérologie (IRIC), Montréal, Québec, Canada. 2Département de pathologie et de biologie cellulaire, Université de Montréal, CP 6128-Succursale Centre-Ville, Montréal, Québec, H3C 3J7 Canada 3Department of Biochemistry and Molecular Biology, Mayo Clinic, College of Medicine, 200 First Street SW, Rochester, MN 55905, États-Unis MeSH keywords: Acétylation, Chromatine, Réparation de l'ADN, Épigenèse génétique, Protéines fongiques, Extinction de l'expression des gènes, Histone, Humains, Saccharomyces cerevisiae, Schizosaccharomyces |
Modifications des nucléosomes et réplication de l’ADN Chez l’humain, environ 2 mètres d’ADN sont compactés sous forme de chromatine dans un noyau cellulaire dont le diamètre est de 10 micromètres. Le nucléosome est l’élément structurel de base de la chromatine et comprend 147 paires de bases d’ADN enroulées à la surface d’un octamère d’histones. Ce dernier est formé à partir d’un tétramère (H3-H4)2 et de deux dimères d’histones H2A-H2B. Plusieurs processus pour lesquels l’accès à l’ADN est primordial, tels la transcription, la réplication et la réparation, nécessitent des altérations transitoires de la chromatine [
1] (→). Ces changements structuraux de la chromatine sont souvent facilités par des modifications covalentes et réversibles des histones. Par exemple, l’acétylation du nucléosome joue un rôle de premier plan dans l’activation de la transcription. La réplication de l’ADN pose quant à elle un défi particulier à la cellule car elle entraîne non seulement une altération des nucléosomes pré-existants, mais aussi la formation de nouveaux nucléosomes à l’échelle du génome (Figure 1A). L’assemblage de novo de nucléosomes durant la réplication est crucial puisqu’une inhibition de la synthèse d’histones lors de la phase S est létale. Durant la réplication, les chaperones d’histones CAF1 et Rtt106 déposent des formes modifiées de H3 et H4 sur l’ADN [
2]. Plusieurs lysines sont acétylées dans les domaines amino-terminaux des molécules de H3 et H4 néo-synthétisées et ce, avant même leur déposition sur l’ADN [
3,
4]. Ces modifications post-traductionnelles sont conservées de la levure à l’homme et jouent des rôles importants, bien que partiellement redondants, dans la voie d’assemblage des nucléosomes. In vivo, la mutation simultanée des lysines acétylées sur les régions amino-terminales de H3 et H4 provoque en effet une diminution marquée de l’assemblage de nucléosomes durant la phase S et une perte de viabilité [
5,
6]. Cependant, bien que cette acétylation amino-terminale des histones néo-synthétisées soit connue depuis presque 40 ans, son rôle exact dans l’assemblage de nucléosomes n’est toujours pas établi [6].
(→) m/s 2008, n° 8-9, p. 715.  | Figure 1.
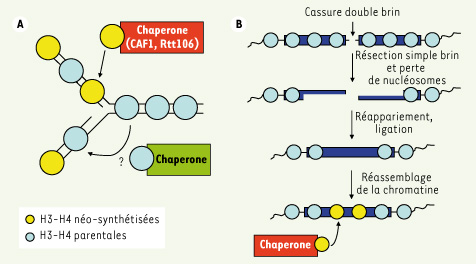
La réplication et la réparation de l’ADN nécessitent un assemblage de nucléosomes à partir d’histones néo-synthétisées. A. La duplication de la chromatine lors de la réplication requiert une biogenèse de nucléosomes. Les chaperones d’histones telles que CAF1 et Rtt106 participent à la déposition de nouvelles histones H3 et H4 derrière la fourche de réplication. Le transfert des histones pré-existantes derrière la fourche dépend lui aussi des chaperones d’histones. B. La réparation de cassures dans l’ADN par appariement d’ADN simple brin (single-strand annealing) requiert une répétition directe de deux séquences homologues (bleu foncé). Après une cassure double brin, la résection simple brin de chaque côté de la cassure permet l’appariement de séquences complémentaires et une réparation de la lésion. L’étape de résection conduit à une perte de nucléosomes. Pour restaurer la chromatine après la réparation de la lésion, des histones néo-synthétisées sont incorporées via leur interaction avec des chaperones. |
|
Fonction de l’acétylation de l’histone H3 chez la levure Chez les levures S. cerevisiae et S. pombe, toutes les molécules d’H3 déposées à travers le génome durant la réplication sont aussi acétylées sur la lysine 56 (K56) [
7,
8]. Cette lysine est située près du point d’entrée et de sortie de l’ADN, mais son acétylation n’altère que marginalement la structure du nucléosome. Néanmoins, les levures porteuses d’un allèle d’H3 non acétylable sur la lysine 56 (H3 K56R) sont extrêmement sensibles aux agents génotoxiques qui endommagent l’ADN lors de la réplication, dont plusieurs sont couramment utilisés en chimiothérapie du cancer. Une collaboration entre nos équipes de recherche a récemment permis d’élucider une fonction importante de l’acétylation d’H3 K56 chez la levure S. cerevisiae. Les nouvelles molécules d’H3 associées à CAF1 et Rtt106 sont acétylées sur K56 et cette modification facilite l’assemblage rapide de nucléosomes durant la réplication en augmentant l’affinité de CAF1 et Rtt106 pour l’histone H3, et ce aussi bien in vitro qu’in vivo [2]. En accord avec l’hypothèse selon laquelle le défaut d’assemblage de nucléosomes contribue aux phénotypes du mutant H3 K56R, des levures mutantes - dans lesquelles les chaperones CAF1 et Rtt106 sont toutes deux absentes - ont une sensibilité aux agents génotoxiques s’approchant de celle de cellules déficientes en acétylation d’H3 K56. Nos résultats sont généralement en accord avec ceux d’un second article paru simultanément. Cet article démontre que l’acétylation d’H3 K56 participe à l’assemblage de nucléosomes couplé à la réparation d’une cassure double brin dans l’ADN [
9]. Ces observations sont importantes (Figure 1B) puisqu’elles établissent que l’une des fonctions de l’acétylation des histones néo-synthétisées consiste à faciliter l’assemblage de nucléosomes couplé à la synthèse d’ADN non seulement durant la réplication, mais aussi lors de la réparation. |
Plusieurs questions restent cependant sans réponse. La double délétion de CAF1 et Rtt106 sensibilise légèrement moins les cellules mutantes aux agents génotoxiques que ne le fait la perte d’acétylation sur H3 K56. Cela suggère que certaines fonctions de l’acétylation d’H3 K56 ne sont pas médiées par ces deux protéines. D’autre part, une surexpression de la sirtuine Hst3, enzyme responsable de la désacétylation d’H3 K56 après sa déposition sur l’ADN, confère aussi aux cellules une forte sensibilité aux agents génotoxiques [
10]. Ce résultat est compatible avec l’idée que cette modification jouerait un rôle important après la déposition d’H3 dans la chromatine. Finalement, il est à propos de noter que les études réalisées jusqu’à présent suggèrent que l’acétylation de H3 K56 n’est présente qu’à faible stœchiométrie chez l’humain [
11]. Il est donc plausible que d’autres sites de modification jouent un rôle similaire à celui de l’acétylation de H3 K56 chez les mammifères. La découverte de ces sites représente une étape importante à franchir dans l’étude de l’assemblage de nucléosomes couplé à la réplication et à la réparation. |
Les travaux de recherche d’Alain Verreault et de Zhiguo Zhang sont respectivement subventionnés par les Instituts de recherche en santé du Canada (IRSC) et « National Institutes of Health » (NIH). Hugo Wurtele bénéficie d’une bourse post-doctorale de la Société de recherche sur le cancer (SRC) du Canada. L’infrastructure de recherche de l’IRIC est financée par le Fonds de la recherche en santé du Québec (FRSQ) et la Fondation canadienne pour l’innovation (FCI).
|
1. Bertin A, Mangenot S. Structure et dynamique de la particule cœur de nucléosome. Med Sci (Paris) 2008; 24 : 715–9. 2. Li Q, Zhou H, Wurtele H, et al. Acetylation of histone H3 lysine 56 regulates replication-coupled nucleosome assembly. Cell 2008; 134 : 244–55. 3. Verreault A, Kaufman PD, Kobayashi, R, et al. Nucleosome assembly by a complex of CAF-1 and acetylated histones H3/H4. Cell 1996; 87 : 95–104. 4. Zhou H, Madden BJ, Muddiman DC, et al. Chromatin assembly factor 1 interacts with histone H3 methylated at lysine 79 in the processes of epigenetic silencing and DNA repair. Biochemistry 2006; 45 : 2852–61. 5. Ma XJ, Wu, J, Altheim, BA, et al. Deposition-related sites K5/K12 in histone H4 are not required for nucleosome deposition in yeast. Proc Natl Acad Sci USA 1998; 95 : 6693–8. 6. Blackwell JS Jr, Wilkinson ST, Mosammaparast N, et al. Mutational analysis of H3 and H4 N termini reveals distinct roles in nuclear import. J Biol Chem 2007; 282 : 20142–50. 7. Masumoto H, Hawke D, Kobayashi R, et al. A role for cell-cycle-regulated histone H3 lysine 56 acetylation in the DNA damage response. Nature 2005; 436 : 294–8. 8. Xhemalce B, Miller KM, Driscoll R, et al. Regulation of histone H3 lysine 56 acetylation in Schizosaccharomyces pombe. J Biol Chem 2007; 282 : 15040–7. 9. Chen CC, Carson JJ, Feser J, et al. Acetylated lysine 56 on histone H3 drives chromatin assembly after repair and signals for the completion of repair. Cell 2008; 134 : 231–43. 10. Maas NL, Miller KM, DeFazio LG, et al. Cell cycle and checkpoint regulation of histone H3 K56 acetylation by Hst3 and Hst4. Mol Cell 2006; 23 : 109–19. 11. Garcia BA, Hake SB, Diaz RL, et al. Organismal differences in post-translational modifications in histones H3 and H4. J Biol Chem 2007; 282 : 7641–55. |