Selon le « dogme central de la biologie », les gènes encodés par l’ADN sont transcrits en des molécules d’ARN messager (ARNm) et celles-ci sont traduites en protéines (Figure 1, A-D). Ce postulat s’applique aussi bien à l’ADN des procaryotes qu’à l’ADN nucléaire, mitochondrial ou chloroplastique des eucaryotes. C’est ainsi que la séquence de l’ARNm reflète fidèlement sa matrice d’ADN, mise à part l’excision de certaines séquences internes - les introns - dans le transcrit primaire d’ARN. Or, ce principe a été remis en cause depuis une dizaine d’années, par suite, notamment, de l’exploration de génomes mitochondriaux d’eucaryotes unicellulaires.
| Figure 1.
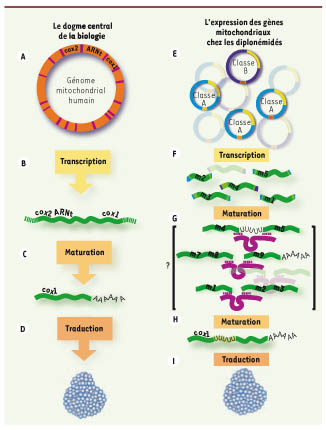
L’expression génique. Selon le dogme central de la biologie, les régions codantes de l’ADN (A) sont transcrites en ARNm qui sont ensuite traduits en protéines (B-D). Les transcrits primaires subissent généralement une maturation (C) en particulier la polyadénylation (quoique rarement dans la mitochondrie) qui mène l’ARNm à maturité. Chez D. papillatum, les chromosomes mitochondriaux des classes A et B portent chacun une petite cassette qui contient un seul fragment (module) de gène (E). Seuls quelques chromosomes du gène cox1 sont considérés ici comme exemple. La portion non codante des chromosomes se compose d’une partie partagée par tous les chromosomes (en jaune) et d’une partie commune aux chromosomes d’une même classe (en bleu). Les différentes cassettes (modules et les séquences qui les encadrent) sont transcrites individuellement avec une partie de la région constante (F). Les modules sont reliés et la séquence est éditée pour former l’ARNm (H). Ces deux processus sont présumés être dirigés par des ARN guides hypothétiques (G).
|
Qu’il s’agisse d’unicellulaires ciliés, de cellules de baobabs ou de cellules humaines, la mitochondrie sert la même fonction - production d’énergie (ATP) - à travers tout le règne des eucaryotes. L’ensemble des gènes mitochondriaux (leur génome) code essentiellement pour les protéines catalysant la respiration cellulaire et la synthèse d’ATP. Traditionnellement, on a qualifié les génomes mitochondriaux de « simples » : puisque l’on croyait qu’ils se composaient d’un seul petit chromosome circulaire contenant une douzaine de gènes. Cette conception est réductionniste puisqu’elle ne s’applique pratiquement qu’aux organismes les plus communément étudiés, soit l’organisme humain et celui d’autres mammifères. Cependant, les études portant sur des eucaryotes moins populaires montrent que tout est permis dans la façon dont se présentent les génomes mitochondriaux : nombre de gènes, nombre et forme de chromosomes et surtout la façon dont l’information génétique est transformée en protéine [ 1].
Récemment, les études du Dr W. Marande, alors étudiant en doctorat dans notre équipe, ont, une fois de plus, mis en évidence la plasticité des génomes mitochondriaux. C’est en étudiant le protiste Diplonema papillatum, du groupe des diplonémidés, qu’a été découvert un génome éclaté dont le mécanisme d’expression des gènes bouleverse le principe central de la biologie.
Le génome mitochondrial en question est extrêmement grand (> 620 kpb) et comporte une centaine de petits chromosomes circulaires de 6 kpb (classe A) et 7 kpb (classe B) (Figure 1E). De plus, chaque gène de ce génome est fragmenté en plusieurs petits morceaux (modules) où chacun des chromosomes ne porte qu’un seul module. Les chromosomes sont structurés de manière systématique et régulière et partagent les caractéristiques suivantes :
- une région non-codante représentant 95 % du chromosome, invariable à l’intérieur d’une même classe ;
- une petite cassette unique pour chaque chromosome comprenant le module génique (d’une taille moyenne de 250 pb) et de courtes régions qui encadrent le module de chaque côté.
Une douzaine de gènes ainsi morcelés ont été retrouvés dans l’ADN mitochondrial de D. papillatum. Mais l’étude [ 2] s’est surtout attardée sur le gène cox1 (spécifique à la sous-unité 1 de la cytochrome oxydase de la chaîne respiratoire) afin d’élucider le mécanisme d’expression des gènes fragmentés.
Chez ce diplonémidé, le gène cox1 est composé de neuf modules : sept sont portés par des chromosomes de classe A et deux par des chromosomes de classe B. La comparaison des séquences génomiques avec celles transcrites en ARN révèle que les modules sont assemblés post-transcriptionnellement. Plus précisément, les cassettes sont transcrites individuellement en neuf ARN différents (Figure 1F), ensuite les régions qui encadrent sont supprimées et les modules sont joints les uns aux autres pour former un ARNm mature prêt à être traduit en protéine. Ce qui intéresse maintenant les chercheurs est la découverte du mécanisme d’assemblage des modules.
C’est en s’attaquant à ce problème qu’un élément surprenant a été mis en évidence : une série de six nucléotides, tous des uridines, a été décelée entre le quatrième et le cinquième module de l’ARNm de cox1 alors qu’elle est absente de l’ADN génomique. Apparemment, les six uridines sont ajoutées secondairement au transcrit : ce phénomène rare est appelé « édition d’ARN » [ 3]. Chez D. papillatum, cette édition produit les acides aminés Leu-Phe-Ser qui sont essentiels à la protéine Cox1. Ce type d’édition impliquant seulement des uridines rappelle celui décrit auparavant chez les kinétoplastidés, le groupe frère partageant l’ancêtre commun le plus récent avec les diplonémidés (qui, avec les euglénidés, forment le grand groupe des euglénozoaires) (Figure 2).
| Figure 2.
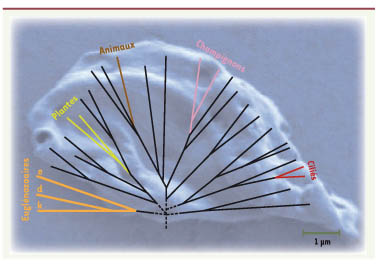
La phylogénie des eucaryotes. Les taxons les plus connus, ceux des plantes, des animaux et des champignons, sont apparentés aux protistes. Ces derniers dépassent largement en nombre et en diversité biologique les trois groupes multicellulaires précédents. L’arrière-plan montre un représentant des diplonémidés, Rhynchopus euleeides. k : kinétoplastidés ; d : diplonémidés ; e : euglénidés ; les trois groupes constituent les euglénozoaires. |
Chez les kinétoplastidés, parasites dont fait partie l’agent de la maladie du sommeil, le génome mitochondrial est également constitué de centaines de molécules circulaires [ 4]. Or leurs gènes sont encryptés à la manière des meilleurs codes secrets puisque la séquence protéique ne s’infère pas directement à partir de la séquence originale d’ADN. En effet, les transcrits sont décryptés grâce à des insertions et des délétions d’uridines, un processus dirigé par de petits ARN guides. Les Drs W. Marande et G. Burger se sont aperçus que plusieurs questions pouvaient trouver des réponses adéquates en invoquant la présence de petits ARN guides chez les diplonémidés. L’hypothèse de travail actuelle stipule que ces mêmes petits ARN présumés non seulement dirigeraient l’édition mais aussi la ligation des modules de gènes éclatés (Figure 1).
Un système si complexe d’expression de gènes suppose des coûts élevés pour la cellule, surtout du point de vue de la machinerie nécessaire à la réplication d’un génome constitué de 95 % de séquences non codantes. Cela étant dit, quel avantage évolutif confèrerait alors le maintien d’un tel appareil pendant plus d’un milliard d’années ? Les spéculations sont multiples. L’une d’elles suppose que la fragmentation des gènes permettrait le réarrangement des modules en de nouvelles combinaisons, créant ainsi de nouvelles fonctions. Cette plasticité procurerait à la cellule une adaptabilité gigantesque face aux pressions sélectives changeantes. Qui aurait pu penser que ces petits eucaryotes pourraient porter en eux un système aussi brillant qui leur permet de s’éclater face aux rigueurs de l’environnement ?