| |
| Med Sci (Paris). 2008 June; 24(6-7): 584–585. Published online 2008 June 15. doi: 10.1051/medsci/20082467584.Inactivation du chromosome X Comment une cellule sait compter jusqu’à deux X Sandrine Augui* and Edith Heard Dynamique nucléaire et plasticité du génome, UMR 218 CNRS, Institut Curie, 26, rue d’Ulm, 75248 Paris Cedex 05, France MeSH keywords: Chromosomes X humains, Extinction de l'expression des gènes, Gènes dupliqués, Humains |
Chez les mammifères, deux chromosomes sexuels de type X et Y sont responsables des différences entre mâles et femelles. Alors que les femelles portent deux chromosomes X, les mâles n’en ont qu’un seul auquel est associé un chromosome Y, beaucoup plus petit et plus pauvre en gènes. Il en résulte donc un déséquilibre entre les deux sexes quant à la quantité de gènes, donc de produits géniques (ARN et protéines), que leurs cellules contiennent. Ce déséquilibre est compensé très tôt au cours de l’embryogenèse via l’inactivation d’un des deux chromosomes X chez la femelle. En d’autres termes, dans chaque cellule de l’embryon, la quasi-totalité des gènes portés par l’un des deux chromosomes X va être transcriptionnellement éteinte. Le X à inactiver étant choisi aléatoirement et indépendamment dans chaque cellule de l’embryon, l’inactivation conduit à la formation d’individus femelles mosaïques dont toutes les cellules n’expriment pas le même chromosome X [
1,
2]. |
Détecter, compter et choisir : les étapes de l’inactivation aléatoire d’un des deux X Cet aspect aléatoire de l’inactivation implique plusieurs contraintes pour la cellule. Celle-ci doit tout d’abord déterminer le nombre de chromosomes X qu’elle contient afin de n’initier l’inactivation qu’en présence de plusieurs X (étape dite de sensing). Elle doit également compter le ratio X : autosome afin de ne conserver qu’un X actif par lot diploïde d’autosomes (comptage) et choisir lequel des deux chromosomes X sera inactivé (choix). Toutes ces étapes, comme l’initiation de l’inactivation en elle-même, sont contrôlées par un locus unique du chromosome X appelé centre d’inactivation (ou Xic). Si les limites du Xic et les différents éléments fonctionnels qu’il contient n’ont pas encore été totalement définis, il est établi que ce locus est nécessaire et suffisant à la mise en place de l’inactivation (pour revue, voir [
3]). Le Xic contient entre autres le gène clé de l’inactivation, Xist, dont l’ARN non codant recouvre le chromosome X qui lui a donné naissance, et induit son extinction. Dans les cellules de l’embryon précoce, comme dans les cellules souches embryonnaires (cellules ES) qui en sont issues, Xist est exprimé à un faible niveau à partir de ses deux allèles. Au cours de la différenciation cellulaire, ces deux allèles vont être régulés différemment : l’un, choisi aléatoirement, est surexprimé pour induire l’inactivation du chromosome qui le porte, tandis que l’autre, au niveau du X restant actif, est progressivement éteint (Figure 1).
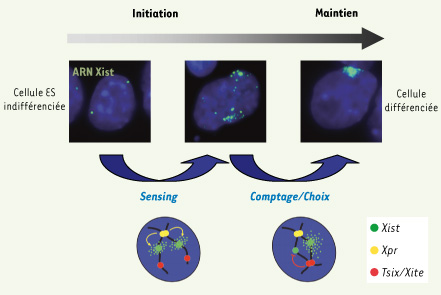
 | Figure 1.
Appariement homologue des Xic et mise en place de l’inactivation. Dans les cellules ES indifférenciées, Xist est exprimé à un faible niveau à partir de ses deux allèles. L’ARN Xist (en vert) est alors visualisable par ARN FISH (hybridation in situ en fluorescence) sous la forme de deux points ponctuels. Au tout début de la différenciation, les deux locus Xpr (représentés en rouge) s’apparient transitoirement, permettant ainsi à la cellule de détecter la présence de ses deux chromosomes X (étape de sensing) [
7]. Le gène Xist est alors activé, induisant un début d’accumulation biallélique de l’ARN en cis. Une fois Xist activé, une seconde phase d’appariement homologue des Xic permet de rendre son expression monoallélique (étapes de comptage/choix). Cette seconde phase d’appariement concerne non seulement la région Xpr mais également la région Tsix/Xite, impliquée dans la régulation négative de Xist [ 7,
10,
11]. Une fois initiée, l’inactivation est stable et maintenue au cours des divisions cellulaires. |
|
Comment compter jusqu’à deux : un nouvel élément du Xic La mise en place de cette régulation différentielle des deux allèles de Xist ainsi que les mécanismes qui la contrôlent soulèvent de nombreuses questions. L’une d’elles consiste à comprendre comment la cellule est capable de déterminer le nombre de chromosomes X qu’elle contient afin d’induire ou non cette surexpression monoallélique de Xist. S’il est établi que c’est le nombre de Xic présents dans le noyau que la cellule recense, l’introduction d’un transgène contenant Xist et son unité régulatrice Tsix/Xite en une copie dans une lignée mâle ne permet pas d’induire l’inactivation, indiquant que ceux-ci ne sont pas reconnus par la cellule comme un Xic surnuméraire [
4]. En outre, dans une lignée femelle dont l’un des X a été délété de Xist, Tsix et Xite, l’inactivation est normalement mise en place au niveau du chromosome X intact, indiquant que cette délétion ne perturbe pas l’étape de sensing [
5]. Il existe donc au sein du Xic des éléments fonctionnels nécessaires à l’étape de sensing, localisés en dehors de la région Xist/Tsix/Xite, qui restent à identifier [
6]. Notre équipe vient de mettre en évidence pour la première fois l’un de ces éléments potentiels. Localisé dans la partie proximale du Xic, ce locus nommé Xpr (X-pairing region) est capable, dans les cellules ES femelles, d’interagir en trans avec son homologue porté par le second chromosome X [7]. Nos données suggèrent que cette interaction, qui a lieu avant la mise en place de l’inactivation, permettrait aux deux chromosomes X de se prévenir mutuellement de leur présence et de rapprocher les deux allèles de Xist afin de mettre en place leur régulation différentielle. L’analyse de lignées ES femelles porteuses d’un transgène Xpr simple copie montre que celui-ci est capable d’aller à la rencontre du locus endogène, indiquant la capacité intrinsèque et unique de cette région à engendrer l’interaction. En outre, le nombre de cellules accumulant l’ARN Xist après différenciation est accru dans ces lignées femelles transgéniques, comparé à une lignée sauvage, suggérant un rôle de l’interaction homologue Xpr-Xpr dans la surexpression de Xist et donc dans l’initiation de l’inactivation (Figure 1). Cette hypothèse est corroborée par les observations faites dans des lignées ES mâles où l’introduction stable d’un transgène Xpr est fortement contre-sélectionnée. Sachant que la surexpression de Xist dans une lignée mâle est létale puisqu’elle induit l’inactivation de l’unique chromosome X, cette contre-sélection pourrait être due à l’activation anormale du gène Xist endogène via l’interaction ectopique des Xpr transgénique et endogène. En l’occurrence, l’unique clone mâle porteur d’un transgène Xpr obtenu s’est avéré instable et montre à l’état indifférencié des accumulations aberrantes de l’ARN Xist, situation qui n’est jamais observée dans des cellules mâles sauvages. |
Un nouveau mode de régulation monoallélique ? Au-delà de sa capacité à faire interagir les deux centres d’inactivation, le locus Xpr pourrait donc avoir un effet trans-activateur sur le gène Xist. Cet effet pourrait matérialiser l’étape de sensing en n’autorisant l’activation de Xist, et donc l’inactivation d’un X, qu’en présence d’au moins deux Xpr, donc de deux chromosomes X. Ce mode de recensement génique via des interactions homologues associées à une activation des gènes par effet en trans offre de très nombreuses perspectives quant à la compréhension générale des mécanismes impliqués dans la régulation monoallélique aléatoire1. Les gènes du chromosome X ne sont en effet pas les seuls à être soumis à ce type de régulation qui concerne prés de 10 % du génome humain comme l’a montré une étude récente [
8]. La découverte du locus Xpr et de son mode d’action dans la mise en place de l’inactivation du chromosome X ouvre donc un nouveau champ de recherche dans le domaine. |
Footnotes |
1. Heard E, Disteche CM. Dosage compensation in mammals: fine-tuning the expression of the X chromosome. Genes Dev 2006; 20 : 1848–67. 2. Gilgenkrantz S. Insubordination et sollicitude du chromosome X humain. Med Sci (Paris) 2005; 21 : 573–7. 3. Avner P, Heard E. X-chromosome inactivation : counting, choice and initiation. Nat Rev Genet 2001; 2 : 59–67. 4. Heard E, Mongelard F, Arnaud D, Avner P. Xist yeast artificial chromosome transgenes function as X-inactivation centers only in multicopy arrays and not as single copies. Mol Cell Biol 1999; 19 : 3156–66. 5. Monkhorst K, Jonkers I, Rentmeester E, et al. X inactivation counting and choice is a stochastic process : evidence for involvement of an X-linked activator. Cell 2008; 132 : 410–21. 6. Augui S, Heard E. Le rendez-vous des chromosomes X. Med Sci (Paris) 2006; 22 : 910–1. 7. Augui S, Filion GJ, Huart S, et al. Sensing X chromosome pairs before X inactivation via a novel X-pairing region of the Xic. Science 2007; 318 : 1632–6. 8. Gimelbrant A, Hutchinson JN, Thompson BR, Chess A. Widespread monoallelic expression on human autosomes. Science 2007; 318 : 1136–40. 9. Gabory A, Dandolo L. Épigénétique et développement : l’empreinte parentale. Med Sci (Paris) 2005; 21 : 390–5. 10. Bacher CP, Guggiari M, Brors B, et al. Transient colocalization of X-inactivation centres accompanies the initiation of X inactivation. Nat Cell Biol 2006; 8 : 293–9. 11. Xu N, Tsai CL, Lee JT. Transient homologous chromosome pairing marks the onset of X inactivation. Science 2006; 311 : 1149–52. |