La régulation précise de la transcription au cours du développement est cruciale pour l’établissement et le maintien du profil d’expression des gènes spécifiques de tissus chez les eucaryotes supérieurs. Les cellules souches embryonnaires (ES) ont la capacité d’activer tous les programmes d’expression de gènes somatiques pendant leur différenciation, et, par conséquent, sont capables de produire tous les types de cellules différenciées de l’organisme. Cette propriété, nommée pluripotence, soulève des questions importantes sur la façon dont la compétence transcriptionnelle des gènes spécifiques de tissus est maintenue dans les cellules ES. Plusieurs études montrent que, dans les cellules ES, des gènes spécifiques de tissus sont déjà « préparés » alors même que leur transcription ne se déroulera que plus tard au cours du développement. Cet état de compétence, où les gènes sont «préparés» mais ne sont pas encore exprimés, est important pour maintenir la pluripotence des cellules ES et empêcher leur différenciation [ 1, 2]. De plus, la chromatine des cellules ES est généralement considérée comme hyperdynamique [ 3]. Dans les cellules ES, certaines régions du génome contiennent des modifications dites « actives » des histones qui forment des marques précoces de compétence transcriptionnelle [ 4, 5]. Il a été montré que ces marques épigénétiques constituent des sites de recrutement de facteurs de transcription alors même que ces gènes ne sont pas encore transcrits [4, 5] (Figure 1).
| Figure 1.
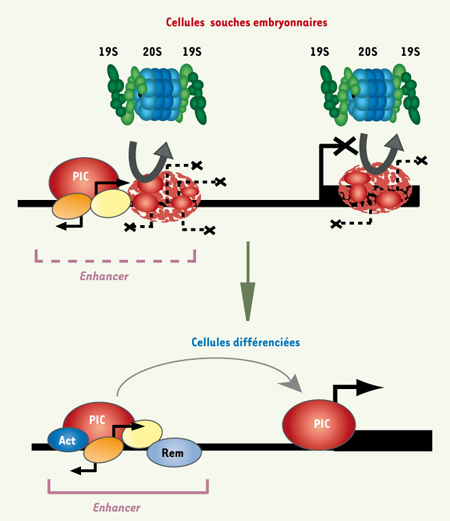
Modèle pour le contrôle de la transcription par le protéasome dans les cellules ES. Les gènes spécifiques de tissus contiennent des enhancers qui agissent comme centres de recrutement des facteurs de transcription [5]. Dans des cellules ES, le protéasome 26S (composé du noyau protéolytique 20S et du domaine régulateur 19S) cible les PIC non-spécifiques (rouge dégradé) assemblés sur des promoteurs cryptiques, pour empêcher la transcription inter- et intragénique (lignes pointillées). La conformation et/ou la composition anormale de ces PIC en feraient des cibles pour la dégradation par le protéasome. Dans les cellules différenciées, le recrutement des activateurs (Act) spécifiques de tissu au niveau des enhancers permet le remodelage de la chromatine par le recrutement des complexes de remodelage de la chromatine (Rem) et l’assemblage des PIC stables et normaux (rouge) sur les promoteurs des gènes spécifiques de tissus. Ceux-ci ne sont plus sujets à la dégradation par le protéasome. |
Les modifications de la chromatine, le recrutement des facteurs généraux de transcription (GTF) comme TFIIA-H et l’assemblage du complexe de pré-initiation (PIC) à partir des GTF sont les points de régulation principaux de la transcription, parce qu’ils indiquent où et quand la transcription par l’ARN polymérase II (Pol II) sera initiée [ 6, 7]. Dans les cellules ES, caractérisées par un environnement chromatinien permissif, le contrôle de la formation du PIC est donc encore plus crucial que dans des cellules différenciées. De plus, l’environnement permissif de la chromatine dans des cellules ES suggère l’existence de mécanismes limitant la liaison non spécifique des facteurs de transcription.
Le protéasome a été récemment impliqué dans le contrôle dynamique de la liaison des facteurs de transcription aux promoteurs des gènes actifs [ 8]. Le protéasome 26S est un complexe multiprotéique très conservé exerçant une activité protéasique et jouant un rôle central dans le contrôle de la stabilité des protéines dans les cellules eucaryotes. Le complexe 26S est organisé en deux sous-complexes, le noyau protéolytique 20S et la particule régulatrice 19S. Le complexe 19S est lui-même fait de deux composants, la base et le couvercle. La base contient des ATPases et des sous-unités non enzymatiques, et le couvercle les protéines qui semblent être responsables de la liaison spécifique des substrats [ 9].
Bien que leur chromatine soit largement ouverte et considérée comme permissive, les cellules ES ne permettent pas la transcription d’une variété de gènes codant pour des facteurs de différenciation cellulaire, ni l’initiation cryptique à partir de régions intergéniques. Ces observations nous ont incité à formuler l’hypothèse selon laquelle le protéasome pourrait contrôler, dans les cellules ES, les processus de transcription non désirés de régions intergéniques ou de gènes de différenciation [ 10]. Pour évaluer cette hypothèse, nous avons sélectionné le locus λ5-VpréB11,, qui n’est pas actif dans les cellules ES, mais est activé pendant les phases précoces de différenciation des progéniteurs des lymphocytes B. Quand des inhibiteurs chimiques du protéasome, ou des ARN interférents inhibant l’expression des sous-unités du protéasome, sont ajoutés aux cellules ES, nous avons observé une augmentation de la transcription de certaines régions intergéniques dans le locus λ5-VpréB1 dans ces cellules. Nous avons également observé que l’inhibition du protéasome dans les cellules ES déclenche l’apparition de nouveaux sites d’initiation de la transcription [10]. Cet effet n’est pas vu dans des cellules différenciées, suggérant qu’il est spécifique aux cellules ES.
Pour explorer plus en détail le rôle du protéasome dans la transcription, nous avons examiné si l’inhibition de son activité aurait un effet direct sur la liaison du PIC ou des facteurs de remodelage de la chromatine au locus λ5-VpreB1, et plus particulièrement aux régions intergéniques dont la transcription est anormale lorsque le protéasome est inhibé. En effet, nous avons constaté que les facteurs comme TBP (sous-unité du complexe TFIID qui fait partie du PIC), Brg1 (brahma-related gene 1, sous-unité du complexe de remodelage de la chromatine SWI/SNF), TRRAP (transformation/transcription domain-associated protein, sous-unité des complexes de remodelage de la chromatine STAGA/TFTC ou NuA4/TIP60), ainsi que la Pol II n’étaient liés à ces régions qu’après l’inhibition du protéasome. Nous avons ensuite démontré que les sous-unités du protéasome, aussi bien celles du 20S et du 19S, étaient liées au locus λ5-VpréB1 dans des conditions normales. De plus, nous avons constaté que ce processus était très dynamique et dépendant de l’activité du protéasome [10].
Nous avons également analysé, dans des cellules ES, les effets de l’inhibition du protéasome sur deux locus additionnels, ceux des gènes de la β-globine et de HoxD4, dont l’expression est régulée pendant le développement. Comme c’était le cas pour le locus λ5-VpréB1, l’inhibition du protéasome entraîne également une augmentation de la transcription de ces deux locus et de la liaison des facteurs de transcription, suggérant que ce mécanisme s’applique peut-être à beaucoup d’autres régions génomiques des cellules ES [10].
De plus, nos données indiquent que les activités des promoteurs sont sensiblement plus fortes dans les cellules ES que cela n’avait été précédemment décrit. Mais cette transcription est continuellement réprimée par le protéasome qui enlève les PIC assemblés sur des promoteurs cryptiques (Figure 1). Nous faisons l’hypothèse que, dans des régions inter-ou intragéniques des cellules ES, les PIC ont une conformation et/ou une composition modifiées par rapport à celles des PIC normaux ; ces derniers peuvent être stabilisés par des activateurs spécifiques dans des cellules différenciées (Figure 1), alors que la structure anormale et non-spécifique de ces PIC dans les cellules indifférenciées de type ES en ferait des cibles pour la dégradation par le protéasome [ 11].
La répression de la transcription qu’exerce de façon non spécifique le protéasome ajoute une nouvelle dimension au contrôle de la pluripotence des cellules ES et identifie une fonction entièrement nouvelle pour le protéasome. Nos résultats suggèrent que les régions génomiques contenant des gènes spécifiques de lignées de différenciation sont dans un état fortement dynamique dans les cellules ES : des facteurs se lient en permanence aux séquences régulatrices, mais sont enlevés par le protéasome. Ce contrôle est susceptible de maintenir les gènes spécifiques de tissus dans un état transcriptionnellement compétent, sans pour cela qu’ils soient transcrits, permettant ainsi leur activation rapide et efficace lors de la différenciation cellulaire.