Les progrès réalisés dans le domaine du séquençage de l’ADN ont permis d’obtenir les séquences des génomes complets de l’homme et de différents organismes modèles (souris, drosophile, rat…). La prochaine étape dans l’analyse du génome consiste à déterminer les fonctions des protéines codées par les gènes identifiés et ainsi à mieux comprendre les mécanismes conduisant à l’apparition de pathologies (diabète, obésité, maladies neurodégénératives par exemple) chez l’homme. L’invalidation d’un gène par mutagenèse chez l’animal, suivie de l’identification des fonctions altérées par la mutation, est l’une des approches utilisées pour connaître la fonction d’une cible d’intérêt.
Les progrès réalisés dans le domaine du séquençage de l’ADN ont permis d’obtenir les séquences des génomes complets de l’homme et de différents organismes modèles (souris, drosophile, rat…). La prochaine étape dans l’analyse du génome consiste à déterminer les fonctions des protéines codées par les gènes identifiés et ainsi à mieux comprendre les mécanismes conduisant à l’apparition de pathologies (diabète, obésité, maladies neurodégénératives par exemple) chez l’homme. L’invalidation d’un gène par mutagenèse chez l’animal, suivie de l’identification des fonctions altérées par la mutation, est l’une des approches utilisées pour connaître la fonction d’une cible d’intérêt.
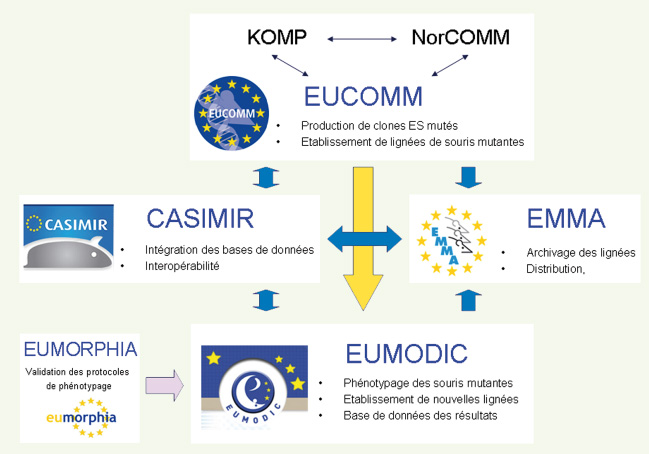
| Figure 1.
Intégration des différents programmes européens de génomique fonctionnelle de la souris. Les lignées de souris mutantes produites par EUCOMM (http://www.eucomm.org) seront phénotypées dans le cadre du projet EUMODIC (http://www.eumodic.org) et archivées et distribuées par EMMA (http://www.emmanet.org) Les stratégies de mise en commun des bases de données génomique et phénotypique seront développées par le programme CASIMIR (http://www.casimir.org.uk). Le programme EUMORPHIA (http://www.eumorphia.org), aujourd’hui terminé, a permis de standardiser les protocoles de phénotypage entre les quatre cliniques européennes (GSF, ICS-MCI, MRC-Harwell, Sanger Institute). Des lignées de souris mutantes seront également établies par les programmes américain KOMP (http://www.knockoutmouse.org) et canadien NorCOMM (http://norcomm.phenogenomics.ca/index.com). La coordination des activités de ces trois programmes sera assurée par l’IKMC. |
La souris est un organisme modèle de choix pour ce faire, pour les raisons suivantes : (1) 99 % des gènes présents chez la souris ont un équivalent chez l’homme ; (2) le génome de la souris est facilement manipulable ; (3) les progrès récents réalisés dans la manipulation du génome permettent de créer des modèles murins de pathologies humaines pertinents ; (4) les fonctions biologiques et biochimiques de la souris sont proches de celles de l’homme [ 1, 2].
Il a été décidé simultanément en Europe [ 3] et en Amérique du Nord [ 4] de procéder à la mutagenèse systématique de l’ensemble des gènes murins codant des protéines dans les cellules souches embryonnaires de souris. Trois programmes de recherche ont été mis en place pour réaliser cette mutagenèse à large échelle, KOMP (Knockout Mouse Project), EUCOMM (EUropean COnditional Mouse Mutagenesis program) et NorCOMM (North american COnditional Mouse Mutagenesis project) [ 5].
Deux approches ont été choisies pour introduire des mutations dans le génome : le piégeage de gènes (gene trapping) et le ciblage de gènes (gene targeting).
La technique de piégeage de gènes repose sur l’insertion au hasard dans le génome d’une construction d’ADN avec un site accepteur d’épissage et une cassette de sélection. L’insertion d’une telle construction dans l’intron d’un gène conduira à la production de transcrits tronqués et donc à l’inactivation du gène d’intérêt. Cette technique présente l’avantage d’être peu onéreuse et nécessite la construction de quelques vecteurs uniquement. Cependant, elle requiert la caractérisation du gène muté au site d’intégration du vecteur et elle n’est pas applicable à tous les gènes.
Le ciblage de gènes est une technique directe qui consiste à introduire par recombinaison homologue une mutation déterminée dans un gène d’intérêt. L’avantage est de pouvoir cibler n’importe quel gène/n’importe quel type de mutation. Cela implique de produire une nouvelle construction pour chaque gène/mutation.
Quelle que soit l’approche choisie, la grande majorité des mutations introduites dans le cadre de ces programmes le seront de façon conditionnelle, afin de pouvoir réaliser des mutations contrôlées dans le temps et dans l’espace des gènes d’intérêt avec le système Cre/LoxP [ 6, 7]. La mutagenèse conditionnelle permet d’étudier chez l’animal adulte la fonction de gènes essentiels au développement embryonnaire, ou encore de cibler le rôle d’un gène dans un organe de l’animal.
Pour chacun des trois programmes, des clones de cellules souches embryonnaires (cellules ES) mutantes pour un gène vont être établis en utilisant l’une des deux approches. Une liste de gènes à cibler en priorité a été établie par EUCOMM. L’IKMC (International Knock-out Mouse Consortium), récemment créé, coordonnera les activités des trois programmes de manière à éviter la redondance du travail et à assurer le ciblage du plus grand nombre possible de gènes [ 8]. À l’issue de ces programmes (en 2010), il est prévu d’avoir approximativement 40 000 clones ES. Une partie de ces clones sera utilisée pour établir des lignées de souris mutantes.
Le fonds génétique est un paramètre fondamental pour l’analyse du phénotype de souris mutantes. Historiquement les souris mutantes étaient établies en utilisant des cellules ES de la lignée 129. Cette lignée, adéquate pour la recombinaison homologue, peut difficilement être utilisée en immunologie, en comportement et en physiologie. Les programmes européens prévoient donc l’établissement des lignées mutantes de préférence sur un fond génétique C57BL/6N. Chaque lignée de souris mutante générée sera archivée dans le cadre du programme européen EMMA (European Mouse Mutant Archive) et mise à disposition de la communauté scientifique internationale.
Les laboratoires demandant l’établissement de lignées de souris mutantes réalisent le plus souvent une étude phénotypique des animaux ciblée sur un domaine d’expertise précis, et n’effectuent pas d’étude couvrant un large spectre de fonctions. Le programme européen EUMODIC (EUropean MOuse DIsease Clinic), qui a débuté en 2007, est un programme pilote de phénotypage systématique utilisant des procédures standardisées d’analyse des principales fonctions biologiques. Quatre centres européens (GSF - National Research Center for Environment and Health Gmbh, Munich/Neuherberg Germany ; Institut Clinique de la Souris - Mouse Clinical Institute, Strasbourg France ; Medical Research Council, Mammalian Genetics Unit, Harwell, UK ; Wellcome Trust Sanger Institute, Hinxton Cambridge, UK) vont réaliser le phénotypage de 650 lignées de souris mutantes. La standardisation des procédures a été établie dans le cadre du programme européen EUMORPHIA [ 9] (European Union MOuse Research for Public Health and Industrial Applications). Les tests incluent des analyses biochimiques et métaboliques, des tests comportementaux, l’étude du système cardiovasculaire, des analyses du squelette, complétées ou non par des études histologiques. Certaines de ces lignées feront l’objet d’un phénotypage secondaire approfondi ciblant une fonction biologique précise ; les analyses seront réalisées par des centres de phénotypage secondaires.
L’exploitation des données obtenues par ces programmes nécessite l’élaboration d’une stratégie d’intégration et d’interopérabilité des bases de données à l’échelle européenne, stratégie développée par le programme CASIMIR (Coordination and Sustainability of International Mouse Informatics Resources). L’ensemble des résultats de ces tests sera ainsi exploitable par la communauté scientifique.
Les programmes européens EUCOMM, EUMODIC, EMMA et CASIMIR s’inscrivent dans une logique de production à grande échelle de modèles murins génétiquement modifiés et d’étude phénotypique systématique de ces animaux. Ces programmes vont générer à moindre coût une multitude d’informations pertinentes accessible à l’ensemble de la communauté scientifique internationale, démontrant la valeur des programmes européens intégrés. Cette source d’informations liée à l’expertise des laboratoires européens représente une base précieuse pour les recherches futures de l’ère post-génomique, avec pour but ultime la découverte de nouvelles cibles thérapeutiques et le développement de médicaments innovants pour le traitement des maladies humaines.