| |
| Med Sci (Paris). 2007 October; 23(10): 850–856. Published online 2007 October 15. doi: 10.1051/medsci/20072310850.Détruisez ce message (ARN) après l’avoir lu ! Sylvie Camier and Bertrand Séraphin* Équipe Labellisée la Ligue, Centre de Génétique Moléculaire, CNRS UPR2167, associée à l’Université Pierre et Marie Curie, Avenue de la terrasse, 91198 Gif-sur-Yvette, France |
L’expression de l’information génétique est un processus essentiel dont la régulation permet aux cellules de s’adapter à de nouvelles conditions, de moduler leur croissance ou de se différencier. Chez les organismes eucaryotes, l’expression des gènes codant les protéines est complexe et fait intervenir de multiples étapes dans les deux principaux compartiments cellulaires [
1] :
-
transcription des gènes en pré-ARN messagers (pré-ARNm) ;
-
maturation des pré-ARNm en ARN messagers (ARNm), leur transport et éventuellement leur localisation dans le cytoplasme ;
-
traduction des ARNm ;
-
et enfin dégradation des ARNm.
La dégradation des ARNm a longtemps été considérée comme une simple étape d’élimination des ARNm. Au cours des dernières années, cette vision a été totalement remise en cause. Les temps de demi-vie des ARNm, révélateurs de leur vitesse de dégradation, peuvent varier énormément. Ainsi chez les mammifères, ils varient de 15 minutes (ARNm c-fos) à 24 heures (ARNm de la globine), montrant que l’élimination des ARN est un processus spécifique et non un processus par défaut [
2]. La dégradation des ARNm est essentielle. En l’absence d’une dégradation rapide des ARNm, une régulation rapide de la production de protéines par la seule induction transcriptionnelle serait impossible. Finalement, de nombreux exemples de dégradation régulée ont maintenant été décrits, attestant de l’importance de ce phénomène dans le contrôle général de la synthèse des protéines. Dans cet article, nous décrivons les principales voies de dégradation des ARNm chez les eucaryotes. Nous montrons le rôle de la dégradation en tant que mécanisme de surveillance de la qualité des ARNm, et son rôle à part entière dans la régulation de l’expression des gènes. |
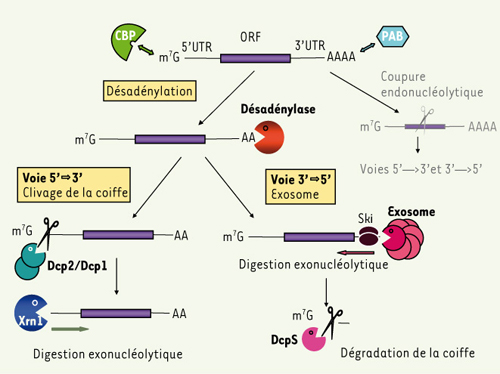
Les acteurs de la dégradation Que de précautions pour manipuler les ARNm au laboratoire afin qu’ils ne soient pas immédiatement dégradés par les ribonucléases ! Et pourtant, dans les cellules, la dégradation des ARNm n’est pas une réaction par défaut, mais un processus actif hautement contrôlé. Dans la cellule, plusieurs facteurs contribuent à la protection des ARNm contre l’action des ribonucléases. Lors de sa synthèse, l’ARNm subit des modifications à ses extrémités 5’ et 3’ : ajouts d’une coiffe et d’une queue poly(A). Ces marques, liées par des protéines spécifiques, les cap-binding protein (CBP) et Poly(A)-binding protein (PAB) le protègent de l’action des exoribonucléases. L’ARNm est également associé à d’autres protéines au sein de larges particules ribonucléoprotéiques (mRNP) qui notamment le protègent de coupures endonucléolytiques non spécifiques. La dégradation d’un ARNm résulte d’un profond remaniement de la mRNP, déclenché par des signaux spécifiques et qui conduit au recrutement des enzymes et activateurs de la dégradation (Figure 1) [
3]. 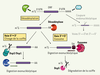 | Figure 1.
Les principales voies cytoplasmiques de dégradation des ARNm normaux. L’événement initial le plus fréquent est une désadénylation. Le corps de l’ARNm est ensuite dégradé par une des 2 voies : 5’→3’ ou 3’→5’. Dans le cas de la voie 5’→3’, la désadénylation est suivie du clivage de la coiffe par l’enzyme Dcp2/Dcp1 puis d’une digestion exonucléolytique par Xrn1. Dans le cas de la voie 3’→5’, la désadénylation est suivie du recrutement du complexe Ski et d’une digestion exonucléolytique par l’exosome ; la coiffe est finalement dégradée par l’enzyme DcpS. En grisé est représentée une voie plus rare, amorcée par une coupure endonucléolytique de l’ARNm, suivie des voies classiques. |
Les diverses enzymes de la dégradation Les voies décrites ci-dessous ont été généralement mises en évidence chez la levure S. cerevisiae et sont, pour l’essentiel, conservées chez les eucaryotes supérieurs. Désadénylation La dégradation cytoplasmique des ARNm chez les eucaryotes débute, le plus souvent, par un raccourcissement de la queue poly(A), on parle de désadénylation. Cette étape est limitante et détermine en grande partie la vitesse de dégradation de l’ARNm. Deux complexes protéiques cytoplasmiques possèdent une activité de désadénylation :
-
le complexe Pan2/Pan3, activé par la PAB associée à la queue poly(A). Il semble intervenir lors d’une étape initiale de désadénylation rapide [
4,
5].
-
le complexe Ccr4/Not, majoritaire chez les mammifères, la drosophile et la levure, inhibé par la PAB. Il contient 2 exonucléases, Ccr4 et Pop2 (ou Caf1) [
6–
9].
L’étape de désadénylation n’inactive pas définitivement l’ARN, celui-ci peut éventuellement être polyadénylé de nouveau ou, dans certains cas, stabilisé par des protéines spécifiques (chez les métazoaires). Cependant le plus souvent, lorsqu’un certain nombre de résidus A restant sur l’ARNm est atteint, de l’ordre de 10 chez la levure ou 30-60 chez les mammifères, l’ARNm s’engage dans une dégradation rapide et irréversible. Deux voies sont possibles pour cette seconde étape : la voie 5’—>3’ ou la voie 3’—>5’, selon le sens de dégradation. Les voies 5’→3’ et 3’→5’ Dans la voie 5’→3’, après désadénylation, la mRNP subit un remodelage conduisant au recrutement et à l’activation de l’enzyme de clivage de la coiffe en 5’ (complexe Dcp1/Dcp2). C’est une étape irréversible, qui fait intervenir de nombreux activateurs et régulateurs [
10,
11]. L’ARN clivé est ensuite dégradé de façon processive par l’exonucléase Xrn1 dans le sens 5’→3’. Dans la voie 3’→5’, la désadénylation est suivie du recrutement d’un complexe exonucléolytique appelé exosome, via l’association des protéines Ski. L’exosome est un complexe d’une dizaine de sous-unités catalysant une digestion de 3’→5’ [
12–
14]. La voie 3’→5’ se termine avec l’hydrolyse de la coiffe par l’enzyme DcpS. Cette même enzyme hydrolyse également la coiffe libérée lors de la dégradation 5’→3’ [
15,
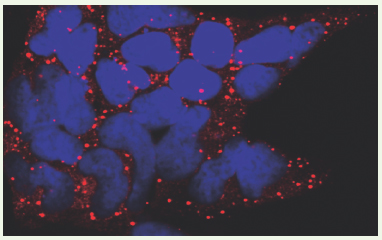
16]. L’exosome existe aussi sous une forme nucléaire, associé à des protéines spécifiques (comme Rrp6). Il intervient alors dans la maturation ou l’élimination de certains ARN codants et non codants. Coupures endonucléolytiques Dans certains cas, la dégradation peut être amorcée par une coupure endonucléolytique, qui donne naissance à 2 fragments d’ARN : un fragment 5’ « coiffé », dépourvu de queue poly(A), pris en charge par l’exosome, et un fragment 3’ sans coiffe pris en charge par l’exonucléase Xrn1. Les endonucléases impliquées restent mal connues, à l’exception de celles dirigées par de petits ARN complémentaires de l’ARNm cible (voir plus loin). Sites de dégradation De nombreuses études récentes ont mis en évidence dans le cytoplasme des cellules de nombreux organismes (mammifères, drosophile, levure) des sites (ou foci) d’accumulation de protéines impliquées dans le métabolisme des ARNm (Figure 2) [
17,
18]. Ces sites qui portent les noms divers de P-bodies ( processing bodies),
Dcp-bodies ( decapping bodies)… contiennent en particulier des acteurs de la voie de dégradation 5’→3’ (Dcp2, Dcp1, Xrn1…). Ces structures dynamiques contiennent également des régulateurs de la traduction, des ARNm non traduits, des composants de la machinerie d’interférence ARN et du NMD ( voir plus loin [
19]). 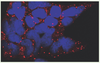 | Figure 2.
Structures cytoplasmiques liées à la dégradation des ARN messagers dans des cellules humaines en culture. L’activateur de clivage de la coiffe, Dcp1 est détecté dans des structures cytoplasmiques liées à la dégradation des ARN messagers et au stockage des ARN messagers non-traduits par immunofluorescence, en rouge. Les noyaux sont marqués par l’iodure de propidium, en bleu (© photo reproduite avec l’aimable autorisation de Nicolas Cougot). |
Les enzymes et activateurs de la dégradation ne sont pas uniquement localisés dans ces foci, on les trouve également diffus dans le cytoplasme. Certaines enzymes comme l’exosome, ou des désadénylases, semblent être localisées de manière diffuse dans le cytoplasme et absentes des foci. La proportion de dégradation totale s’effectuant dans les foci cytoplasmiques et la localisation intracellulaire des autres évènements de dégradation restent à déterminer, ce qui pose la question de l’organisation spatiale de la dégradation des ARNm dans les cellules. |
Contrôle de qualité des ARNm Un rôle majeur de la dégradation est l’élimination des ARNm dont la maturation est incorrecte ou qui contiennent des erreurs. Les systèmes de surveillance de la qualité des ARNm se répartissent en 2 grandes catégories selon qu’ils font intervenir ou non la traduction des ARNm. Dans le noyau Un premier niveau de contrôle détecte, dans le noyau, la présence de défauts de maturation (absence de coiffe, erreurs d’épissage, défauts dans la queue poly(A)…), qui s’accompagnent souvent de défauts d’export vers le cytoplasme [
20]. Des études récentes chez la levure montrent que ces ARNm déficients sont dégradés par l’exosome nucléaire, à la suite de leur polyadénylation par une poly(A) polymérase spécifique (complexe TRAMP). Dans ce cas, la polyadénylation n’est plus une marque protectrice mais un signal de dégradation, comme cela est observé pour les ARN bactériens. Le complexe TRAMP est aussi impliqué dans la dégradation de formes aberrantes d’ARN non codants et de transcrits cryptiques CUT (cryptic unstable transcripts) synthétisés par l’ARN polymérase II à partir de régions intergéniques du génome de levure et probablement d’autres génomes eucaryotes [
21–
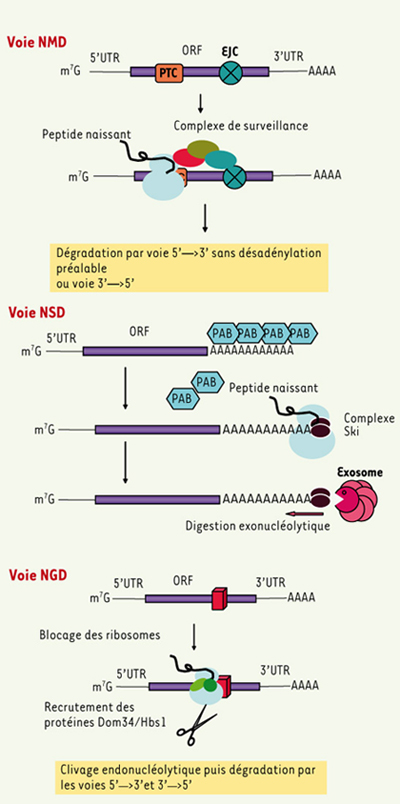
23]. Dans le cytoplasme Le second niveau de contrôle est cytoplasmique. Il est connecté à la traduction et permet l’élimination d’ARNm qui conduiraient à la synthèse de protéines aberrantes, potentiellement délétères pour la cellule. Trois types d’ARNm aberrants sont concernés (Figure 3) : les ARNm possédant un codon stop précoce (voie NMD pour non-sense-mediated decay), les ARNm ne possédant pas de codon stop (voie NSD pour non-stop decay) et les ARNm sur lesquels les ribosomes sont bloqués, piégeant ainsi les facteurs de traduction (voie NGD pour no-go decay).  | Figure 3.
Les principales voies cytoplasmiques de surveillance de la qualité des ARNm. Voie NMD. La voie NMD représentée est celle observée chez les mammifères, elle fait intervenir l’EJC (complexe déposé au niveau des jonctions exon-exon) dans le mécanisme de reconnaissance d’un codon stop précoce (PTC, premature termination codon). La reconnaissance du PTC conduit à l’assemblage du complexe de surveillance qui recrute les enzymes de dégradation. Voies NSD et NGD. En l’absence de codon stop (voie NSD) ce qui conduit à un blocage du ribosome à l’extrémité de l’ARNm, ou lorsque le ribosome est bloqué sur l’ARNm en cours de traduction (voie NGD, par exemple, à la suite de la présence d’une structure secondaire), les complexes Ski ou Dom34/Hbs1 se lient à l’ARNm et recrutent les enzymes de dégradation : exosome ou endonucléase (non identifiée à ce jour). |
La voie NMD La présence d’un codon stop précoce conduit, par un mécanisme non encore élucidé, à l’assemblage d’un complexe de surveillance comprenant 3 protéines conservées (Upf1, Upf2 et Upf3) [
24]. Chez les métazoaires, des protéines supplémentaires (protéines Smg) et une régulation par phosphorylation sont nécessaires. L’assemblage du complexe de surveillance conduit au recrutement des enzymes de dégradation. Chez l’homme et la levure, les 2 voies classiques sont activées : clivage de la coiffe et action de l’exonucléase Xrn1 sans désadénylation préalable, ou désadénylation puis dégradation par l’exosome. Chez la drosophile en revanche, il y a d’abord coupure endonucléolytique du message au niveau du codon stop puis élimination des fragments résultants par les voies classiques 5’→3’ et 3’→5’. Les mécanismes permettant de différencier un codon stop précoce du codon stop normal semblent très différents selon les organismes. Chez les mammifères, à chaque événement d’épissage, un complexe protéique appelé EJC (exon-junction complex) est déposé au niveau de la jonction exon-exon [
25]. Le codon stop étant très majoritairement localisé dans le dernier exon, la présence d’un codon stop en amont d’une jonction exon-exon suggère que ce codon serait précoce. De fait la présence d’un complexe EJC en aval d’un codon stop est perçue comme un signal de dégradation de l’ARNm correspondant. D’autres mécanismes de reconnaissance d’un codon stop précoce incluent la présence d’une longue région 3’ non traduite (région 3’ UTR ou 3’-untranslated region), de certaines séquences spécifiques en aval du codon stop… Il est intéressant de noter que la voie NMD ne détruit pas uniquement des ARNm aberrants mais aussi des ARNm « normaux » (par exemple des ARNm possédant dans leur région 5’ une très courte séquence codante ou uORF (upstream open-reading frame) et participe ainsi à la régulation de la dégradation de ces ARNm. Les voies NSD et NGD La voie NSD [
26] décrite chez la levure et les mammifères fait intervenir les protéines Ski. Celles-ci se lient à l’extrémité 3’ de l’ARNm dépourvu de codon stop et sur laquelle se retrouve un ribosome bloqué, puis recrutent l’exosome. La découverte d’une voie NGD chez la levure est très récente. La pause des ribosomes sur un site interne de l’ARNm conduit à son clivage endonucléolytique [
27]. Les 2 fragments d’ARN résultants sont dégradés par les voies classiques. |
La dégradation est un phénomène contrôlé Au cours de sa vie, un ARNm s’associe au sein de diverses mRNP avec des protéines qui vont déterminer son sort : maturation, export, localisation, stockage, traduction (éventuellement plusieurs fois), dégradation. Toutes ces étapes sont contrôlées, régulées et connectées entre elles. La dégradation est, en particulier, fortement connectée à la traduction. Ainsi, les protéines qui lient la coiffe et la queue poly(A) sont à la fois nécessaires à une traduction efficace et pour protéger l’ARNm de la dégradation. On a également observé que certains ARNm dont la traduction est inhibée par des protéines spécifiques se retrouvent stockés dans les foci de dégradation [
28]. Ce stockage est réversible, l’ARNm pouvant ensuite être de nouveau traduit, ou bien dégradé. Des études globales ont montré que la part de la dégradation dans la régulation des quantités d’ARNm peut être considérable. Par exemple, lors de l’activation des lymphocytes T (réponse immunitaire), 50 % des effets observés sur les quantités d’ARNm sont dus à une régulation de leur stabilité [
29]. Protéines régulatrices La plupart des protéines régulant la dégradation des ARNm se fixent sur l’ARNm lui-même via des séquences spécifiques situées dans les régions 5’ UTR, 3’ UTR ou parfois dans la région codante. Fréquemment ces protéines activent la dégradation par une accélération de la désadénylation. Le nombre de ces régulateurs est trop important pour permettre leur description de manière exhaustive. Deux exemples bien caractérisés ont été choisis pour illustrer ce propos. Protéines Puf Les protéines Puf représentent une famille de protéines reconnaissant des séquences spécifiques trouvées dans certains ARNm [
30]. Ces ARNm peuvent contenir plusieurs sites de liaison identiques ou différents, augmentant ou modulant leur dégradation. Des analyses globales ont révélé qu’un type de protéine Puf reconnaît généralement une classe d’ARNm codant pour des protéines de fonction voisine ou de localisation identique (par exemple Puf3 de levure et la mitochondrie). Certaines protéines Puf accélèrent la déadénylation et le clivage de la coiffe [
31]. Protéines se liant aux séquences ARE (AU-rich elements) Chez les vertébrés, certains ARNm, souvent de demi-vie courte comme les ARNm des cyclines, cytokines, facteurs de croissance, protooncogènes… possèdent des séquences riches en résidus adénine et uracile dans leur région 3’ UTR [
32]. Ces séquences sont reconnues par des protéines déstabilisatrices (tristétraproline, BRF1, AUF-1, KSRP…) qui agissent en recrutant les enzymes de dégradation (désadénylases, exosome…) [
33]. Dans d’autres conditions, elles peuvent lier des protéines stabilisatrices qui entrent en compétition avec les protéines déstabilisatrices (comme Hu-R de la famille ELAV, [
34]) ou pourraient inhiber directement la machinerie de dégradation. Certaines de ces protéines régulent parfois aussi la traduction, renforçant encore les liens fonctionnels entre dégradation et traduction. Petits ARN régulateurs La découverte de l’interférence ARN, qui valut en 2006 le prix Nobel à A. Fire et C. Mello [
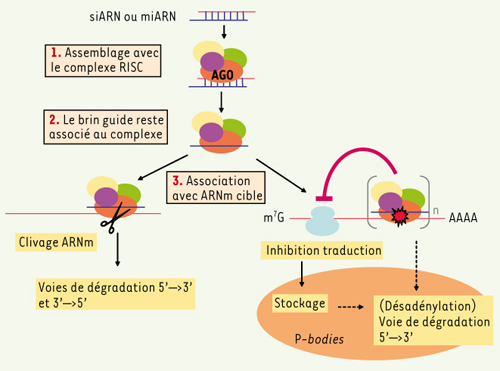
39], a révélé l’existence d’un nouveau mode de dégradation des ARN via l’association de petits ARN (Figure 4) [
35–
37]. Cette dégradation est différente de celle, non spécifique, observée chez les mammifères lors de la réponse antivirale (réponse interféron), déclenchée par de longs ARN double-brin et faisant intervenir la protéine kinase PKR et la RNase L [
38]. Elle est absente chez la levure S. cerevisiae. 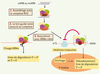 | Figure 4.
Les différents modes d’interférence ARN cytoplasmique. Dans un premier temps, un petit ARN double-brin (d’origine exogène ou endogène) s’associe au complexe RISC, puis un seul brin reste associé et guide le complexe vers un ARNm-cible. Deux cas de figure sont ensuite observés : soit l’endonucléase Argonaute est activée, conduisant à une coupure de l’ARNm et à la dégradation des fragments résultants par les voies classiques, soit l’endonucléase n’est pas activée, il y a alors inhibition de la traduction de l’ARNm suivie éventuellement d’une dégradation par les voies classiques, dans les focus de dégradation. |
Des petits ARN double-brin, d’origine exogène (le plus souvent siARN, small interfering ARN) ou endogène (le plus souvent miARN, microARN), sont dans un premier temps pris en charge par le complexe RISC (RNA-induced silencing complex). Un seul des deux brins, le brin-guide, reste associé au complexe et va s’hybrider à son ARNm cible. Le sort de l’ARNm dépend ensuite de la complémentarité entre brin-guide et ARNm, et de l’organisme (voir Encadré).
Dans le cas d’une complémentarité totale (miARN de plantes, ou siARN), il y a clivage endonucléolytique par la protéine Argonaute du complexe RISC puis dégradation des fragments résultants par les voies classiques. Pour les miARN de la plupart des animaux, la complémentarité avec l’ARNm cible est partielle et celui-ci n’est pas clivé. On le retrouve dans les focus de dégradation, où il peut subir deux sorts : inhibition de sa traduction ou dégradation par les voies classiques via une accélération de la désadénylation puis clivage de la coiffe. |
De plus en plus la dégradation des ARNm apparaît comme une étape hautement contrôlée jouant un rôle primordial dans l’expression des gènes. Tout comme la synthèse d’ARN est le résultat des actions combinées des facteurs liant l’ADN et/ou affectant la structure de la chromatine, la dégradation d’un ARNm est le résultat des interactions entre cet ARNm et de multiples facteurs. Les études récentes ont permis d’identifier les principaux acteurs impliqués dans ce processus. Il est maintenant important de comprendre comment leurs actions sont concertées. Ces travaux permettront de découvrir comment les voies de signalisation cellulaires utilisent la dégradation des ARNm pour contrôler le fonctionnement de la cellule. Ces recherches auront aussi pour but d’élucider la coordination des diverses fonctions cellulaires avec la dégradation, en particulier la traduction, tant du point de vue mécanistique que du point de vue spatial.

|
1. Alberts A, Johnson A, Lewis J, et al.
Molecular biology of the cell, 4th ed. New York : Garland Science, 2002. 2. Meyer S, Temme C, Wahle E. Messenger RNA turnover in eukaryotes: pathways and enzymes. Crit Rev Biochem Mol Biol 2004; 39 : 197–216. 3. Garneau NL, Wilusz J, Wilusz CJ. The highways and byways of mRNA decay. Nat Rev Mol Cell Biol 2007; 8 : 113–26. 4. Brown CE, Sachs AB. Poly(A) tail length control in Saccharomyces cerevisiae occurs by message-specific deadenylation. Mol Cell Biol 1998; 18 : 6548–59. 5. Yamashita A, Chang TC, Yamashita Y, et al. Concerted action of poly(A) nucleases and decapping enzyme in mammalian mRNA turnover. Nat Struct Mol Biol 2005; 12 : 1054–63. 6. Daugeron MC, Mauxion F, Seraphin B. The yeast POP2 gene encodes a nuclease involved in mRNA deadenylation. Nucleic Acids Res 2001; 29 : 2448–55. 7. Finoux AL, Seraphin B. In vivo targeting of the yeast Pop2 deadenylase subunit to reporter transcripts induces their rapid degradation and generates new decay intermediates. J Biol Chem 2006; 281 : 25940–7. 8. Tucker M, Staples RR, Valencia-Sanchez MA, et al. Ccr4p is the catalytic subunit of a Ccr4p/Pop2p/Notp mRNA deadenylase complex in Saccharomyces cerevisiae. EMBO J 2002; 21 : 1427–36. 9. Bianchin C, Mauxion F, Sentis S, et al. Conservation of the deadenylase activity of proteins of the Caf1 family in human. RNA 2005; 11 : 487–94. 10. Cougot N, van Dijk E, Babajko S, Seraphin B. Cap-tabolism. Trends Biochem Sci 2004; 29 : 436–44. 11. Simon E, Camier S, Seraphin B. New insights into the control of mRNA decapping. Trends Biochem Sci 2006; 31 : 241–3. 12. Mitchell P, Petfalski E, Shevchenko A, et al. The exosome: a conserved eukaryotic RNA processing complex containing multiple 3’->5’ exoribonucleases. Cell 1997; 91 : 457–66. 13. Buttner K, Wenig K, Hopfner KP. The exosome: a macromolecular cage for controlled RNA degradation. Mol Microbiol 2006; 61 : 1372–9. 14. Dziembowski A, Lorentzen E, Conti E, Seraphin B. A single subunit, Dis3, is essentially responsible for yeast exosome core activity. Nat Struct Mol Biol 2007; 14 : 15–22. 15. Liu H, Rodgers ND, Jiao X, Kiledjian M. The scavenger mRNA decapping enzyme DcpS is a member of the HIT family of pyrophosphatases. EMBO J 2002; 21 : 4699–708. 16. Van Dijk E, Le Hir H, Seraphin B. DcpS can act in the 5’-3’ mRNA decay pathway in addition to the 3’-5’ pathway. Proc Natl Acad Sci USA 2003; 100 : 12081–6. 17. Cougot N, Babajko S, Seraphin B. Cytoplasmic foci are sites of mRNA decay in human cells. J Cell Biol 2004; 165 : 31–40. 18. Eulalio A, Behm-Ansmant I, Izaurralde E. P bodies: at the crossroads of post-transcriptional pathways. Nat Rev Mol Cell Biol 2007; 8 : 9–22. 19. Parker R, Sheth U. P bodies and the control of mRNA translation and degradation. Mol Cell 2007; 25 : 635–46. 20. Olesen JR, Libri D, Jensen TH. A link between transcription and mRNP quality in Saccharomyces cerevisiae. RNA Biol 2005; 2 : 45–8. 21. Kadaba S, Krueger A, Trice T, et al. Nuclear surveillance and degradation of hypomodified initiator tRNAMet in S. cerevisiae. Genes Dev 2004; 18 : 1227–40. 22. Wyers F, Rougemaille M, Badis G, et al. Cryptic pol II transcripts are degraded by a nuclear quality control pathway involving a new poly(A) polymerase. Cell 2005; 121 : 725–37. 23. LaCava J, Houseley J, Saveanu C, et al. RNA degradation by the exosome is promoted by a nuclear polyadenylation complex. Cell 2005; 121 : 713–24. 24. Amrani N, Sachs MS, Jacobson A. Early nonsense: mRNA decay solves a translational problem. Nat Rev Mol Cell Biol 2006; 7 : 415–25. 25. Le Hir H, Moore MJ, Maquat LE. Pre-mRNA splicing alters mRNP composition: evidence for stable association of proteins at exon-exon junctions. Genes Dev 2000; 14 : 1098–108. 26. Vasudevan S, Peltz SW, Wilusz CJ. Non-stop decay: a new mRNA surveillance pathway. Bioessays 2002; 24 : 785–8. 27. Doma MK, Parker R. Endonucleolytic cleavage of eukaryotic mRNAs with stalls in translation elongation. Nature 2006; 440 : 561–4. 28. Brengues M, Teixeira D, Parker R. Movement of eukaryotic mRNAs between polysomes and cytoplasmic processing bodies. Science 2005; 310 : 486–9. 29. Cheadle C, Fan J, Cho-Chung YS, et al. Control of gene expression during T cell activation: alternate regulation of mRNA transcription and mRNA stability. BMC Genomics 2005; 6 : 75. 30. Wickens M, Bernstein DS, Kimble J, Parker R. A PUF family portrait: 3’UTR regulation as a way of life. Trends Genet 2002; 18 : 150–7. 31. Goldstrohm AC, Hook BA, Seay DJ, Wickens M. PUF proteins bind Pop2p to regulate messenger RNAs. Nat Struct Mol Biol 2006; 13 : 533–9. 32. Barreau C, Paillard L, Osborne HB. AU-rich elements and associated factors: are there unifying principles ? Nucleic Acids Res 2005; 33 : 7138–50. 33. Lykke-Andersen J, Wagner E. Recruitment and activation of mRNA decay enzymes by two ARE-mediated decay activation domains in the proteins TTP and BRF-1. Genes Dev 2005; 19 : 351–61. 34. Peng SS, Chen CY, Xu N, Shyu AB. RNA stabilization by the AU-rich element binding protein, HuR, an ELAV protein. EMBO J 1998; 17 : 3461–70. 35. Tang G. siRNA and miRNA: an insight into RISCs. Trends Biochem Sci 2005; 30 : 106–14. 36. Parker JS, Barford D. Argonaute: a scaffold for the function of short regulatory RNAs. Trends Biochem Sci 2006; 31 : 622–30. 37. Rana TM. Illuminating the silence: understanding the structure and function of small RNAs. Nat Rev Mol Cell Biol 2007; 8 : 23–36. 38. Samuel CE. Antiviral actions of interferons. Clin Microbiol Rev 2001; 14 : 778–809. 39. Harel-Bellan A. Prix Nobel de Médecine à Andrew Z. Fire et Craig c. Mello : silence, on désactive les gènes. Med Sci (Paris) 2006; 22 : 993–4. |