| |
| Med Sci (Paris). 2007 January; 23(1): 29–31. Published online 2007 January 15. doi: 10.1051/medsci/200723129.Dynamique de la chromatine lors de la réparation des lésions de l’ADN Sophie E. Polo1* and Geneviève Almouzni2* 1Wellcome Trust/Cancer Research UK Gurdon Institute, Cambridge, Royaume-Uni 2UMR 218 CNRS, Institut Curie, 26, rue d’Ulm, 75248 Paris Cedex 5, France MeSH keywords: Chromatine, Altération de l'ADN, Réparation de l'ADN, Réarrangement des gènes, Histone, Humains, Modèles génétiques |
L’ADN, support de l’information génétique, est aussi une macromolécule très réactive, cible de nombreux agents chimiques ou physiques susceptibles d’engendrer des lésions. La réparation fidèle de ces lésions par des mécanismes spécifiques garantit le maintien de l’intégrité du génome [
1]. Toutefois, dans le noyau des cellules eucaryotes, l’ADN est organisé en chromatine [
2], et c’est donc sur ce type de substrat qu’agissent les machineries de réparation de l’ADN. L’organisation en chromatine assure non seulement la compaction de l’ADN au sein du noyau cellulaire mais elle module également l’information génétique, contribuant ainsi à définir l’identité cellulaire. En effet, l’unité de base de la chromatine, baptisée nucléosome, comprend un octamère de protéines histones - un tétramère (H3-H4)2, flanqué par deux dimères H2A-H2B -, autour duquel s’enroule la double hélice d’ADN [
3]. Les protéines histones peuvent être modifiées (par acétylation, phosphorylation, méthylation…), ces modifications étant associées à différents états de la chromatine, plus ou moins permissifs pour l’expression des gènes [
4]. Par ailleurs, la nature même des histones, qui se déclinent sous différentes formes appelées « variants » [
5], contribue à la spécialisation des nucléosomes. Les modifications des histones et l’existence de variants fournissent ainsi un répertoire élargi d’information qualifiée d’« épigénétique ». Transmissible au cours des générations cellulaires et potentiellement réversible, cette information assure une régulation fine de l’expression du message contenu dans la séquence d’ADN sans impliquer de mutations [
6]. Au même titre que la stabilité du génome, on peut s’interroger sur l’importance du maintien de l’intégrité de l’information épigénétique lors d’une exposition à des agents génotoxiques. La réponse cellulaire aux dommages de l’ADN se conçoit non seulement au niveau de l’ADN lui-même mais aussi au niveau de son organisation en chromatine. |
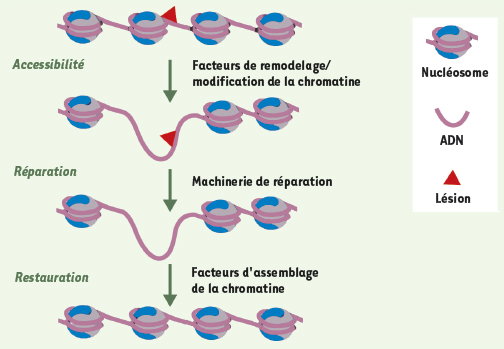
Réarrangements de la chromatine au cours de la réponse aux dommages de l’ADN Lorsque l’ADN est endommagé, les contraintes structurales imposées par son organisation en chromatine peuvent gêner la détection et la réparation des lésions. L’hypothèse qui prévaut actuellement pour rendre compte de la manière dont s’exerce la réparation des lésions de l’ADN au sein de la chromatine est le modèle access-repair-restore (accessibilité-réparation-restauration) [
7,
8] (Figure 1). Ce modèle propose que, lors d’un stress génotoxique, la chromatine se désorganise localement afin de faciliter l’accès de la machinerie de réparation aux lésions de l’ADN (access). Cette étape impliquerait des facteurs de remodelage de la chromatine et des enzymes de modification des histones. Après réparation de l’ADN (repair), l’organisation initiale de la chromatine devrait être restituée (restore). Cette dernière étape impliquerait des facteurs d’assemblage de la chromatine. Évidemment, selon la fidélité de la restitution de l’organisation initiale (nature des variants d’histones et leurs modifications), il sera possible ou non de préserver l’intégrité de l’information épigénétique pour un locus donné.  | Figure 1.
Dynamique de la chromatine lors de la réparation de l’ADN : modèle acces-repair-restore. La réparation des lésions de l’ADN au sein de la chromatine implique : (1) l’accès de la machinerie de réparation aux lésions de l’ADN ( access) ; (2) la réparation de ces lésions assurant la stabilité de l’information génétique ( repair) ; (3) la restitution de la structure initiale de la chromatine ( restore) (adapté de [ 8]). |
Le défi pour la réparation de lésions dans la chromatine est donc à concevoir à deux niveaux. Tout d’abord, concernant l’accessibilité des lésions de l’ADN au sein de la chromatine : à quel point la structure doit-elle être désorganisée pour permettre la réparation ? Ensuite, la question qui en découle concerne la restitution fidèle ou non de la structure de la chromatine après réparation de l’ADN. En particulier, on peut se demander à quel point l’information épigénétique portée par les histones est préservée. La dynamique des histones aux sites de dommages étant encore mal caractérisée, deux hypothèses peuvent être envisagées : (1) un mécanisme conservateur impliquant un simple glissement des octamères d’histones dans la région lésée, suivi de leur repositionnement ; ou (2) une éviction locale d’histones, qui peuvent ensuite être recyclées et/ou remplacées par de nouvelles histones. |
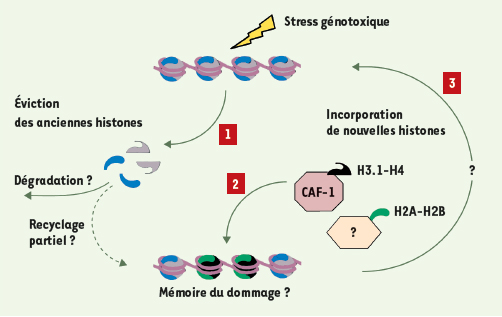
Dynamique locale des histones aux sites de dommages de l’ADN Afin de suivre la mise en place de nouvelles histones aux sites de dommages de l’ADN, une nouvelle approche expérimentale a été développée. Elle s’appuie sur la transfection transitoire de cellules humaines en culture avec un vecteur exprimant le variant d’histone H3.1 sous forme étiquetée (e-H3.1), afin de le distinguer de la forme endogène. Ce système, couplé à une irradiation UV localisée, permet de visualiser un recrutement local de nouvelles histones e-H3.1 aux sites de dommages [
9]. Cette incorporation constitue une réponse locale, restreinte à la région nucléaire irradiée. Elle est dépendante de l’activité réparatrice des lésions UV et fait directement intervenir le facteur d’assemblage de la chromatine CAF-1 (chromatin assembly factor-1) [9]. Ce facteur, connu pour sa capacité à promouvoir l’assemblage de la chromatine couplé à la synthèse d’ADN in vitro, aussi bien lors de la réplication que de la réparation [
10], est également associé de manière spécifique au variant d’histone H3.1 dans les cellules humaines [
11]. Il est intéressant de remarquer que la mise en place de nouvelles histones H3.1 intervient non seulement dans le contexte de dommages UV mais aussi en réponse à d’autres dommages, tels que ceux engendrés par micro-irradiation laser (principalement des cassures et des oxydations dans la molécule d’ADN) [9]. La restitution de l’organisation de la chromatine après réparation de l’ADN in vivo implique donc une incorporation localisée de nouvelles histones H3.1 aux sites de dommages (Figure 2).  | Figure 2.
Dynamique locale des histones en réponse au stress génotoxique. La mise en place de nouvelles histones aux sites de lésions de l’ADN (2) fait intervenir le facteur d’assemblage en chromatine CAF-1, qui dépose H3.1-H4, et potentiellement d’autres chaperons d’histones. Elle requiert l’éviction préalable des histones nucléosomiques pré-existantes (1), dont le devenir reste à déterminer. Celles-ci peuvent en effet être dégradées et/ou partiellement recyclées. Les facteurs impliqués dans leur éviction sont encore à identifier. L’incorporation de nouvelles histones, potentiellement différentes des histones pré-existantes, aux sites de dommages pose la question du maintien de l’intégrité épigénétique au sein de la chromatine lésée, qui nécessite de restituer à l’identique les anciennes marques épigénétiques (modifications et variants d’histones) (3). La présence prolongée de ces nouvelles histones (avec de nouvelles marques) pourrait cependant jouer un rôle dans la mémoire des dommages subis par la cellule. |
|
Incorporation de nouvelles histones aux sites de dommages : implications fonctionnelles Ces données ont des implications à plusieurs niveaux sur le plan fonctionnel. Tout d’abord, la mise en place de nouvelles histones H3 (vraisemblablement en association avec H4) implique une désorganisation totale du nucléosome, H3 et H4 occupant le centre de l’octamère d’histones. Une dynamique des histones H2A et H2B est donc également à envisager et il est probable que d’autres chaperons d’histones, impliqués à différents niveaux dans l’assemblage de la chromatine, participent à la dynamique de la chromatine endommagée (Figure 2). Par ailleurs, l’incorporation de nouvelles histones H3.1 n’exclut pas la mise en place d’autres variants de H3 par d’autres chaperons d’histones, ni le recyclage partiel des anciennes histones. Cependant, la restitution de l’organisation de la chromatine couplée à la réparation des lésions de l’ADN in vivo ne pouvant se réduire à un recyclage strict des histones pré-existantes, l’éviction, au moins partielle, sinon totale, de ces anciennes histones apparaît nécessaire (Figure 2). Des données récentes mettant en évidence une éviction locale d’histones au niveau d’une cassure double-brin de l’ADN chez la levure Saccharomyces cerevisiae viennent conforter cette hypothèse [
12]. Ce mécanisme s’ajouterait au remodelage de la chromatine et à la modification covalente des histones pour augmenter l’accessibilité de la chromatine endommagée. Il reste à évaluer dans quelles proportions se mélangent anciennes et nouvelles histones aux sites de réparation de l’ADN, et quels sont les facteurs déterminant leur importance relative. Si la mise en place de nouvelles histones aux sites de dommages participe à la restitution de l’organisation de la chromatine, son impact sur la stabilité épigénétique dans les régions de chromatine lésée doit être évalué. En effet, les histones nouvellement incorporées ne sont vraisemblablement pas en tous points identiques aux anciennes. On peut donc s’attendre à ce que des perturbations locales du profil de modification des histones surviennent, mais aussi à des changements dans la distribution des variants d’histones. Au-delà de son impact sur la stabilité épigénétique, l’incorporation locale de nouvelles histones peut aussi être envisagée comme un système de marquage de la chromatine, qui permettrait de garder en mémoire les dommages subis par la cellule. Ces nouvelles données montrent que la mise en place d’histones sur l’ADN permet de coupler, localement et temporellement, le maintien de l’intégrité du génome et de l’organisation de la chromatine en réponse au stress génotoxique. La restitution de l’organisation de la chromatine après réparation de l’ADN pourrait se révéler critique pour l’inactivation des mécanismes de signalisation des dommages (points de contrôle du cycle cellulaire), la stabilité chromosomique, ou encore la survie des cellules après exposition aux agents génotoxiques. Ces travaux ouvrent des perspectives intéressantes sur un nouveau mécanisme dont il faudra évaluer l’importance fonctionnelle à court et à long terme. Cette dynamique des histones est également à prendre en compte pour mieux comprendre l’impact de certains traitements endommageant l’ADN comme la radiothérapie ou la chimiothérapie. |
Ligue Nationale contre le Cancer (Équipe labellisée la Ligue), Commissariat à l’Énergie Atomique (CEA) (LRC n° 26), Contrat RTN (HPRN-CT-2002-00238), Programme Incitatif et Collaboratif « Paramètres Épigénétiques » entre l’Institut Curie et le CEA, Réseau d’Excellence Épigénome (LSHG-CT-2004-503433), ACI-DRAB (n° 04393) et Canceropôle. S. E. P. est financée par le Cancer Research UK.
|
1. Hoeijmakers JH. Genome maintenance mechanisms for preventing cancer. Nature 2001; 411 : 366–74. 2. Kornberg RD. Structure of chromatin. Annu Rev Biochem 1977; 46 : 931–54. 3. Luger K, Mader AW, Richmond RK, Sargent DF, Richmond TJ. Crystal structure of the nucleosome core particle at 2.8 Å resolution. Nature 1997; 389 : 251–60. 4. Peterson CL, Laniel MA. Histones and histone modifications. Curr Biol 2004; 14 : R546–51. 5. Kamakaka RT, Biggins S. Histone variants: deviants ? Genes Dev 2005; 19 : 295–310. 6. Russo VEA, Martienssen RA, Riggs AD. Epigenetic mechanisms of gene regulation. Plainview, NY : Cold Spring Harbor Laboratory Press, 1996. 7. Smerdon MJ. DNA repair and the role of chromatin structure. Curr Opin Cell Biol 1991; 3 : 422–8. 8. Green CM, Almouzni G. When repair meets chromatin. First in series on chromatin dynamics. EMBO Rep 2002; 3 : 28–33. 9. Polo SE, Roche D, Almouzni G. New histone incorporation marks sites of UV repair in human cells. Cell 2006; 127 : 481–93. 10. Mello JA, Almouzni G. The ins and outs of nucleosome assembly. Curr Opin Genet Dev 2001; 11 : 136–41. 11. Tagami H, Ray-Gallet D, Almouzni G, Nakatani Y. Histone h3.1 and h3.3 complexes mediate nucleosome assembly pathways dependent or independent of DNA synthesis. Cell 2004; 116 : 51–61. 12. Tsukuda T, Fleming AB, Nickoloff JA, Osley MA. Chromatin remodelling at a DNA double-strand break site in Saccharomyces cerevisiae. Nature 2005; 438 : 379–83. |