Nous poursuivons dans ce numéro la publication de la série d’articles émanant du GDR (rôle du calcium dans l’expression des gènes en conditions normales et pathologiques). Voir le début de cette série dans M/S n° 12, décembre 2006, page 1019.
Comme tous les organismes, les plantes intègrent en permanence les fluctuations de l’environnement, et modulent en conséquence leur croissance et leur développement. Parmi les systèmes de signalisation servant de relais entre la perception des stimulus et les réponses biologiques, le calcium participe, en tant que second messager, à la régulation de nombreux processus cellulaires, et au routage d’informations jusqu’au compartiment nucléaire où sont activés les programmes génétiques appropriés [ 1]. Les travaux menés sur les systèmes végétaux ont apporté des données originales sur la signalisation calcique nucléaire, et les résultats illustrant les multiples rôles du calcium et de la calmoduline sur l’expression génique, sont présentés dans cet article.
Présente chez tous les eucaryotes, la calmoduline (CaM) est une protéine capable de percevoir les variations du taux de calcium par des motifs de type EF-hand, qui ont une affinité élevée pour cet ion [ 2]. Le complexe Ca2+/CaM interagit et module l’activité de protéines cibles, qui présentent des structures et des fonctions variées. À côté d’une forme typique de CaM conservée chez tous les eucaryotes, les plantes possèdent un répertoire particulier de protéines apparentées à la CaM, dont les fonctions commencent à être décrites [ 3]. Différents travaux sur les CaM végétales et leurs cibles montrent l’importance de la signalisation calcique dans les processus associés à l’expression génique. En particulier, l’étude d’une forme atypique de CaM, présente dans le tabac, a révélé son intervention dans le contrôle du gene silencing [ 4]. Ce mécanisme de régulation de l’expression génique, initialement identifié chez les plantes, est un processus général chez les eucaryotes, qui agit par la dégradation ciblée d’ARN messagers. Des approches génétiques conduisant à moduler le niveau d’expression de cette CaM de tabac ont démontré son rôle essentiel dans la suppression du phénomène de gene silencing. D’autres travaux relatifs à la distribution sub-cellulaire des pools de CaM, indiquent une dynamique de répartition de la CaM entre le cytosol et le noyau. Par exemple, la CaM 53 du pétunia et la CaM 61 du riz, deux isoformes qui se distinguent de la CaM typique par la présence d’un motif de prénylation à leur extrémité carboxy-terminale, se trouvent ancrées au plasmalemme lorsque les plantes sont exposées à la lumière. En revanche, un passage à l’obscurité provoque leur accumulation dans le noyau, ce qui indique une mobilisation nucléaire de ces CaM lors d’un changement de régime lumineux [ 5]. La localisation différentielle de ces isoformes se traduit par des fonctions distinctes, car le phénotype de plantes transformées avec la CaM 53 est différent de celui observé chez les plantes exprimant la protéine altérée dans son site de prénylation et présente en permanence dans le noyau. Toutefois, les rôles respectifs de la CaM 53 au niveau du plasmalemme et du noyau restent à préciser.
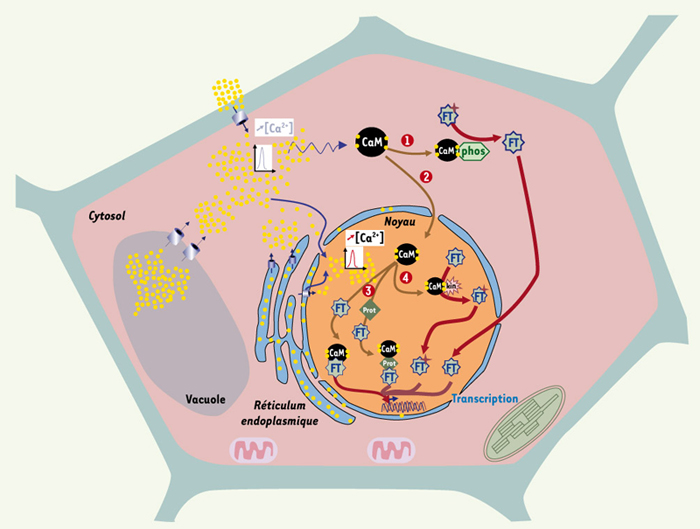
L’analyse des cibles des CaM végétales, dans laquelle notre équipe s’investit depuis quelques années, a aussi contribué à préciser les modes d’action de la signalisation calcique sur l’expression génique (Figure 1). Les CaM régulent l’activité de facteurs de transcription, et une étude récente illustre leur action sur un facteur MYB [ 6]. Deux isoformes de CaM de soja interagissent in vitro avec ce facteur et modulent de façon différentielle sa capacité de liaison à l’ADN ; la CaM 4, une isoforme induite par le stress salin, augmente cette activité alors que la CaM 1, une forme exprimée de manière constitutive, l’inhibe. L’expression ectopique de la CaM 4 dans des plantes transgéniques provoque une augmentation de la transcription de gènes de stress régulés par le facteur MYB, et conduit à une meilleure tolérance au stress salin. Au contraire, l’expression génique et la résistance au stress ne sont pas affectées dans des plantes transformées avec la CaM 1. La CaM intervient également de manière indirecte sur l’activité des facteurs de transcription. Nos travaux ont montré qu’une protéine nucléaire ne possédant ni domaine de liaison aux acides nucléiques ni activité catalytique, interagit avec la CaM [ 7]. Cette protéine qui se révèle être un régulateur négatif de la croissance lors d’une exposition des plantes à des conditions défavorables, contribue potentiellement à réguler l’expression génique par association à des facteurs de transcription. Par ailleurs, la modification des facteurs de transcription par des kinases et des phosphatases dont l’activité est régulée par la CaM, apparaît conservée chez les plantes (Figure 1). Des collègues du GDR 2688 ont récemment identifié, chez la luzerne, une kinase nucléaire dont l’activité est régulée par le calcium et la CaM [ 8]. Cette enzyme joue un rôle clé dans l’établissement d’une relation symbiotique entre la plante et des bactéries fixatrices d’azote du genre Rhizobium. Ses substrats potentiels sont des facteurs de transcription de type GRAS. Enfin, une phosphatase, cible de la CaM, dont la répartition nucléo-cytoplasmique varie selon les conditions physiologiques, pourrait assurer une fonction similaire à la calcineurine animale [ 9].
| Figure 1.
Représentation schématique des modes d’action de la calmoduline (CaM) sur la régulation de la transcription dans la cellule végétale. 1. Sous l’effet d’une élévation du taux de calcium dans le cytosol, la CaM active une phosphatase (phos) qui modifie un facteur de transcription (FT), ultérieurement transféré dans le noyau. 2. La CaM est importée dans le noyau en vue d’y activer des protéines nucléaires. 3. La CaM répond à un signal calcique nucléaire et interagit avec des facteurs de transcription ou d’autres protéines (prot) impliquées dans des complexes transcriptionnels. 4. La CaM active une kinase nucléaire (kin) qui modifie des facteurs de transcription. |
En conclusion, les travaux sur les CaM végétales ont contribué à révéler des liens étroits entre l’expression génique et la signalisation calcique par les rôles directs ou indirects de leurs cibles nucléaires sur la régulation de la transcription et d’événements post-transcriptionnels.