| |
| Med Sci (Paris). 2006 June; 22(6-7): 626–632. Published online 2006 June 15. doi: 10.1051/medsci/20062267626.Synergie entre les complexes de transcription et de maturation des ARN messagers Amélie Parent and Martin Bisaillon* Département de Biochimie, Faculté de Médecine, Université de Sherbrooke, 3001, 12e Avenue Nord, Sherbrooke, Québec, J1H 5N4 Canada |
Chez les eucaryotes, les ARN pré-messagers (ARN pré-m) transcrits par l’ARN polymérase II (ARN pol II) subissent plusieurs modifications co-transcriptionnelles avant d’être transportés sous forme mature (ARN messager [ARNm]) dans le cytoplasme pour y être traduits efficacement en différentes protéines. Chacune de ces modifications met en jeu de nombreux facteurs protéiques qui agissent au sein d’un vaste réseau d’interactions. On trouve, tout d’abord, l’ajout d’une structure coiffe à l’extrémité 5’ des ARNm, l’épissage des séquences non codantes (introns) et la synthèse d’une queue de polyadénosines (queue de poly[A]) à l’extrémité 3’. Bien que chacune de ces étapes puisse être étudiée de façon indépendante, les données actuelles démontrent clairement que les modifications auxquelles elles conduisent sont fortement interconnectées et qu’elles influencent les fonctions particulières et l’efficacité de chacune d’elles. Puisque ces processus de modification ont lieu de façon coordonnée avec la transcription, l’ARN polymérase II (ARN pol II) joue également un rôle clé dans la régulation de ces événements. |
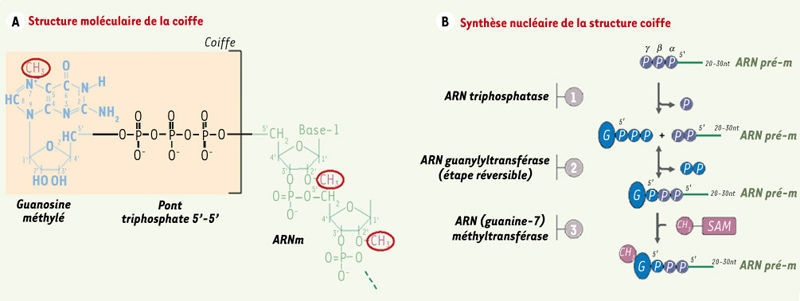
Synthèse de la structure coiffe La maturation des ARN pré-m est un événement qui joue un rôle critique dans l’expression des gènes eucaryotes. Elle débute tout d’abord par l’ajout d’une structure coiffe, m7GpppN-, à l’extrémité 5’ des acides ribonucléiques néosynthétisés. La coiffe est constituée d’un résidu guanosine méthylé en position N7 qui est relié au premier nucléotide retrouvé en 5’ de l’ARNm via une liaison 5’-5’ triphosphate (Figure 1A) [
1]. Mentionnons que chez les eucaryotes, les nucléotides adjacents à la coiffe ( m7GpppNpN-) peuvent également être méthylés à hauteur de la position 2 de leur groupement ribose. Ces structures sont alors désignées coiffe 0 ( m7GpppNpN-), coiffe 1 ( m7Gppp m2NpN-) ou coiffe 2 ( m7Gppp m2Np m2N-) selon le nombre de nucléotides méthylés [
2]. Bien que le rôle spécifique de ces méthylations sur les nucléotides adjacents à la coiffe demeure inconnu, certaines informations suggèrent que leur présence pourrait accroître la liaison des ribopolymères aux ribosomes [ 2].  | Figure 1.
Synthèse de la structure coiffe. A. Structure moléculaire de la coiffe. La structure coiffe est composée d’un résidu guanosine méthylé en position N7 et lié via un pont 5’-5’ triphosphate au nucléotide situé à l’extrémité 5’ de l’ARNm. Dans le cytoplasme de certains eucaryotes, un groupement méthyle peut être ajouté à la position 2 des riboses trouvés dans les nucléotides adjacents à la coiffe. B. Synthèse nucléaire de la structure coiffe. Le phosphate-γ trouvé à l’extrémité 5’ de l’ARN pré-m est initialement hydrolysé par l’ARN triphosphatase. Par la suite, l’ARN guanilyltransférase transforme une molécule de GTP en GMP pour ensuite la transférer à l’extrémité diphosphorylée de l’ARN pré-m. Il y a alors formation d’un pont 5’-5’ triphosphate. La synthèse de la coiffe se termine par la méthylation du résidu guanosine en position N7 par l’ARN (guanine-7) méthyltransférase via l’intermédiaire du composé S-adénosyl-L-méthyonine (SAM). |
La formation de la structure coiffe est catalysée par une succession de trois réactions enzymatiques qui ont lieu dans le noyau cellulaire. Celles-ci impliquent donc l’action d’une ARN triphosphatase (RTase), d’une ARN guanylyltransférase (GTase) et d’une ARN (guanine-7)-méthyltransférase (MTase) (Figure 1B) [
3,
4]. La coiffe est une structure fondamentale chez les eucaryotes où elle exerce un rôle critique dans le métabolisme de leur ARNm. On la trouve en action dans le prolongement du temps de demi vie des ARNm puisqu’elle fait office de barrière aux exonucléases qui agissent dans la direction 5’->3’ [4–
6]. L’excision de séquences introniques (processus d’épissage) est également facilitée par l’intermédiaire du complexe CBP20-CBP80 (cap binding protein 20 et 80) lié à la structure coiffe qui permet le recrutement du complexe d’épissage [
5]. Notons que la liaison de l’hétérodimère à la coiffe contribue également à créer un encombrement stérique important qui constitue un obstacle aux nucléases [4]. Il a été montré que ce même intermédiaire toujours lié à la coiffe participe au transport des ARN matures du noyau vers le cytoplasme pour assurer leur traduction [4–6]. Enfin, la structure coiffe est d’une importance cruciale pour la traduction des protéines. En effet, le facteur eIF-4E, un constituant important du complexe de traduction eIF-4F, interagit avec le groupement méthyle du résidu guanosine de la coiffe. Cette interaction joue un rôle critique dans le recrutement des ARNm aux ribosomes, favorisant de ce fait l’amorce de la formation du complexe de traduction et une synthèse protéique efficace [4, 6–
8]. Mécanisme d’épissage Les transcrits d’ARN nouvellement synthétisés présentent des séquences codantes (exons) qui sont interrompues par de nombreuses séquences non codantes (introns). Un processus général nommé épissage permet d’éliminer ces dernières par la réalisation de deux étapes de trans-estérification. Les exons d’une séquence codante peuvent ainsi se joindre entre eux pour donner naissance à un ARN à maturité qui pourra être traduit en une protéine dont la fonction sera précise (Figure 2A) [ 4,
9,
10]. Dans le cas de l’épissage alternatif, la sélection de différentes combinaisons d’exons (par l’exclusion de certains exons en plus des introns) à partir d’un même gène conduit à la formation de plusieurs profils d’ARNm bien spécifiques. Lors de la traduction, ces profils dicteront l’apparition de différents isoformes protéiques dont la fonction leur sera propre. Il s’agit donc d’un moyen de régulation génétique important chez les eucaryotes en plus de contribuer à leur diversité protéomique [ 9]. 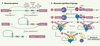 | Figure 2.
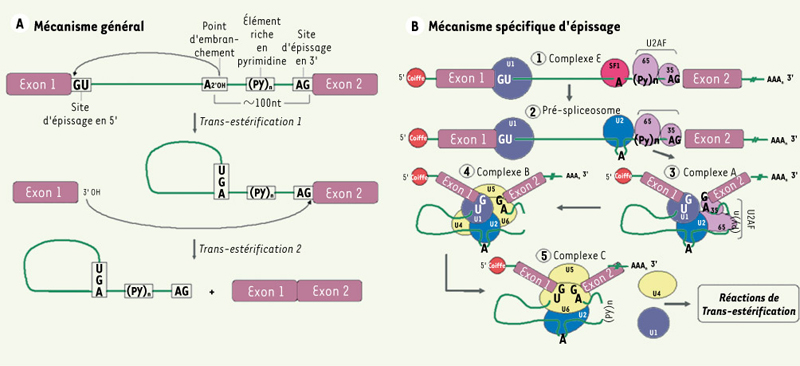
Épissage. A. Mécanisme général. 1. L’épissage est dirigé par des séquences consensus qui sont situées à la jonction exon/intron (site d’épissage en 5’) et à la jonction intron/exon (site d’épissage en 3’). Au site d’épissage en 5’, on trouve la séquence AG½GURAGU (barre verticale : site d’épissage; nucléotides soulignés : séquence invariable; R : purine). Un point d’embranchement constitué d’une séquence dont le résidu adénosine est hautement conservé : YNCURAY (Y : pyrimidine ; N : nucléotide aléatoire) et qui est suivi d’un élément riche en pyrimidine est présent environ 100 nucléotides avant le site d’épissage en 3’. Le site d’épissage en 3’ de l’intron est caractérisé par le motif YAG
½RNNN.2. Le clivage de séquences non codantes est un processus nucléaire réalisé par deux étapes de trans-estérifications. Ainsi, la liaison 3’-5’ phosphodiester du résidu guanosine présent au site d’épissage en 5’ subit une attaque nucléophile par le résidu adénosine hautement conservé trouvé au point d’embranchement. Il y a alors formation d’une interaction covalente entre ces deux nucléotides permettant à l’intron d’adopter la forme d’un lasso. L’extrémité 3’ libre de l’exon en amont de l’intron à épisser réalise la deuxième trans-estérification par l’attaque nucléophile de la liaison 3’-5’ phosphodiester du résidu guanosine au site d’épissage en 3’. Il y a alors détachement de l’intron déjà sous forme de lasso et liaison entre les deux exons adjacents.B. Mécanisme spécifique. Les deux étapes de trans-estérification sont rendues possibles grâce à un complexe catalytique macromoléculaire nommé spliceosome. Le spliceosome est constitué de cinq petites ribonucléoprotéines nucléaires snRNP (small nuclear ribonucleoprotein). Chacune d’elles est constituée d’un petit ARN nucléaire associé à plusieurs protéines pour former les snRNP U1, U2, U4, U5 et U6. De nombreuses protéines accessoires sont également présentes dans le spliceosome dont les protéines SF1 et le dimère U2AF. La formation du spliceosome sous forme active doit préalablement se réaliser afin de permettre l’excision intronique via les deux étapes de trans-estérification. Pour ce faire, un assemblage en cinq étapes est nécessaire. Il débute par la formation du complexe E où il y a appariement de U1 au site d’épissage en 5’, de SF1 au résidu adénosine très conservé à hauteur du point d’embranchement, de U2AF-65 à l’élément riche en pyrimidine et de U2AF-35 au site d’épissage en 3’. Par la suite, le pré-spliceosome est formé par la délocalisation de SF1 après la liaison de U2 au point d’embranchement. Cette association n’inclut toutefois pas le résidu adénosine afin qu’il soit disponible pour la première attaque nucléophile. Le complexe A apparaît grâce à la réorganisation du pré-spliceosome qui implique la formation d’interactions entre les facteurs U1, U2 et U2AF. Cela contribue au rapprochement des exons favorisant leur éventuelle liaison. L’arrivée du tri-snRNP U4/U5/U6 au sein des séquences consensus d’épissage entraîne la formation du quatrième complexe, le complexe B. Sa présence permet de libérer le point d’embranchement de même que le site d’épissage en 3’. Finalement, le complexe catalytique, ou complexe C, est formé par l’association de U6 et U2 au site d’épissage en 5’ déclenchant le départ de U1 et U4. Les étapes de trans-estérification peuvent alors avoir lieu. |
Ces processus sont rendus possibles grâce à un complexe macromoléculaire nommé spliceosome dont l’assemblage regroupe cinq sous-complexes. La création de ces sous-complexes fait appel à de petites ribonucléoprotéines nucléaires snRNP (U1, U2, U4, U5, U6) (small nuclear
ribonucleoprotein) de même que de nombreuses protéines accessoires telles que SF1, U2AF et les protéines de la famille SR (sérine/arginine-riches) (Figure 2B) [3, 4, 9]. Ainsi, les nombreuses réorganisations qui ont lieu entre ces facteurs entraînent l’apparition du complexe catalytique capable d’excision intronique et de ligation exonique. Mécanisme de polyadénylation La dernière étape de la maturation des ARN pré-m suppose une maturation de leur extrémité 3’. Pour ce faire, un clivage endonucléolytique se produit à cette extrémité, laissant ensuite la place à l’ajout d’une queue de poly(A) (Figure 3B) [ 4,
11,
12]. Trois séquences regroupées sous l’appellation de signal de polyadénylation dirigent l’apparition de la queue de poly(A) (Figure 3A) [ 4, 11,
13] ainsi qu’un minimum de six facteurs polymériques qui doivent être recrutés dans le noyau : CPSF, CstF, CFI et CFII, l’ARN polymérase II (ARN pol II) et la poly(A) polymérase (PAP) [ 4, 11, 13]. 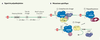 | Figure 3.
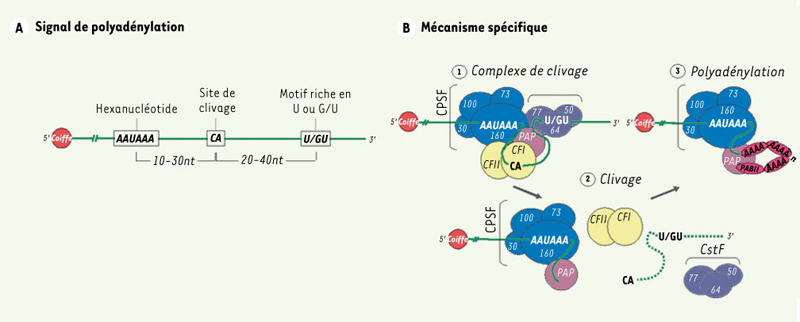
Polyadénylation. A. Signal de polyadénylation. La formation de la queue de polyadénosines est dirigée par le signal de polyadénylation composé de trois séquences. On retrouve ainsi un hexanucléotide hautement conservé, AAUAAA, situé à 10-30 bases du site de clivage, un motif riche en U ou en G/U (downstream element : DSE) positionné 20-40 bases en aval du site de clivage et le site de clivage, lui-même constitué du dinucléotide CA. La position exacte de ce dernier site est déterminée par la distance existant entre l’hexanucléotide et la séquence riche en U ou en G/U. L’étendue de la distance est également une indication de la longueur que peut atteindre la queue poly(A) pour chaque ARNm. Celle-ci varie normalement de 200 à 300 résidus adénosines. B. Mécanisme spécifique. 1. Afin de permettre une polyadénylation complète, un minimum de six facteurs polymériques doivent être recrutés dans le noyau. On retrouve tout d’abord le facteur protéique CPSF (cleavage/polyadenylation specific factor) qui participe à la fois à la réaction de clivage et à celle de polyadénylation. Il est constitué de quatre sous-unités nommées selon leur poids moléculaire respectif : CPSF-160, CPSF-100, CPSF-73 et CPSF-30. Le facteur CstF, constitué des sous-unités CstF-77, CstF-64 et CstF-50, est pour sa part engagé dans l’activité de clivage sans que sa présence ne soit obligatoire lors de la polyadénylation. Les facteurs CFI et CFII (cleavage factors I
and II) sont essentiels pour le processus de clivage puisqu’ils possèdent chacun une activité endonucléolitique. L’extrémité carboxyterminale (CTD) de l’ARN polymérase II (Pol II) fait également partie de la machinerie de polyadénylation. L’enzyme responsable de la synthèse de la queue poly(A), la poly(A) polymérase (PAP), est généralement requise pour obtenir un complexe de clivage actif. Notons que son activité de polymérisation est strictement exclusive à l’incorporation d’adénosines sur n’importe quel substrat d’ARN. Cependant, après le clivage du substrat, l’élongation de la queue poly(A) peut avoir lieu mais seulement en présence du complexe protéique CPSF. Pour augmenter la vitesse d’élongation tout en régulant la longueur de la queue poly(A), la protéine de liaison à la queue poly(A), PAB II (poly(A)-binding protein II), interagit avec ce segment d’adénosines. 2. Le processus de maturation nucléaire de l’extrémité 3’ de l’ARN pré-m débute par la formation du complexe de clivage. Pour ce faire, il y a liaison de CPSF et CstF à l’hexanucléotide et à la séquence riche en U ou en G/U. CFI et II sont ensuite recrutés au site de clivage. Le complexe est complété par la triple association de PAP à CPSF, CstF et CFI. La coupure de l’ARN entraîne la dégradation éventuelle du produit de clivage en 3’ par des exonucléases cellulaires de même que la libération des facteurs CstF, CFI et CFII. L’activité de polyadénylation de PAP débute alors très lentement à l’extrémité 3’ de l’ARN pré-m jusqu’à l’arrivée de PAB II. Ainsi, à chaque groupe de 10 résidus adénosines nouvellement synthétisés, PAB II s’y apparie, causant l’accélération de la processivité de la polymérase. Une fois la longueur maximale de la queue poly(A) atteinte, l’activité de PAP ralentit et s’arrête complètement avec la dissociation du complexe PAP-CPSF. |
Plusieurs rôles sont associés à la présence de ce segment d’adénosines. Entre autres, il participe au transport de l’ARNm du noyau vers le cytoplasme tout comme la structure coiffe. Il contribue également à stabiliser l’ARNm en procurant une protection de l’extrémité 3’ contre une dégradation précoce par des nucléases. Enfin, l’efficacité du démarrage de la traduction est également augmentée à la suite de l’adoption d’une conformation circulaire de l’ARNm par la présence de la queue poly(A) [11]. |
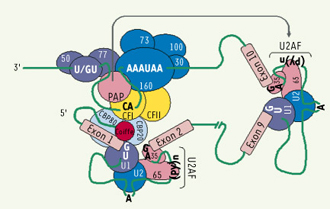
Interactions entre les processus de maturation de l’ARNm Certes, les processus de synthèse de la structure coiffe, d’épissage et de polyadénylation ont été découverts isolément, cependant des systèmes d’analyses in vitro ont mis en évidence une synergie entre ces différentes étapes de maturation [4]. La première se manifeste par l’interaction fonctionnelle entre la structure coiffe et l’épissage de l’intron qui lui est immédiatement adjacent. Cette interaction a lieu via le complexe protéique CBP20-CBP80 lié à la coiffe. L’hétérodimère interagit avec les snRNP U1 et U6 (Figure 2B) retrouvé au niveau de certains sous-complexes (complexe E et complexe C) du spliceosome, augmentant leur stabilité à l’ARN [4]. Ceci a pour conséquence d’accentuer la rapidité de formation de ces complexes et, du coup, d’accélérer le clivage de l’intron. Toutes ces interactions ont un impact majeur sur l’épissage de la totalité de l’ARN pré-m. En effet, il a été montré que l’absence du complexe CBP80 d’un extrait nucléaire cause une inhibition marquée de l’excision de tous les introns des ARN non matures. L’efficacité de l’épissage global est, de ce fait, fortement influencée par la présence de la coiffe [4] (Figure 4). 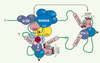 | Figure 4.
Interaction entre les processus de maturation de l’ARNm. Les facteurs protéiques engagés dans chacun des processus de maturation peuvent interagir entre eux. En effet, la coiffe régule l’assemblage progressif du spliceosome dans l’intron qui lui est immédiatement adjacent. Le complexe CBP20-CBP80 lié à la coiffe interagit avec U1, augmentant sa stabilité à l’ARN. Cela permet une formation plus rapide du complexe E. CBP20-CBP80 aurait également un impact sur la formation du complexe catalytique C puisqu’il interagit avec le facteur riboprotéique U6. Ce même complexe interagit aussi avec le facteur CFI du processus de polyadénylation, participant de ce fait à la stimulation du clivage de l’extrémité 3’ de l’ARN pré-m. La poly(A) polymérase contribue, pour sa part, à stimuler l’épissage de l’intron directement en amont du signal de polyadénylation en stabilisant U2AF-65. Les sites fonctionnels d’épissage de ce même intron peuvent également favoriser le processus de polyadénylation de l’extrémité 3’ de l’ARN pré-m. |
Toutefois, il existe encore peu de données susceptibles d’éclairer la relation entre la structure coiffe et le processus de maturation de l’extrémité 3’ de l’ARN pré-m. Malgré tout, on constate bien une association entre le complexe CBP20-CBP80 et certains facteurs participant au clivage endonucléolytique [
14]. Cette affinité a pour objectif premier d’augmenter l’efficacité du clivage par la stabilisation de CPSF et de CstF à l’ARN. On retrouve toutefois une influence positive sur l’élongation de la queue de poly(A) qui demeure modeste (Figure 4) [4, 14]. En effet, la délétion du complexe CBP20-CBP80 a montré une faible diminution au niveau de l’efficacité de l’élongation de la queue de poly(A) sans en affecter les complexes de polyadénylation [14]. Enfin, il faut noter que la protéine PAB I (poly(A)-binding protein I) associée à la queue de poly(A) peut interagir avec le polypeptide eIF4G, un composé important du complexe de traduction eIF4F qui lie la structure coiffe. Cette interaction joue un rôle primordial dans la circularisation des ARNm afin d’accroître leur stabilité en protégeant leurs extrémités contre les exonucléases cellulaires. De plus, cette circularisation permet de stimuler la traduction des ARNm en favorisant le recyclage rapide des ribosomes près du codon déclencheur de la traduction [
15]. Les facteurs participant à l’épissage et à la maturation de l’extrémité 3’ peuvent également avoir une influence positive l’un par rapport à l’autre. Dans un premier temps, le clivage d’un intron situé immédiatement en amont du signal de polyadénylation augmentera l’efficacité d’excision et d’ajout d’une queue de poly(A) en raison de l’apparition d’interactions entre le spliceosome et le complexe de polyadénylation. Celles-ci seront reflétées principalement par l’association entre les facteurs U1 snRNP et CPSF [
16]. Certaines observations laissent également croire que cette même ribonucléoprotéine U1 aurait une affinité pour des séquences du site de polyadénylation. De plus, notons que PAP peut contribuer à stimuler l’épissage de ce même intron en stabilisant le facteur accessoire (U2AF-65) associé au segment riche en pyrimidine du site d’épissage (Figure 4) [16]. |
Relation existant entre la transcription et la maturation de l’ARNm Domaine carboxyterminal de l’ARN polymérase II La synthèse des ARNm est réalisée uniquement par l’ARN pol II dont la grosse sous-unité (Rpb1) possède un domaine carboxyterminal (CTD) qui participe à la coordination des étapes nécessaires à la maturation des ARN pré-m [
17,
18]. Le CTD est composé de nombreuses répétitions de la séquence consensus YSPTSPS dont le nombre varie d’un organisme à l’autre (26 chez les levures et 52 chez les mammifères) [ 1, 4, 17]. Par ailleurs, la proportion de phosphorylation du CTD diffère durant les divers stades de la transcription. C’est ainsi que lors de la formation du complexe de pré-déclenchement et durant l’étape préliminaire de la transcription, le CTD est sous une forme hypophosphorylée. L’ARN pol II est alors nommée ARN pol IIA. Après la synthèse des 20 à 30 premiers nucléotides de l’ARN pré-m, l’activité de la polymérase ralentit pour faire place à la phosphorylation du CTD dans les sérines-2 et -5 de la séquence consensus Y SPT SPS. On est alors en présence de l’ARN pol IIO [ 1, 17–
19]. Transcription et structure coiffe La synthèse de la structure coiffe est un processus de maturation qui est directement couplé à la transcription. En effet, la GTase et la MTase sont toutes deux recrutées à l’ARN pol II en raison de leur capacité à interagir avec sa portion CTD hyperphosphorylée [ 1, 3, 17]. Cette association pour la GTase, qui a lieu avec les phosphosérines en position 5 de l’heptapeptide YSPTSPS, a pour effet d’activer l’enzyme à la suite de l’apparition d’un changement de conformation. Cela résulte — outre le fait de stimuler la formation de l’intermédiaire GTase-GMP — en une augmentation de l’affinité du substrat GTP pour la GTase [ 17]. Chez les levures, il a été montré que l’activité de la GTase est stimulée à la suite de la formation d’une interaction avec la Rtase. Cette stimulation permet à la Gtase de former efficacement le lien covalent enzyme-GMP en augmentant l’affinité de la protéine pour le substrat GTP [
20]. Chez les mammifères, les activités de la GTase et de la RTase sont présentes sur le même polypeptide. Le besoin de coordonner l’activité de la GTase via la RTase n’est donc pas nécessaire [ 1]. Une fois la synthèse de la coiffe terminée, il y a déphosphorylation partielle des sérines-5 entraînant le départ du complexe RTase-GTase du CTD. La MTase demeure, pour sa part, accrochée au CTD et migre avec la polymérase jusqu’à la formation de l’extrémité 3’ de l’ARN pré-m. Cela laisse supposer une participation de la protéine durant les processus de transcription et/ou de terminaison [ 3, 17, 20]. Des études génétiques effectuées chez la levure suggèrent que la protéine kinase kin28 est probablement requise pour le recrutement des enzymes participant à la synthèse de la structure coiffe au niveau de l’ARN pol II [
21]. Bien que d’autres kinases possèdent la capacité de phosphoryler le domaine CTD de l’ARN pol II in vitro, les études génétiques utilisant divers mutants thermosensibles suggèrent fortement que kin28 est utilisée de façon exclusive lors de la phosphorylation des sérines-5 du domaine CTD. Enfin, notons que d’autres évidences génétiques et physiques démontrent que la protéine hSPT5, impliquée dans l’élongation transcriptionnelle, interagit avec la GTase [
22]. Cette interaction a pour effet de stimuler spécifiquement l’activité GTase. Transcription et épissage Chez les métazoaires, certains gènes regroupent des séquences introniques qui sont éliminées par le processus d’épissage des transcrits d’ARN nouvellement synthétisés. Afin d’augmenter l’efficacité et la précision de ce processus, la présence du domaine CTD est d’une importance marquée. En effet, des expériences in vivo ont démontré que l’utilisation d’une polymérase tronquée au niveau du domaine CTD conduit à la synthèse d’un ARN pré-m dont l’épissage ne peut être réalisé [
23]. De plus, la forme phosphorylée de la polymérase semble également avoir un impact positif au niveau de l’efficacité de l’épissage puisqu’elle est responsable de l’initiation de la formation du spliceosome actif [ 23]. Toutes ces informations suggèrent que le CTD est un domaine crucial pour cibler efficacement les facteurs d’épissage au site de transcription [ 21]. Enfin, notons que des anticorps dirigés contre le CTD ont la capacité d’inhiber l’épissage in vitro. Des études récentes ont démontré qu’un groupe de protéines désignées SCAF (facteurs associés au domaine CTD) seraient sans doute responsables du lien unissant la transcription au processus d’épissage [
24]. Ces facteurs contiennent des régions très similaires aux domaines retrouvés dans les protéines SR présentes au niveau du spliceosome. De plus, il a récemment été démontré que les facteurs SCAF interagissent avec le domaine CTD de l’ARN pol II [
25]. Enfin, notons que la protéine SCAF8 semble interagir spécifiquement avec le domaine CTD phosphorylé. Des travaux de localisation démontrent clairement que la protéine se retrouve au site de transcription [
26]. Toutes ces données appuient le rôle des protéines SCAF dans l’établissement de liens entre la transcription et l’épissage. Une autre protéine, la protéine p52, pourrait également constituer un élément important permettant de relier l’épissage et la transcription [
27]. Cette protéine constitue un activateur puissant de la transcription. Cette capacité semble résulter de l’interaction entre la protéine et des activateurs de transcription ainsi qu’avec des facteurs généraux de la transcription. Des études récentes démontrent que la protéine p52 interagit également avec certaines protéines SR telles que ASF/SF2 et peut influencer l’efficacité de l’épissage dans un modèle in vitro [
28]. Ces propriétés suggèrent donc que la protéine p52 peut contribuer au lien entre la transcription et l’épissage. Transcription et polyadénylation Il y a plusieurs années, certaines analyses ont conduit à la prédiction de l’existence d’un lien entre les processus de transcription et de polyadénylation puisque des études ont montré que l’étape de terminaison de la transcription par l’ARN pol II était dépendante du signal de poly(A). Aujourd’hui, ces perspectives ont été confirmées grâce à différentes expériences utilisant des mutants de délétion de l’ARN pol II. Ainsi, ces travaux ont démontré que les ARNm synthétisés par une ARN pol II ne possédant pas de domaine CTD ne sont pas efficacement polyadénylés. L’ARN pol II est donc intimement couplée à la maturation de l’extrémité 3’ de l’ARN pré-m. De ce fait, le recrutement des facteurs de polyadénylation s’effectue au site de transcription via le domaine CTD. Ces facteurs se concentrent alors localement, rendant la polyadénylation plus efficace [ 3]. Le recrutement peut débuter très tôt lors de la transcription. En effet, trois des quatre sous-unités du complexe CPSF sont transférées à l’ARN pol II après l’amorce de la transcription et y demeurent associées tout au long de l’élongation [ 3]. De plus, lors de la formation du complexe de clivage, le CTD contribue à le rendre catalytiquement actif. Il faut toutefois mentionner que le niveau de phosphorylation du CTD n’a aucune influence sur l’activité du complexe [ 3]. L’existence d’un lien entre l’ARN pol II et la polyadénylation suggère fortement que l’étape de clivage endonucléolytique de l’extrémité 3’ des ARNm survient rapidement à la suite de la transcription du signal de polyadénylation. En fait, des travaux récents démontrent la présence de séquences G-riches situées près du site de polyadénylation et qui favorisent l’arrêt transitoire de l’ARN pol II [
29]. Cette pause pourrait ainsi permettre aux facteurs impliqués dans la polyadénylation d’agir rapidement avant que l’ARN pol II poursuive la transcription des séquences situées en aval du site de polyadénylation. |
Notre compréhension des détails moléculaires et mécanistiques de chacune des étapes de la synthèse et de la maturation de l’ARNpré-m à l’ARNm a grandement évolué grâce à l’étude approfondie de chacune de ces étapes. Bien que chaque activité puisse se dérouler indépendamment in vitro, elles sont appelées à se déployer de façon coordonnée in vivo. Le niveau de complexité de ces interactions demeure toutefois important. Pour cette raison, de nouveaux liens intermoléculaires seront sans doute mis en évidence dans un proche avenir. Des analyses thermodynamiques des diverses réactions permettraient, sans doute, d’accroître nos connaissances sur les événements de maturation. Mentionnons également que le domaine CTD de l’ARN pol II joue un rôle important dans plusieurs étapes de la transcription de même qu’au stade de la maturation post-transcriptionnelle (édition). Une caractérisation plus approfondie de son fonctionnement moléculaire devrait donc mener à une meilleure compréhension des mécanismes de régulation de la trancription et de la maturation des ARNm. |
Footnotes |
1. Bisaillon M. La structure-coiffe des ARN messagers. Med Sci (Paris) 2001; 17 : 312–9. 2. Furuichi Y, Shatkin AJ. Characterization of cap structures. Meth Enzymol 1989; 180 : 164–76. 3. Hirose Y, Manley JL. RNA polymerase II and the integration of nuclear events. Genes Dev 2000; 14 : 1415–29. 4. Proudfoot NJ, Furger A, Dye MJ. Integrating mRNA processing with transcription. Cell 2002; 108 : 501–12. 5. McKendrick L, Thompson E, Ferreira J, et al. Interaction of eukaryotic translation initiation factor 4G with the nuclear cap-binding complex provides a link between nuclear and cytoplasmic functions of the m(7) guanosine cap. Mol Cell Biol 2001; 21 : 3632–41. 6. Shatkin AJ. Capping of eucaryotic mRNAs. Cell 1976; 9 : 645–53. 7. Varani G. A cap for all occasions. Structure 1997; 5 : 855–8. 8. Baron-Benhamou J, Fortes P, Inada T, et al. The interaction of the cap-binding complex (CBC) with eIF4G is dispensable for translation in yeast. RNA 2003; 9 : 654–62. 9. Black DL. Mechanisms of alternative pre-messenger RNA splicing. Annu Rev Biochem 2003; 72 : 291–336. 10. Jurica MS, Moore MJ. Capturing splicing complexes to study structure and mechanism. Methods 2002; 28 : 336–45. 11. Colgan DF, Manley JL. Mechanism and regulation of mRNA polyadenylation. Genes Dev 1997; 11 : 2755–66. 12. Bienroth S, Keller W, Wahle E. Assembly of a processive messenger RNA polyadenylation complex. EMBO J 1993; 12 : 585–94. 13. Proudfoot N, O’Sullivan J. Polyadenylation: a tail of two complexes. Curr Biol 2002; 12 : R855–7. 14. Flaherty SM, Fortes P, Izaurralde E, et al. Participation of the nuclear cap binding complex in pre-mRNA 3’ processing. Proc Natl Acad Sci
USA 1997; 94 : 11893–8. 15. Gray NK, Jeffery MC, Dickson KS, Wickens M. Multiple portions of poly(A)-binding protein stimulate translation in vivo. EMBO J 2000; 19 : 4723–33. 16. Bauren G, Belikov S, Wieslander L. Transcriptional terminaison in the Balbiani ring 1 gene is closely coupled to 3’-end formation and excision of the 3’-termial intron. Genes Dev 1998; 12 : 2759–69. 17. Cramer P, Srebrow A, Kadener S, et al. Coordination between transcription and pre-mRNA processing. FEBS Lett 2001; 498 : 179–82. 18. Sims 3rd RJ, Belotserkovskaya R, Reinberg D. Elongation by RNA polymerase II : the short and long of it. Genes Dev 2004; 18 : 2437–68. 19. Sims 3rd RJ, Mandal SS, Reinberg D. Recent highlights of RNA-polymerase-II-mediated transcription. Curr Opin Cell Biol 2004; 16 : 263–71. 20. Howe KJ. RNA polymerase II conducts a symphony of pre-mRNA processing activities. Biochim Biophys Acta 2002; 1577 : 308–24. 21. Rodriguez CR, Cho EJ, Keogh MC, et al. Kin28, the TFIIH-associated carboxy-terminal domain kinase, facilitates the recruitment of mRNA processing machinery to RNA polymerase II. Mol Cell Biol 2000; 20 : 104–12. 22. Wen, Y, Shatkin AJ. Transcription elongation factor hSPT5 stimulates mRNA capping. Genes Dev 1999; 13 : 1774–9. 23. Hirose Y, Tacke R, Manley JL. Phosphorylated RNA polymerase II stimulates pre-mRNA splicing. Genes Dev 1999; 13 : 1234–9. 24. Yuriev A, Patturajan M, Litingtung Y, et al. The C-terminal domain of the largest subunit of RNA polymerase II interacts with a novel set of serine/arginine-rich protein. Proc Natl Acad Sci USA 1996; 93 : 6975–80. 25. Greenleaf AL. Positive patches and negative noodles : linking RNA processing to transcription. Trends Biochem 1993; 18 : 117–9. 26. Patturajan M, Wei X, Berezney R, Corden JL. A nuclear matrix protein interacts with the phosphorylated C-terminal domain of RNA polymerase II. Mol Cell Biol 1998; 18 : 2406–15. 27. Ge H, Si Y, Roeder RG. Isolation of cDNAs encoding novel transcription coactivators p52 and p75 reveals an alternate regulatory mechanism of transcriptional activation. EMBO J 1998; 17 : 6723–9. 28. Ge H, Si Y, Wolffe AP. A novel transcriptional coactivator, p52, functionnally interacts with the essential splicing factor ASF/SF2. Mol Cell 1998; 2 : 751–9. 29. Yonaha M, Proudfoot NJ. Specific transcriptional pausing activates polyadenylation in a coupled in vitro system. Mol Cell 1999; 3 : 593–600. |