| |
| Med Sci (Paris). 2006 May; 22(5): 519–524. Published online 2006 May 15. doi: 10.1051/medsci/2006225519.La régulation négative de la biogenèse des ribosomes Une nouvelle voie de contrôle du cycle cellulaire par le suppresseur de tumeurs Arf ? Olivier Ayrault, Lætitia Andrique, Christian-Jacques Larsen, and Paule Séité* EA 3805, Équipe d’Oncologie Moléculaire, Pôle Biologie-Santé, 40, avenue du recteur Pineau, 86022 Poitiers Cedex, France |
La protéine suppresseur de tumeurs p14ARF/p19arf contrôle négativement le cycle cellulaire par la voie désormais classique Arf*1-Mdm2-p53. Néanmoins, la protéine peut également inhiber la prolifération dans des modèles cellulaires déficients en p53, ce qui a conduit à postuler l’existence d’autres voies de régulation mettant en jeu de nouvelles cibles. De récents résultats montrent qu’Arf joue un rôle dans la biogenèse des ribosomes et exerce son contrôle aux niveaux transcriptionnel et post-transcriptionnel. En réponse à des signaux hyperprolifératifs, cette activité pourrait ainsi ralentir la prolifération, et associée à d’autres mécanismes, permettre l’arrêt du cycle cellulaire. |
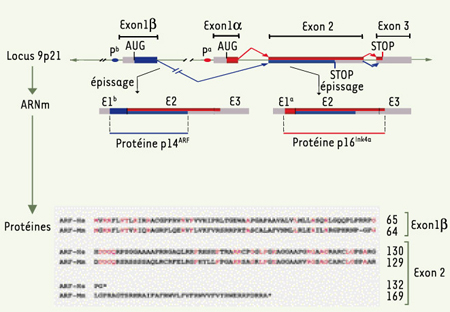
Protéine Arf et régulation négative de la prolifération Le locus Ink4a/Arf localisé sur le bras court du chromosome 9 (9p2.1) chez l’homme est fréquemment remanié dans les tumeurs solides (adénocarcinomes pancréatiques, mélanomes, cancers du poumon) et hémopathies malignes (leucémies aiguës lymphoblastiques). L’importance de son implication dans ces tumeurs s’explique en partie par son épissage atypique qui lui permet de générer à partir de deux exons communs (les exons 2 et 3) et deux exons alternatifs (les exons 1α et 1β) deux protéines entièrement différentes (Figure 1).
 | Figure 1.
Le locus INK4a/Arf : structure et expression. Le locus Ink4a/Arf sur la bande chromosomique 9p21 chez l’homme contient deux exons alternatifs, les exons 1 α et 1 β distants d’environ 15 kilobases. À partir du promoteur α (Pα), les exons 1α, 2 et 3 donnent naissance au transcrit α puis à la protéine suppresseur de tumeurs p16 Ink4a. Le transcrit β est obtenu à partir du promoteur β (Pβ) par un épissage alternatif entre les exons 1β, 2 et 3 et un décalage du cadre de lecture dans l’exon 2. Il donne naissance à la protéine suppresseur de tumeurs p14 ARF chez l’homme, p19 arf chez la souris. Les protéines humaine et murine sont des protéines nucléolaires, de respectivement 132 et 169 acides aminés, qui présentent une conservation de 61 % pour l’exon 1 et 44 % pour l’exon 2. Cette différence se retrouve sur certains aspects fonctionnels lors de la régulation du cycle cellulaire. Les résidus les plus conservés entre les espèces sont représentés en rose. ARF-Hs : Homo sapiens p14 ARF ; ARF-Mm : Mus musculus p19 arf (d’après [
27]). |
Le transcrit 1α-2-3 code la protéine p16Ink4a qui se lie aux complexes cycline D-CDK4/6, inhibe la phosphorylation de pRβ, et bloque la progression du cycle en G1/S. Le transcrit 1β-2-3 se démarque du précédent par la juxtaposition des exons 1β et 2 qui décale le cadre de lecture au début de l’exon 2 [
1]. Il donne naissance à une protéine Arf (alternative reading frame) mise en évidence initialement chez la souris (p19arf) [
2]. La protéine humaine p14ARF, plus courte, a été identifiée ultérieurement [
3] (Figure 1). Un premier mécanisme explicatif des propriétés inhibitrices d’Arf sur le cycle cellulaire implique la protéine p53. L’augmentation de l’expression d’Arf par des stimulus potentiellement oncogéniques (c-Myc, E2F-1, ras muté, E1A…), tout en activant l’expression de Mdm2 (HDM2 chez l’homme), inhibe l’activité E3-ubiquitine-ligase de cette dernière vis-à-vis de p53. La stabilisation de p53 permet alors l’arrêt de prolifération et l’induction de l’apoptose. Ces événements s’accompagnent parfois d’une séquestration nucléolaire de Mdm2 par Arf [
4] qui n’est toutefois pas nécessaire à l’arrêt du cycle [
5]. Quoi qu’il en soit, chez la souris, la région codée par l’exon1β semble nécessaire et suffisante pour assurer la localisation nucléolaire de p19arf, la liaison à Mdm2 et l’arrêt du cycle cellulaire. En revanche, pour la forme humaine de la protéine (p14ARF), la région codée par l’exon 2 contribue notamment à la localisation nucléolaire et à la liaison à HDM2. Un ensemble de résultats ne peut être expliqué par ce modèle : (1) la réintroduction de p19arf dans des fibroblastes embryonnaires murins où p53 Mdm2 et p19arf sont absentes stoppe la prolifération cellulaire [
6] ; (2) des souris p53 et p19arf nulles ont un spectre de tumeurs plus important que les animaux portant une seule délétion [
7] ; (3) chez l’homme, en l’absence de p53, la région codée par l’exon1β de p14ARF est nécessaire et suffisante pour se lier à E2F-1, l’un des acteurs de la voie Rb, et inhiber son activité transactivatrice nécessaire à la transition G1/S [
8]. Ces données suggèrent qu’Arf peut inhiber la prolifération par le biais d’autres voies que la voie Arf-Mdm2-p53. En particulier, plusieurs études récentes indiquent une intervention directe de la protéine dans le contrôle de la biogenèse des ribosomes. |
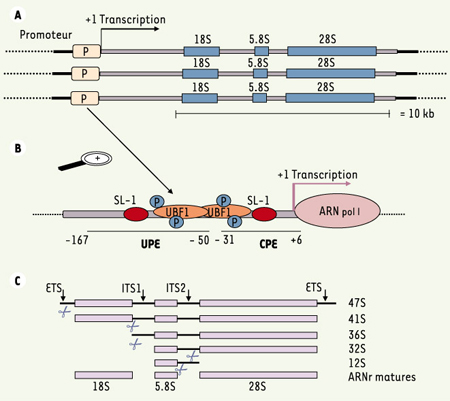
Bref rappel de la biogenèse des ribosomes La coordination entre la biosynthèse des ribosomes et la prolifération cellulaire est encore mal connue. Chez l’homme, les gènes de l’ADN ribosomique sont principalement disposés en clusters sur des régions chromosomiques regroupées dans l’ADN nucléolaire. Chaque unité transcriptionnelle est constituée des gènes correspondant aux ARN 18S, 28S et 5,8S et de séquences régulatrices qui sont transcrits par l’ARN polymérase I sous forme d’un ARNr précurseur 47S (Figure 2A et 2B). Schématiquement, après sa transcription, l’ARNr 47S subit deux modifications majeures : conversion des uridines en pseudo-uridine et méthylation de certaines séquences en 2’-O-méthyl nucléosides. Enfin, le précurseur subit une série de clivages par des ARNases (dont la nucléophosmine/B23 dont nous reparlerons plus loin) aboutissant aux formes matures des ARNr 5,8S, 28S et 18S [
9] (Figure 2C). Toutes ces étapes ont lieu au sein du nucléole tandis que l’ARNr 5S est transcrit par l’ARN polymérase III dans le nucléoplasme avant d’être importé dans le nucléole. Pour constituer les ribosomes, les ARNr matures s’associent séquentiellement avec plus de 70 protéines ribosomiques et vont former une particule pré-ribosomique 90S qui subira une série de clivages par des ARNases pour former la petite (40S) et la grande sous-unité (60S).
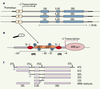 | Figure 2.
Synthèse des ARN ribosomiques (ARNr). A. Structure du locus ADNr. Les gènes des ARNr 18S, 5,8S et 28S sont répartis sur 250 à 300 unités transcriptionnelles regroupées au sein de l’ADN nucléolaire. Ils sont séparés par des régions qui seront transcrites (ETS et ITS) puis excisées lors des étapes de maturation. ETS : external transcribed sequence, ITS : internal transcribed sequence. B. Facteurs principaux de l’initiation de la transcription du pré-ARNr 47S. La mise en place du complexe transcriptionnel sur le promoteur est initiée par la phosphorylation du facteur UBF1. UBF1 phosphorylé recrute le complexe SL1, constitué d’une TATA binding protein (TBP) et de trois facteurs associés (TAF). L’ARN polymérase I peut ensuite se fixer sur le site d’initiation de la transcription. UPE : upstream promoter element ; CPE : core promoter element ; UBF1 : upstream binding factor 1 ; SL1 : selectivity factor 1. C. Étapes de maturation du pré-ARN47S en ARNr 18S, 5,8S et 28S. Le pré-ARN 47S subit une série de clivages séquentiels qui éliminent successivement les régions ETS et ITS. La nucléophosmine NPM/B23 intervient plus particulièrement dans la maturation de la forme 32S. |
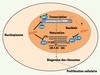 | Figure 3.
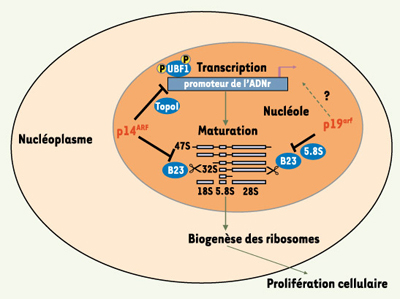
Arf, un régulateur négatif de la biogenèse des ribosomes. La protéine suppresseur de tumeurs Arf inhibe la prolifération cellulaire en l’absence de p53, en régulant négativement la biogenèse des ribosomes. Plusieurs arguments expérimentaux ont permis de mettre en évidence que la forme humaine de la protéine (p14ARF) intervient à deux niveaux : en inhibant la phosphorylation du facteur de transcription UBF1, elle diminue le taux de transcription du pré-ARNr 47S, et en entraînant la dégradation de l’endonucléase NPM/B23, elle ralentit la maturation de l’ARNr. L’homologue murin p19arf, en interagissant avec NPM/B23 et le petit ARNr 5,8S au sein de particules ribonucléoprotéiques, altère le clivage de l’ARNr 32S. Dans le modèle murin, il ne semble pas exister de contrôle au niveau transcriptionnel. |
Une coordination spatiale et temporelle et une régulation extrêmement précise permettent ainsi la synthèse d’environ 40 ribosomes par seconde dans la cellule proliférante. Cette régulation s’exerce à différents niveaux de la biogenèse des ribosomes comme nous allons le voir ci-après. Le promoteur des gènes des ARNr est constitué d’un domaine appelé CPE (core promoter element) qui contient le site d’initiation de la transcription et d’une région dite UPE pour upstream promoter element
(Figure 2B). Chez les eucaryotes, en dépit d’un faible pourcentage d’homologie au niveau des séquences primaires, il existe une conservation notable de la structure tridimensionnelle des promoteurs de l’ADNr. Toute modification ou altération de cette structure, nécessaire à la fixation des facteurs de transcription, conduit à une inhibition de ce mécanisme qui constitue déjà un premier niveau de contrôle. La transcription de l’ARNr 47S est dépendante de quatre facteurs principaux d’initiation de la transcription : TIF-1A, TIF-1B (SL1 chez l’homme), TIF-1C, et UBF [
10]. Dans les cellules quiescentes, le facteur UBF est hypophosphorylé et transcriptionnellement inactif. Pour permettre l’entrée des cellules en G1, UBF est phosphorylé et peut alors initier la formation du complexe de transcription [
11]. En se liant au promoteur, il recrute et stabilise le complexe protéique SL1 (TATA-binding protein et facteurs associés), active le recrutement de l’ARN polymérase I et inhibe les interactions protéines-ADN non spécifiques, notamment avec l’histone H1. L’arrêt de synthèse des ribosomes précédant l’entrée en mitose résulte de la phosphorylation de SL1 par le couple CDK1-cycline B, phosphorylation qui empêche la formation du complexe UBF/SL1 [
12]. L’inhibition de cette kinase pendant la mitose est capable de restaurer la synthèse du pré-ARNr mais pas sa maturation, indiquant que transcription et maturation répondent à deux mécanismes de régulation indépendants. La régulation de la synthèse est directement reliée aux exigences de la cellule en synthèse protéique, notamment pendant la phase de doublement de l’ADN en phase S [
13]. |
p14ARF inhibe la transcription du précurseur 47S des ARN ribosomiques Au laboratoire, nous avons mis en évidence une interaction physique et fonctionnelle entre p14ARF et la topo-isomérase I (Topo I), dont l’activité catalytique consiste à relaxer la chromatine, en particulier lors de la mise en place des complexes de transcription. L’interaction des deux protéines au sein du nucléole et la stimulation de l’activité catalytique de Topo I qui en découle in vitro sont deux fonctions dépendant de la région codée par l’exon 2 du locus Ink4a/Arf [
14,
15]. Le complexe p14ARF-Topo I est localisé dans le compartiment granulaire du nucléole, site d’accumulation des ARNr en cours de maturation et d’assemblage pour former des particules pré-ribosomiques. Parallèlement, la topo-isomérase I est indispensable à la transcription du pré-ARNr 47S, en permettant la relaxation de la région promotrice hautement condensée de l’ADNr. Par immunoprécipitation de chromatine, nous avons mis en évidence une interaction spécifique de p14ARF avec ce promoteur, en association avec Topo I [
16]. À l’aide de constructions utilisant un gène rapporteur luciférase, nous avons montré que la fixation de la protéine exogène p14ARF sur le promoteur de l’ADNr humain inhibe de 70 % l’activité de ce promoteur 48 heures après la transfection. Cette régulation transcriptionnelle est également observée in vivo où une diminution de 50 % du pré-ARNr 47S néosynthétisé est constatée sous l’effet de l’expression de p14ARF. Ces expériences ont été réalisées sur la lignée d’adénocarcinome broncho-alvéolaire H358, déficiente pour p53 et exprimant faiblement p14ARF [
17]. Puisque l’expression ectopique de p14ARF dans ces cellules entraîne un arrêt de prolifération visible après 48 heures, puis l’apoptose après 96 heures, ces résultats suggèrent, qu’en l’absence de p53 fonctionnelle, p14ARF pourrait agir comme un régulateur négatif de la transcription des ARNr, en relation avec l’arrêt du cycle. Toutefois, l’implication de Topo I dans ce processus reste inconnue à ce jour et la recherche d’un lien entre l’arrêt du cycle et l’activité d’endonucléase apoptotique de Topo I [
18] n’a pas abouti. En revanche, nous avons mis en évidence un complexe physique entre p14ARF et le facteur de transcription UBF et montré que sous l’effet de l’expression de p14ARF, UBF est hypophosphorylé. Plusieurs proto-oncogènes influencent la synthèse des ribosomes en régulant l’activité transcriptionnelle du facteur UBF par le biais de sa phosphorylation. Ainsi, la caséine kinase II surexprimée dans les leucémies et certaines tumeurs solides, peut phosphoryler spécifiquement UBF et induire de façon anarchique la synthèse des ribosomes [
19]. UBF est également la cible des complexes CDK4-cycline D1 et CDK4-cycline E [
20]. Des signaux extracellulaires tels que l’EGF sont capables d’activer la voie ERK1/2 et de phosphoryler UBF pour l’activer [
21]. Inversement, la phosphatase PP2A (tumour suppressor protein phosphatase 2), nécessaire à la déphosphorylation de UBF, est fréquemment inactivée dans les cancers, entraînant un déséquilibre de synthèse des ribosomes au cours de la progression normale du cycle cellulaire [
22]. À partir de ces données, nous proposons un modèle d’inhibition de la transcription du pré-ARNr 47S dans lequel p14ARF, en empêchant la phosphorylation d’UBF, altère la formation du complexe d’initiation de la transcription et donc la synthèse du précurseur ARNr 47S. À ce jour, aucune donnée concernant la protéine murine p19arf et son rôle dans le contrôle de la transcription des ARNr n’est connue. En revanche, les formes humaine (p14ARF) et murine (p19arf) semblent clairement impliquées dans le contrôle post-transcriptionnel et la maturation des ARNr. |
Arf inhibe la maturation du précurseur 47S des ARN ribosomiques Le groupe de C.J. Sherr a récemment montré, chez la souris, que p19arf était capable d’inhiber la maturation du pré-ARNr 47S dont les clivages ultérieurs induisent normalement la production des ARNr 28S, 18S et 5,8S [
23]. Cette équipe a observé une diminution de la maturation des ARNr 12S, 18S et du 28S dans un système cellulaire où p19arf est induit. Ce phénomène ne semble pas s’accompagner d’une diminution de la transcription de l’ARNr 47S lui-même. Le même groupe a pu montrer que l’effet de p19arf n’était pas directement lié à l’arrêt du cycle cellulaire. En effet, en mimant un arrêt du cycle cellulaire par une synchronisation des cellules ou par des agents bloquants, on continue d’observer une inhibition de la maturation des ARNr 18S et 28S par p19arf. Le phénomène semble donc bien dirigé contre la maturation des ribosomes et non la synthèse des ARNr eux-mêmes. De plus, l’inhibition de la maturation des ribosomes par p19arf requiert la partie aminoterminale de la protéine (résidus 1-14) correspondant à l’exon 1β et est totalement indépendante de la présence de Mdm2 ou de p53. D’une manière intéressante, cette inhibition est accompagnée d’une interaction entre p19arf et l’ARNr mature 5,8S. La séquence 1-14 de p19arf, qui paraît importante dans l’inhibition de la maturation des ARNr et l’arrêt du cycle cellulaire, est celle qui interagit avec l’ARNr 5,8S. Mais compte tenu de la longue demi-vie des ribosomes, l’arrêt prolifératif des cellules ne semble pas résulter d’un déficit en ribosomes. Ces résultats soulignent davantage un rôle de coordination entre prolifération cellulaire et production des ribosomes [23]. Une autre étude a permis d’isoler dans des fibroblastes de souris un complexe de très haut poids moléculaire (environ 5 MDa) qui contient p19arf et des protéines nucléolaires participant à la biogenèse des ribosomes, parmi lesquelles la nucléophosmine (NPM/B23) [
24]. L’une des fonctions biologiques de NPM/B23 est le clivage des ARNr immatures. Ainsi, in vitro, il a été montré que NPM/B23 clive le second ITS-2 (internal transcribed spacer-2) et permet la conversion du précurseur 32S en ARNr 28S et ARNr 5,8S (Figure 2). Ce clivage précède l’action d’autres exonucléases qui finalisent la maturation de l’ARNr 28S. La séquence aminoterminale 1-14 de p19arf est nécessaire à l’interaction avec NPM/B23 qui perd alors son activité de clivage au niveau de l’ITS-2. Par ailleurs, p19arf et NPM/B23 interagissent toutes les deux avec l’ARNr 5,8S. Ces données suggèrent qu’Arf interagit avec NPM/B23 dans les particules pré-ribosomiques, ce qui ralentit, voire inhibe la maturation des ARNr [24]. La protéine humaine p14ARF, par son extrémité aminoterminale, interagit également avec les 113 premiers résidus d’acides aminés de NPM/B23 [
25]. Dans plusieurs lignées cellulaires (U2OS, SaoOS-2, WI38) infectées par un adénovirus recombinant exprimant p14ARF, on observe une diminution significative de la quantité de NPM/B23, et ce dès 24 heures après l’infection. Cet effet semble spécifique puisqu’une autre protéine majeure du nucléole (C23) ne se lie pas à Arf. Cette diminution est corrélée à l’ubiquitinylation de la protéine NPM/B23 et sa destruction par le protéasome 26S. En revanche, ni HDM2, ni p53, n’affectent NPM/B23 dans ces mêmes cellules. Il est à noter que, contrairement au modèle murin, p14ARF n’interagit pas avec l’ARNr 5,8S [
26]. D’un point de vue biologique, l’implication de NPM/B23 dans la tumorigenèse et son mode d’action restent flous. Des mutations dans le gène codant NPM/B23 ont été décrites chez des patients atteints d’hémopathies malignes, et particulièrement des proliférations myéloïdes (leucémies aigües myéloblastiques LAM) [
28–
31]. Ces mutations conduisent à l’expression d’une protéine altérée dans la région carboxyterminale, ce qui engendre un signal d’export nucléaire (NES) responsable de la délocalisation cytoplasmique de la protéine mutante. L’équipe de C. Sherr a récemment montré [
32] que la co-transfection des formes sauvage NPM/B23 et mutante NPMc de la protéine dans des cellules NIH-3T3 a pour effet de délocaliser également la protéine sauvage, ainsi que p19arf à laquelle elle est associée dans le nucléole au sein de complexes multimoléculaires de grande taille [24]. Si la surexpression de NPM n’altère pas les fonctions dépendantes de p53 de la protéine Arf, la surexpression de NPMc, en délocalisant p19arf dans le cytoplasme, les atténue. En revanche, la régulation négative de la biogenèse des ribosomes, fonction indépendante de p53, n’est pas altérée par NPMc, alors que la sumoylation de NPM est inhibée [
33]. S’il est tentant de spéculer que ces hémopathies sont engendrées par la seule perturbation des fonctions de Arf sous l’effet de l’expression de NPMc, il faut également prendre en compte les conséquences de l’expression de la protéine mutante sur la protéine NPM endogène, possible gène suppresseur de tumeurs, dont les fonctions sont également altérées. |
Différents arguments expérimentaux indiquent que la protéine suppresseur de tumeurs Arf exerce une fonction régulatrice sur la prolifération cellulaire via un contrôle de la biogenèse des ribosomes. En l’état actuel de nos connaissances, ce contrôle s’effectue essentiellement sur la composante ARN de ces complexes ribonucléoprotéiques et ceci à deux niveaux : (1) transcriptionnel, nous avons montré que p14ARF inhibe fortement la synthèse du pré-ARNr 47S ; (2) post-transcriptionnel, p14ARF et p19arf ralentissent la maturation du précurseur 32S en ARNr 28S et 5,8S. L’existence d’une régulation à deux niveaux peut s’expliquer dans la mesure où il apparaît clairement qu’aucun des deux mécanismes de régulation n’est, à lui seul, capable d’inhiber complètement la production d’ARNr. De plus, la demi-vie des ribosomes dans la cellule est telle que la diminution de la transcription ou le ralentissement de la maturation des ARNr ne suffisent pas à induire l’arrêt du cycle. Il est probable, en revanche, que la mise en jeu consécutive de ces deux mécanismes puisse accroître l’efficacité globale de l’arrêt de prolifération. Cette nouvelle voie de contrôle a initialement été mise en évidence dans des modèles cellulaires chez lesquels la voie de régulation classique Arf-Mdm2-p53 n’est pas fonctionnelle, suggérant qu’il pourrait s’agir d’une voie « de secours » en réponse à des signaux hyperprolifératifs. Toutefois, Itahana et al. ont montré que la protéine humaine p14ARF était également capable de ralentir la maturation des ARNr dans des cellules exprimant p53, ce qui oriente vers un mécanisme généralisé. Il est important de noter, qu’à l’exception de la voie Arf-Mdm2-p53 qui fonctionne de manière identique chez la souris et l’homme, des divergences existent par ailleurs entre les deux protéines : l’inhibition de la transcription du pré-ARN 47S n’est pas observée dans les modèles murins dépourvus de p53 et le blocage de la maturation n’est pas démontré dans les cellules de souris exprimant une p53 fonctionnelle. Sur ce point, nombre d’arguments existent aujourd’hui qui mettent en évidence que ces deux protéines, qui partagent seulement 50 % d’homologie, peuvent utiliser des voies différentes et des partenaires spécifiques pour assurer leur fonction suppresseur de tumeurs. |
Footnotes |
1. Duro D, Bernard O, Della Valle V, et al. A new type of p16INK4/MTS1 gene transcript expressed in B-cell malignancies. Oncogene 1995; 11 : 21–9. 2. Quelle DE, Zindy F, Ashmun RA, Sherr CJ. Alternative reading frames of the INK4a tumor suppressor gene encode two unrelated proteins capable of inducing cell cycle arrest. Cell 1995; 83 : 993–1000. 3. Della Valle V, Duro D, Bernard O, Larsen CJ. The human protein p19ARF is not detected in hemopoietic human cell lines that abundantly express the alternative beta transcript of the p16INK4a/MTS1 gene. Oncogene 1997; 15 : 2475–81. 4. Pomerantz J, Schreiber-Agus N, Liegeois NJ, et al. The Ink4a tumor suppressor gene product, p19Arf, interacts with MDM2 and neutralizes MDM2’s inhibition of p53. Cell 1998; 92 : 713–23. 5. Llanos S, Clark PA, Rowe J, Peters G. Stabilization of p53 by p14ARF without relocation of MDM2 to the nucleolus. Nat Cell Biol 2001; 3 : 445–52. 6. Carnero A, Hudson JD, Price CM, Beach DH. p16INK4A and p19ARF act in overlapping pathways in cellular immortalization. Nat Cell Biol 2000; 2 : 148–55. 7. Weber JD, Jeffers JR, Rehg JE, et al. p53-independent functions of the p19ARF tumor suppressor gene. Genes Dev 2000; 14 : 2358–65. 8. Eymin B, Karayan L, Seite P, et al. Human ARF binds E2F1 and inhibits its transcriptional activity. Oncogene 2001; 20 : 1033–41. 9. Hannan KM, Hannan RD, Rothblum LI. Transcription by polymerase I. Front Biosci 1998; 3 : 376–98 10. Russell J, Zomerdijk JC. RNA-polymerase-I-directed rDNA transcription, life and works. Trends Biochem Sci 2005; 30 : 87–96. 11. Voit R, Kuhn A, Sander EE, Grummt I. Activation of mammalian ribosomal gene transcription requires phosphorylation of the nucleolar transcription factor UBF. Nucleic Acids Res 1995; 23 : 2593–9. 12. Grummt I. Regulation of mammalian ribosomal gene transcription by RNA polymerase I. Prog Nucleic Acid Res Mol Biol 1999; 62 : 109–54. 13. Moss T, Stefanovsky VY. At the center of eukaryotic life. Cell 2002; 109 : 545–8. 14. Karayan L, Riou JF, Seite P, et al. Human ARF protein interacts with topoisomerase I and stimulates its activity. Oncogene 2001; 20 : 836–48. 15. Ayrault O, Karayan L, Riou JF, et al. Delineation of the domains required for physical and functional interaction of p14ARF with human topoisomerase I. Oncogene 2003; 22 : 1945–54. 16. Ayrault O, Andrique L, Larsen CJ, Seite P. Human Arf tumor suppressor specifically interacts with chromatin containing the promoter of rRNA genes. Oncogene 2004; 23 : 8097–104. 17. Eymin B, Leduc C, Coll JL, et al. p14ARF induces G2 arrest and apoptosis independently of p53 leading to regression of tumours established in nude mice. Oncogene 2003; 22 : 1822–35. 18. Pourquier P, Kohlhagen G, Urasaki Y. Induction of topoisomerase I cleavage complexes by staurosporine in apoptotic leukaemia cells : could topoisomerase I act as an apoptotic endonuclease ? Proc Am Assoc Cancer Res 2001; 42 : 303. 19. Voit R, Schnapp A, Kuhn A, et al. The nucleolar transcription factor mUBF is phosphorylated by casein kinase II in the C terminal hyperacidic tail which is essential for transactivation. EMBO J 1992; 11 : 2211–8. 20. Voit R, Hoffmann M, Grummt I. Phosphorylation by G1-specific cdk-cyclin complexes activates the nucleolar transcription factor UBF. EMBO J 1999; 18 : 1891–9. 21. Stefanovsky VY, Pelletier G, Hannan R, et al. An immediate response of ribosomal transcription to growth factor stimulation in mammals is mediated by ERK phosphorylation of UBF. Mol Cell 2001; 8 : 1063–73. 22. Klein J, Grummt I. Cell cycle-dependent regulation of RNA polymerase I transcription : the nucleolar transcription factor UBF is inactive in mitosis and early G1. Proc Natl Acad Sci USA 1999; 96 : 6096–101. 23. Sugimoto M, Kuo ML, Roussel MF, Sherr CJ. Nucleolar Arf tumor suppressor inhibits ribosomal RNA processing. Mol Cell 2003; 11 : 415–24. 24. Bertwistle D, Sugimoto M, Sherr CJ. Physical and functional interactions of the Arf tumor suppressor protein with nucleophosmin/B23. Mol Cell Biol 2004; 24 : 985–96. 25. Itahana K, Bhat KP, Jin A, et al. Tumor suppressor ARF degrades B23, a nucleolar protein involved in ribosome biogenesis and cell proliferation. Mol Cell 2003; 12 : 1151–64. 26. Andrique L, Ayrault O, Larsen CJ, Séité P. In vitro and in vivo analysis of the interaction between 5,8S rRNA and ARF protein reveal a new difference between murine p19ARF and human p14ARF. Oncogene 2005; 24 : 2580–4. 27. Zhang Y, Xiong Y. Control of p53 ubiquitination and nuclear export by MDM2 and ARF. Cell Growth Differ 2001; 12 : 175–86. 28. Piazza F, Gurrieri C, Pandolfi PP. The theory of APL. Oncogene 2001; 20 : 7216–22. 29. Duyster J, Bai RY, Morris SW. Translocations involving anaplastic lymphoma kinase (ALK). Oncogene 2001; 20 : 5623–37. 30. Falini B, mecucci C, Tiacci E, et al. Cytoplasmic nucleophosmin in acute myelogenous leukemia with a normal karyotype. N Engl J Med 2005; 352 : 254–66. 31. Grisendi S, Pandolfi PP. NPM mutations in acute myelogenous leukemia. N Engl J Med 2005; 352 : 291–2. 32. Den Besten V, Kuo ML, Williams RT, Sherr CJ. Myeloid leukaemia-associated nucleophosmin mutants perturb p53-dependent and independent activities of the Arf tumor suppressor protein. Cell Cycle 2005; 4 : 1593–8. 33. Tago K, Chiocca S, Sherr CJ. Sumoylation induced by the Arf tumor suppressor : a p53-independent function. Proc Natl Acad Sci USA 2005; 102 : 7689–94. |