| |
| Med Sci (Paris). 2006 May; 22(5): 507–513. Published online 2006 May 15. doi: 10.1051/medsci/2006225507.eIF4E et étapes décisionnelles du développement embryonnaire Quand la traduction module le développement Nathalie Oulhen and Patrick Cormier* Station biologique de Roscoff, Cycle cellulaire et développement, Unité Mer et Santé (UMR 7150), Université Pierre-et-Marie Curie (EI 37), Centre national de la recherché scientifique (CNRS), Institut national des sciences de l’univers (INSU), BP 74, 29682 Roscoff Cedex, France |
La synthèse protéique, qui suit et affine le contrôle exercé lors de la transcription, représente une étape importante de l’expression des gènes [
1]. Un contrôle efficace de la traduction des ARNm aboutit à une modulation rapide des quantités de protéines dans un contexte temporel et spatial, à l’échelle de la cellule et des tissus. La traduction se déroule classiquement en trois phases : l’initiation, l’élongation et la terminaison. Bien que des contrôles puissent s’appliquer à chacune de ces trois phases, l’initiation de la synthèse protéique est une phase régulatrice primordiale de la synthèse protéique [
2]. Parmi la machinerie complexe de la traduction, le facteur eIF4E est une cible majeure de la régulation de l’initiation [
3], en réponse à différents stimulus. Ce facteur d’initiation joue également un rôle crucial dans le contrôle du cycle cellulaire [
4,
5], et a été impliqué dans plusieurs mécanismes de l’embryogenèse [
6–
9]. Après avoir situé le facteur eIF4E dans les étapes précoces de l’initiation de la traduction, nous verrons comment cet acteur moléculaire intervient dans le contrôle général ou spécifique de la synthèse des protéines, au cours de la gamétogenèse, de la fécondation et du développement embryonnaire de différents organismes modèles (drosophile, nématode, oursin et xénope). |
Contrôle de la traduction Chez les organismes eucaryotes, l’extrémité 5’ non traduite (5’UTR1) des ARNm est coiffée par une molécule de m7GTP (où « m » est le groupe méthyle de la guanosine positionnée sur le N7). Cette coiffe facilite le transport des ARNm du noyau vers le cytoplasme, stabilise les ARNm [
10] et, surtout, est essentielle pour la traduction « dépendante de la coiffe » (cap-dependent) [
11]. L’interaction de cette coiffe m7GTP avec les facteurs d’initiation permet de recruter en 5’UTR des ARNm le complexe 43S, composé de la sous-unité 40S du ribosome et du complexe ternaire (eIF2/GTP/ARNt-méthionine) initiateur. L’étape d’initiation fait intervenir au moins onze facteurs protéiques [4], dont eIF4E, eIF4G et eIF4A, qui forment le complexe eIF4F, une cible clé du contrôle de l’initiation de la traduction. Le facteur eIF4E est une protéine très conservée chez les eucaryotes, qui s’associe à la coiffe m7GTP des ARNm. Pour stimuler la synthèse protéique, eIF4E s’associe à eIF4G, une protéine de haut poids moléculaire jouant le rôle de plateforme pour recruter des partenaires tels qu’eIF4A, une ARN hélicase, et eIF3, qui apporte le complexe 43S sur l’ARNm (Figure 1). Le facteur eIF4G recrute PABP, une protéine qui, liée à la queue poly (A) des messagers, relie ainsi les extrémités 3’ et 5’UTR et stabilise les complexes d’initiation [
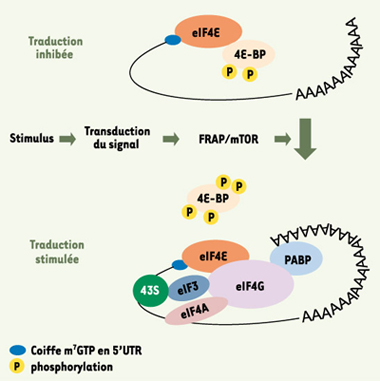
12]. Ce modèle en boucle permet d’expliquer la corrélation existant entre la poly-adénylation et l’efficacité du recrutement des ARNm. 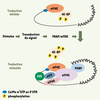 | Figure 1.
Contrôle de l’initiation de la traduction dépendante de la coiffe dans les cellules de mammifères. Lorsqu’eIF4E est lié à la protéine 4E-BP hypophosphorylée, la synthèse protéique est inhibée. L’addition d’un stimulus (facteurs de croissance ou insuline) aux cellules de mammifères déclenche une cascade de phosphorylations impliquant la phospho-inositide (PI) 3-kinase, la protéine kinase B/Akt et la kinase sensible à la rapamycine FRAP/mTOR, aboutissant à l’hyperphosphorylation de 4E-BP et à sa dissociation d’eIF4E. Celui-ci est alors libre de recruter eIF4G et de stimuler la synthèse protéique. La protéine de pontage eIF4G s’associe à eIF4A, une ARN hélicase, ainsi qu’à eIF3, responsable du recrutement du complexe 43S en 5’UTR de l’ARNm. Le facteur eIF4G est également capable de recruter la protéine PABP, elle-même associée à la queue poly (A) des ARNm, formant ainsi le lien entre les régions 5’- et 3’UTR pour stimuler la traduction. |
Bien que majoritaire, la traduction des ARNm dépendante de la coiffe n’est pas l’unique mécanisme de recrutement des ARNm dans les polysomes : certains le sont par un mécanisme alternatif, qui permet l’entrée directe du ribosome en 5’UTR [
13]. Ce mécanisme est fondé sur la présence de séquences IRES sur les ARNm [
14], séquences qui privilégient leur traduction dans des conditions physiologiques particulières de la cellule, notamment en mitose [
15]. De même, au cours du développement embryonnaire de la drosophile, la traduction des ARNm antennapedia et ultrabithorax est hautement contrôlée par des séquences IRES [
16]. Au cours de la régulation de la traduction dépendante de la coiffe, la disponibilité d’eIF4E pour son partenaire eIF4G est un point essentiel. La petite protéine 4E-BP inhibe de manière compétitive l’association entre eIF4E et eIF4G ; chez les mammifères, les trois protéines 4E-BP1, -2 et -3 se lient à eIF4E et sont capables d’inhiber la traduction. En revanche, un seul orthologue a, pour l’instant, été décrit chez les invertébrés comme la drosophile [
17] ou l’oursin [
8]. Dans les cellules de mammifères en culture, le niveau de phosphorylation des 4E-BP contrôle leur association à eIF4E : le facteur 4E-BP1 hypophosphorylé interagit avec eIF4E, tandis que son hyperphosphorylation provoque sa dissociation d’eIF4E. En réponse à un stimulus hormonal du type insuline ou facteurs de croissance, la phosphorylation de 4E-BP1 est en aval d’une cascade de phosphorylation impliquant la phospho-inositide (PI) 3-kinase, la protéine kinase B/Akt et la kinase sensible à la rapamycine FRAP/mTOR (Figure 1). L’état de phosphorylation de 4E-BP contrôle finement son association avec eIF4E dans les cellules somatiques de mammifères. Bien que majoritaire dans l’ensemble des systèmes étudiés, ce mécanisme post-traductionnel n’est cependant pas le seul à intervenir dans le contrôle du complexe eIF4E/4E-BP : ainsi, l’analyse des régulations traductionnelles en réponse à la fécondation chez l’oursin a permis d’identifier un mécanisme annexe et efficace pour libérer eIF4E de son répresseur. |
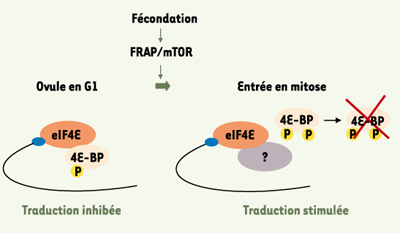
Fécondation chez l’oursin Chez l’oursin, la synthèse protéique qui accompagne les mécanismes d’activation induits par la fécondation est indispensable à l’entrée dans le cycle cellulaire et à la poursuite du développement embryonnaire. L’augmentation de la traduction est indépendante de la transcription, et le contrôle traductionnel s’exerce principalement lors de l’initiation (Figure 2). 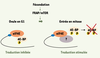 | Figure 2.
Dégradation de 4E-BP en réponse à la fécondation chez l’oursin. Chez l’oursin, avant la fécondation, eIF4E est lié à 4E-BP. La fusion des gamètes active la kinase FRAP/mTOR : le répresseur 4E-BP est alors rapidement phosphorylé et dégradé. Après fécondation, eIF4E est disponible pour des partenaires, encore non identifiés, qui permettront de stimuler la synthèse protéique dépendante de la coiffe des ARNm. |
La protéine eIF4E et l’inhibiteur 4E-BP sont présents dans les ovules d’oursin [8]. Après la fécondation, 4E-BP, rapidement dissocié d’eIF4E, est dégradé [
18] : cette dégradation, qui représente un nouveau mécanisme de régulation de la traduction identifié pour la première fois chez l’oursin, pourrait être impliqué dans des mécanismes cellulaires tels que l’apoptose [
19] et la prolifération [
20] des cellules de mammifères. La dégradation de 4E-BP induite par la fécondation est sensible à l’inhibiteur de la kinase FRAP/mTOR, la rapamycine, qui inhibe la première division cellulaire. Enfin, la micro-injection de peptides mimant le site d’association entre 4E-BP et eIF4E dans les ovules affecte, de manière similaire aux effets de la rapamycine, la première division mitotique de l’embryon d’oursin [
21]. Ce résultat implique que la libération d’eIF4E de son répresseur 4E-BP est un événement nécessaire à l’entrée dans le cycle cellulaire de l’embryon précoce d’oursin. L’analyse des contrôles traductionnels au cours de la gamétogenèse a permis d’identifier de nouveaux acteurs qui, à l’instar de 4E-BP, sont capables de s’associer à eIF4E et d’inhiber compétitivement son interaction avec la protéine eIF4G. De tels partenaires pourraient influencer la traduction d’une population d’ARNm nécessaires pour la gamétogenèse, comme la formation des spermatozoïdes chez le nématode. |
Spermatogenèse chez le nématode Chez C. elegans, la spermatogenèse commence aux derniers stades larvaires : les spermatogonies prolifèrent par mitose, puis entrent en méiose jusqu’au stade pachytène, pour former les spermatocytes primaires. Ces derniers finissent leur première division méiotique pour donner les spermatocytes secondaires, puis poursuivent une deuxième division pour aboutir à des cellules haploïdes, les spermatides. Celles-ci subiront une maturation pour donner les spermatozoïdes. Chez C. elegans, où cinq formes d’eIF4E ont été identifiées, le taux d’eIF4E est élevé lors de la formation de spermatozoïdes [
7]. L’une des cinq isoformes d’eIF4E, IFE-1, interagit avec une protéine nommée PGL-1. Cette interaction s’effectue dans les granules P, des structures, riches en ARNm et en protéines, qui seraient déterminantes dans la formation de la lignée germinale. Lorsque l’expression d’IFE-1 est expérimentalement inhibée par interférence par l’ARN, il se produit un retard dans la spermatogenèse, et le sperme obtenu est déficient [7]. PGL-1 possède le site consensus d’association à eIF4E, et l’isoforme IFE-1 d’eIF4E ne peut plus interagir avec eIF4G lorsqu’elle est liée à PGL-1 : l’interaction IFE-1/PGL-1, qui baisse à partir du stade pachytène dans les spermatocytes primaires, permettrait la synthèse, à un instant précis, des ARNm spécifiques nécessaires à la formation des spermatozoïdes. La protéine IFE-1 pourrait également jouer un rôle indépendant de la traduction. Chez plusieurs organismes, eIF4E facilite le passage d’ARNm spécifiques du noyau vers le cytoplasme, après son entrée dans le noyau grâce au transporteur 4E-T [
22] : IFE-1 servirait ainsi de navette pour apporter dans le cytoplasme, à un instant donné, des ARNm spécifiques de la spermatogenèse, afin qu’ils puissent y être traduits [7]. L’existence des répresseurs de la protéine eIF4E tels que 4E-BP et PGL-1 ne permet cependant pas d’expliquer comment ce facteur général de la traduction peut contrôler la traduction d’un ARNm spécifique, ou d’une population d’ARNm. En effet, eIF4E ne possède pas d’activité intrinsèque lui permettant de recruter un ARNm donné. L’étude de régions cis régulatrices situées en 3’UTR de différents ARNm, ainsi que l’identification de nouveaux partenaires d’eIF4E au cours du développement embryonnaire ont permis d’apporter des éléments de réponse à cette question importante pour la connaissance des mécanismes de la régulation de l’expression des gènes. |
Traduction et facteurs trans/cis régulateurs Le lien que forme le complexe eIF4E/eIF4G entre les régions 5’ et 3’UTR stimule l’activité de traduction des ARNm. À l’inverse, la fixation d’un facteur protéique trans-régulateur négatif sur une région cis-régulatrice d’un ARNm affecte sa traductibilité. Dans l’embryon de drosophile, l’ARNm codant pour la protéine Bicoid est localisé, et sa traduction spatialement contrôlée, dans la région antérieure. Un élément de réponse de la protéine Nanos est présent dans la séquence 3’UTR de bicoid et cette région est capable de lier Pumilio [
23], suggérant une régulation traductionnelle de bicoid. La protéine Bicoid est non seulement un facteur de transcription à homéodomaine, qui contrôle la transcription de gènes de segmentation, mais aussi un régulateur traductionnel de caudal : elle interagit avec la région 3’UTR de caudal, et cette interaction est nécessaire à la répression de l’expression de la protéine Caudal dans la partie antérieure, l’ARNm caudal étant uniformément réparti dans l’embryon. Une séquence cis-régulatrice similaire à celle présente dans bicoid est également trouvée dans la région 3’UTR de l’ARNm codant pour la protéine GLP-1 [
24] de C. elegans. L’ARNm glp-1 code pour un récepteur de type Notch, qui intervient dans la voie de transduction en réponse aux interactions entre cellules et permet la spécification des axes dorsoventral et gauche-droite de l’embryon [
25]. L’ARNm glp-1 est soumis à au moins deux niveaux de contrôle traductionnel : un niveau temporel, la traduction de glp-1 étant inhibée dans les ovocytes et juste après la fécondation, puis activée après la première division, et un niveau spatial, glp-1 n’étant traduit que dans les blastomères antérieurs (sa traduction est inhibée dans les blastomères postérieurs). La région cis-régulatrice similaire à l’élément de réponse de Nanos observé chez la drosophile serait impliquée dans la répression de la traduction de glp-1 : l’établissement de la polarité embryonnaire pourrait donc comporter des mécanismes de contrôle traductionnel conservés au cours de l’évolution. Un signal cis-régulateur localisé à l’extrémité 3’UTR d’un ARNm est donc susceptible d’influencer le recrutement de celui-ci par la machinerie de traduction ; le fait que l’initiation de la traduction s’effectue en 5’UTR de l’ARNm suggère qu’il existe une communication entre les deux extrémités de l’ARNm, ce qui est en accord avec le modèle en boucle décrit précédemment. De nouveaux acteurs, impliqués dans le contrôle traductionnel de protéines jouant un rôle clé dans des étapes importantes de la maturation méiotique ou de la polarité antéropostérieure (A-P) des embryons, ont été identifiés. Ces acteurs contrôlent la traduction d’ARNm définis en associant le mécanisme commun d’inhibition compétitive d’eIF4G avec eIF4E à un mécanisme d’ancrage spécifique sur ces ARNm, via des facteurs trans-et cis-régulateurs. |
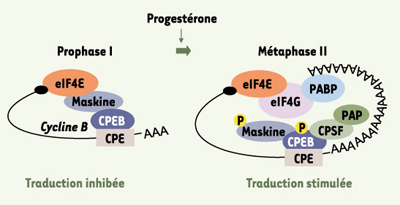
Maturation méiotique de l’ovocyte chez le xénope À l’issue de l’ovogenèse, les ovocytes ne sont pas systématiquement prêts à la fécondation ; ils doivent franchir une succession d’étapes, finement contrôlées, qui aboutit à un gamète femelle mature : l’ensemble de ces événements est appelé la maturation méiotique. L’ovocyte de xénope est naturellement bloqué en prophase de la première division méiotique (prophase I). La reprise des divisions est induite par la progestérone, qui provoque l’entrée de l’ovocyte dans le cycle cellulaire jusqu’au stade métaphase de la seconde division méiotique, stade auquel la méiose est à nouveau bloquée, jusqu’à la fécondation. Cette entrée dans le cycle cellulaire induit une augmentation de l’activité de synthèse protéique nécessaire pour le déroulement des étapes de la maturation méiotique. La poly-adénylation cytoplasmique de l’ARNm est un mécanisme hautement contrôlé au cours de la maturation méiotique de l’ovocyte, ainsi qu’au cours du développement embryonnaire [
26]. La poly-adénylation requiert deux éléments présents en 3’UTR : un hexanucléotide AAUAAA et un CPE, dont la séquence consensus est U4-5A1-2U. Cette dernière est portée par les ARNm des protéines telles que Mos, la Cycline B1 et les kinases Wee1 et Aurora. Dans les ovocytes en prophase I, la Maskine, identifiée comme un nouveau partenaire d’eIF4E, inhibe la synthèse protéique en empêchant la formation du complexe eIF4E/eIF4G. Dans le même temps, elle est associée à la protéine CPEB (Figure 3). Après stimulation par la progestérone, la protéine CPEB est phosphorylée par la kinase Aurora, ce qui stabilise la fixation du facteur CPSF en 3’UTR : celui-ci recrute la polymérase PAP, responsable de l’allongement de la queue poly (A) en 3’UTR. La protéine PABP interagit avec la queue poly (A) nouvellement synthétisée et avec eIF4G. Le complexe tend à déplacer la Maskine d’eIF4E, et la phosphorylation de la Maskine par cdk1 diminue son affinité pour eIF4E. Le retrait de la Maskine d’eIF4E laisse ainsi la place à eIF4G, qui recrute alors les partenaires impliqués dans la synthèse des protéines nécessaires à la maturation méiotique [
27]. 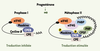 | Figure 3.
Poly-adénylation et recrutement d’ARNm au cours de la maturation méiotique de l’ovocyte de xénope. Dans l’ovocyte de xénope, avant la maturation méiotique induite par la progestérone, la traduction est réprimée par la protéine Maskine, qui limite le recrutement d’eIF4G sur eIF4E. La Maskine est également liée à la protéine CPEB, qui interagit en 3’UTR avec des ARNm tels que celui codant pour la Cycline B. La progestérone induit la reprise des synthèses protéiques après induction d’une voie de phosphorylation de la CPEB et de la Maskine. La phosphorylation de la CPEB stabilise l’association du CPSF, qui recrute PAP, polymérase responsable de l’allongement de la queue poly (A) de l’ARNm. La protéine PABP s’associe à la queue poly (A) nouvellement synthétisée, ainsi qu’au facteur eIF4G, ce complexe déplaçant la Maskine d’eIF4E : la protéine eIF4G associée à eIF4E peut alors participer au recrutement d’ARNm contrôlé de manière dépendante de la coiffe. |
La maturation méiotique permet donc d’illustrer le lien entre la poly-adénylation active d’un ARNm et son recrutement efficace au sein de la machinerie de la traduction. Le développement embryonnaire de la drosophile, en aval de contrôles transcriptionnels importants, requiert lui aussi des mécanismes de contrôles traductionnels sophistiqués pour la mise en place des axes embryonnaires. |
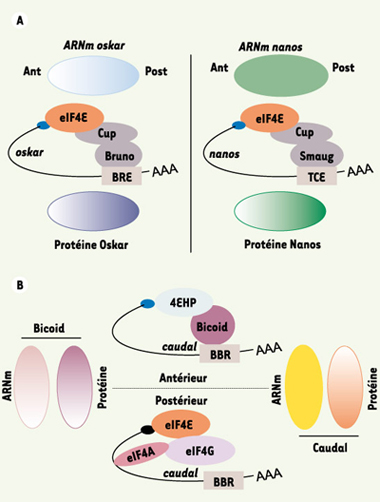
Mise en place des axes embryonnaires chez la drosophile Sans négliger l’importance incontestée des mécanismes de contrôle de l’expression des ARNm intervenant en amont de la traduction, le contrôle de la synthèse protéique est primordial pour l’établissement de la polarité embryonnaire A-P chez la drosophile. L’ARNm oskar s’accumule transitoirement dans la partie antérieure de l’embryon avant d’être relocalisé, par un mécanisme dépendant des microtubules, dans la partie postérieure, où s’effectue sa traduction [
28]. Pour s’assurer que la protéine sera uniquement synthétisée à l’arrière de l’embryon, la traduction est réprimée dans la région antérieure par l’intervention de Cup. Possédant le site consensus d’association à eIF4E, cette protéine est capable d’empêcher l’interaction d’eIF4E avec eIF4G (Figure 4A). Ce mécanisme implique la fixation de la protéine Bruno sur des séquences BRE en 3’UTR de l’ARNm oskar ; Bruno recrute alors Cup, et forme un complexe qui inhibe la traduction d’oskar. Au pôle postérieur des embryons, ce complexe est rompu par un mécanisme encore peu compris : la protéine Cup pourrait être phosphorylée à la manière des 4E-BP pour se dissocier d’eIF4E et laisser la place à eIF4G ; la protéine eIF4G pourrait également recruter des partenaires permettant de dissocier l’interaction entre Cup et eIF4E [28] ; enfin, la levée de l’inhibition pourrait être le résultat d’un allongement de la queue poly (A) de l’ARNm oskar qui, par un mécanisme similaire à celui de la rupture du complexe eIF4E/Maskine, permettrait de déstabiliser le complexe eIF4E/Cup [
29]. 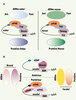 | Figure 4.
eIF4E et polarité embryonnaire. A. Contrôle traductionnel d’ARNm spécifiques par inhibition compétitive du complexe eIF4E/ eIF4G et ancrage via des facteurs trans- et cis-régulateurs. L’axe antéropostérieur de la drosophile nécessite la formation de gradients des protéines Oskar et Nanos. L’ARNm oskar est préférentiellement localisé dans la région postérieure des embryons, tandis que l’ARNm nanos se retrouve sur toute la surface embryonnaire. Le contrôle traductionnel est essentiel dans les deux cas pour s’assurer que les deux protéines ne s’exprimeront que dans la région postérieure. Le facteur eIF4E se lie à Cup, qui elle-même fixe des partenaires, Bruno ou Smaug, pour former des complexes spécifiques avec les ARNm oskar et nanos en 3’UTR. Ces associations, qui inhibent la formation de complexes eIF4E/eIF4G, doivent être rompues localement pour permettre aux protéines d’être synthétisées au bon endroit. B. Contrôle traductionnel d’un ARNm spécifique par un mécanisme de liaison compétitive à la coiffe. L’ARNm caudal est présent dans tout l’embryon, alors que sa protéine n’est exprimée que dans la région postérieure. Cette régulation fait intervenir 4EHP, une protéine homologue à eIF4E, qui d’un côté fixe la coiffe m7GTP pour entrer en compétition avec eIF4E, et de l’autre s’associe avec Bicoid, capable de reconnaître spécifiquement l’ARNm caudal sur la séquence BBR. Ce complexe inhibe la synthèse de Caudal dans la majorité de l’embryon. L’absence de Bicoid dans la région postérieure permet la production localisée de la protéine Caudal. |
De façon similaire, la protéine Nanos n’est présente qu’au pôle postérieur des embryons. Or, seulement 4 % des ARNm nanos se concentrent au pôle postérieur : il existe donc un système qui réprime la traduction de ces ARNm dans le reste de l’embryon. L’ARNm nanos possède une séquence TCE de 90 nucléotides en 3’UTR. Celle-ci est formée de deux structures « tige-boucle » permettant de recruter des facteurs inhibiteurs de la traduction [
30]. La structure est capable de recruter Smaug, une protéine qui, comme Bruno précédemment, possède un site de liaison pour Cup. Ce complexe eIF4E/Cup/Smaug inhibe la traduction de nanos dans l’embryon de drosophile (Figure 4A). Cependant, les ARNm nanos localisés au pôle postérieur doivent être traduits, et la répression doit donc être annulée : le mécanisme de dérépression, encore peu connu, nécessiterait la localisation postérieure d’Oskar, cette dernière étant capable de fixer Smaug et d’inhiber ainsi son interaction avec Cup. La protéine Oskar séquestrerait ainsi Smaug et permettrait la synthèse locale de Nanos [
31]. Par la suite, Nanos se fixe sur deux autres protéines, et exerce un effet inhibiteur sur la synthèse de Hunchback en région postérieure, pour déterminer la polarité embryonnaire. L’ensemble des mécanismes de contrôle de la traduction illustrés précédemment fait intervenir globalement un mécanisme commun d’inhibition compétitive de la protéine eIF4G par des protéines capables d’associer eIF4E sur un site consensus de liaison. Récemment identifié dans l’embryon de drosophile, un mécanisme de contrôle complètement différent vient compléter les possibilités de modulation de la traduction dépendante de la coiffe d’un ARNm spécifique. |
4EHP, un antagoniste d’eIF4E impliqué dans le contrôle de l’expression de Caudal chez la drosophile La mise en place de l’axe A-P nécessite également l’expression localisée du gène caudal dans la partie postérieure des embryons, alors que son ARNm est présent de façon homogène sur toute la surface embryonnaire. Cette régulation traductionnelle, indispensable pour la localisation précise de la protéine Caudal, fait intervenir les protéines 4EHP et Bicoid, qui vont réprimer la traduction de l’ARNm caudal dans la région antérieure. La protéine 4EHP, tout comme eIF4E, est capable de lier la coiffe m7GTP des ARNm, mais pas eIF4G. Si les deux protéines liant la coiffe sont réparties uniformément dans les embryons, Bicoid n’est présente que dans la région antérieure, résultat de la localisation spécifique de son ARNm [
32]. De par sa répartition, la protéine Bicoid se fixe sur une séquence BBR en 3’UTR de l’ARNm caudal uniquement dans la région antérieure (Figure 4B), où elle participe à l’inhibition traductionnelle de caudal. Dans un premier temps, Bicoid avait été identifiée comme s’associant à eIF4E [
33]. Récemment, il a été démontré que la protéine Bicoid n’interagit pas avec eIF4E, mais s’associe à la protéine homologue 4EHP [
34]. Elle favorise donc la fixation de 4EHP sur l’ARNm caudal au détriment d’eIF4E dans la région antérieure de l’embryon. La protéine 4EHP étant incapable de recruter eIF4G, l’ARNm caudal n’est pas traduit dans la région antérieure. La protéine 4EHP, inhibiteur spécifique de caudal, représente une nouvelle stratégie pour inhiber efficacement la traduction d’un ARNm défini. |
Les différents exemples cités précédemment soulignent l’importance de la régulation traductionnelle dans différentes étapes décisionnelles du développement embryonnaire. Les niveaux de contrôle de la disponibilité d’eIF4E, qui vont des régulations post-traductionnelles de répresseurs généraux, comme 4E-BP (phosphorylation et dégradation), à l’identification de nouveaux partenaires tels que Cup ou Bicoid, amènent à porter un regard nouveau sur la synthèse protéique. Nous avons volontairement limité notre argumentation au rôle central d’eIF4E et de ses partenaires, mais il est important de noter que, en aval du recrutement du 43S, d’autre mécanismes traductionnels, affectant notamment le balayage du 43S en 5’UTR [
35], la réunion des deux sous-unités ribosomiques [
36,
37] ou encore l’élongation [
38], sont susceptibles de moduler la production d’une protéine. Le contrôle du développement embryonnaire a souvent été réduit à l’expression de gènes « maîtres », correspondant à des facteurs de transcription. Cette vision réduisait les acteurs de la machinerie traductionnelle à des gènes « de ménage », seulement impliqués dans la traduction des ARNm présents. L’efficacité de la régulation traductionnelle et l’identification d’acteurs à double rôle au niveau de la transcription et de la traduction [
39], tel Bicoid, démontrent que chaque niveau de la régulation des gènes est capital. Ainsi, les régulations traductionnelles peuvent toucher l’expression de facteurs de transcription permettant à leur tour de synthétiser de nouveaux ARNm à traduire. Les « valets » contrôleraient alors les « maîtres »… La connaissance de ces réseaux et des régulations affectant les facteurs de traduction, ainsi que la recherche de nouveaux acteurs, se révèlent donc essentielles à la compréhension des premiers instants de la vie.

|
Les auteurs tiennent à remercier leurs collègues de l’équipe Cycle cellulaire et développement de l’UMR 7150, à la station biologique de Roscoff. L’équipe Cycle cellulaire et développement bénéficie du support financier de l’Association pour la recherche contre le Cancer (ARC n° 4247, 3507, France), de la Ligue nationale contre le Cancer (Délégation départementale du Finistère, France) et du Conseil régional de Bretagne.
|
Footnotes |
1. Mathews MB, Sonenberg N, Hershey JWB. Origins and principles of translational control. In : Hershey JWB, Mathews MB, Sonenberg N, eds. Translational control of gene expression. Cold Spring Harbor, New York : Cold Spring Harbor Laboratory Press : 2000 : 1–31. 2. Cormier P. Translational factors: from protein synthesis to cell cycle regulation and tumorigenesis. Med Sci (Paris) 2000; 16 : 378–85. 3. Gingras AC, Raught B, Sonenberg N. eIF4 initiation factors: effectors of mRNA recruitment to ribosomes and regulators of translation. Annu Rev Biochem 1999; 68 : 913–63. 4. Cormier P, Pyronnet S, Salaun P, et al. Cap-dependent translation and control of the cell cycle. In : Meijer L, Jezequel A. Roberge M, eds. Progress in cell cycle research. Roscoff : Éditions Life in Progress, 2003; 5 : 469–75. 5. Mamane Y, Petroulakis E, Rong L, et al. eIF4E-from translation to transformation. Oncogene 2004; 23 : 3172–9. 6. Klein PS, Melton DA. Induction of mesoderm in Xenopus laevis embryos by translation initiation factor 4E. Science 1994; 265 : 803–6. 7. Amiri A, Keiper BD, Kawasaki I, et al. An isoform of eIF4E is a component of germ granules and is required for spermatogenesis in C. elegans. Development 2001; 128 : 3899–912. 8. Cormier P, Pyronnet S, Morales J, et al. eIF4E association with 4E-BP decreases rapidly following fertilization in sea urchin. Dev Biol 2001; 232 : 275–83. 9. Robalino J, Joshi B, Fahrenkrug SC, Jagus R. Two zebrafish eIF4E family members are expressed and functionally divergent. J Biol Chem 2004; 279 : 10532–41. 10. Varani G. A cap for all occasions. Structure 1997; 5 : 855–8. 11. Von Der Haar T, Gross JD, Wagner G, Mc Carthy JE. The mRNA cap binding protein eIF4E in post transcriptional gene expression. Nat Struct Mol Biol 2004; 11 : 503–11. 12. Kahvejian A, Svitkin YV, Sukarieh R, et al. Mammalian poly(A)-binding protein is a eukaryotic translation initiation factor, which acts via multiple mechanisms. Genes Dev 2005; 19 : 104–13. 13. Pelletier J, Sonenberg N. Internal initiation of translation of eukaryotic mRNA directed by a sequence derived from poliovirus RNA. Nature 1988; 334 : 320–5. 14. Vagner S, Galy B, Pyronnet S. Irresistible IRES. Attracting the translational machinery to internal ribosome entry sites. EMBO Rep 2001; 2 : 893–8. 15. Le Breton M, Cormier P, Belle R, et al. Translational control during mitosis. Biochimie 2005; 87 : 805–11. 16. Ye X, Fong P, Iizuka N, et al.
Ultrabithorax and Antennapedia 5’untranslated regions promote developmentally regulated internal translation initiation. Mol Cell Biol 1997; 17 : 1714–21. 17. Bernal A, Kimbrell DA. Drosophila Thor participates in host immune defense and connects a translational regulator with innate immunity. Proc Natl Acad Sci USA 2000; 97 : 6019–24. 18. Salaün P, Pyronnet S, Morales J, et al. eIF4E/4E-BP dissociation and 4E-BP degradation in the first mitotic division of the sea urchin embryo. Dev Biol 2003; 255 : 428–39. 19. Morley SM, Coldwell MJ, Clemens MJ. Initiation factor modifications in the preapoptotic phase. Cell Death Diff 2005; 12 : 571–84. 20. Clemens MJ. Targets and mechanisms for the regulation of translation in malignant transformation. Oncogene 2004; 23 : 3180–8. 21. Salaun P, Boulben S, Mulner-Lorillon O, et al. Embryonic-stage-dependent changes in the level of eIF4E-binding proteins during early development of sea urchin embryos. J Cell Sci 2005; 118 : 1385–94. 22. Dostie J, Ferraiuolo M, Pause SA, Sonenberg N. A novel shuttling protein, 4E-T, mediates the nuclear import of the mRNA 5’ cap-binding protein, eIF4E. EMBO J 2000; 19 : 3142–56. 23. Gamberi C, Peterson DS, Gottlieb E. An anterior function for the Drosophila posterior determinant Pumilio. Development 2002; 129 : 2699–710. 24. Evans TC, Crittenden SL, Kodoyianni V, Kimble J. Translational control of maternal glp-1 mRNA establishes an asymetry in the C. elegans embryo. Cell 1994; 77 : 183–94. 25. Priess JR, Schnabel H, Schnabel R. The glp-1 locus and cellular interaction in early C. elegans embryos. Cell 1987; 51 : 601–11. 26. Richter JD. Cytoplasmic polyadenylation in development and beyond. Microbiol Mol Biol Rev 1999; 63 : 446–56. 27. Barnard D, Cao Q, Richter JD. Differential phosphorylation controls Maskin association with eukaryotic translation initiation factor 4E and localization on the mitotic apparatus. Mol Cell Biol 2005; 25 : 7605–15. 28. Nakamura A, Sato K, Hanyu-Nakurama K. Drosophila Cup is an eIF4E binding protein that associates with Bruno and regulates oskar mRNA in oogenesis. Dev Cell 2004; 6 : 69–78. 29. Wilhelm JE, Smibert CA. Mechanisms of translational regulation in Drosophila. Biol Cell 2005; 97 : 235–52. 30. Forrest KM, Clark IE, Jain RA, Gavis ER. Temporal complexity within a translational control element in the nanos mRNA. Development 2004; 131 : 5849–57. 31. Nelson M, Leidal AM, Smibert CA. Drosophila Cup is an eIF4E-binding protein that functions in Smaug-mediated translational repression. EMBO J 2004; 23 : 150–9. 32. Johnstone O, Lasko P. Translational regulation and RNA localization in Drosophila oocytes and embryos. Annu Rev Genet 2001; 35 : 365- 406. 33. Niessing D, Blanke S, Jackle H. Bicoid associates with the 5’-cap-bound complex of caudal mRNA and represses translation. Genes Dev 2002; 16 : 2576–82. 34. Cho PF, Poulin F, Cho-Park YA, et al. A new paradigm for translational control: inhibition via 5’-3’ mRNA tethering by Bicoid and the eIF4E cognate 4E-HP. Cell 2005; 121 : 411–23. 35. Beckmann K, Grskovic M, Gebauer F, Hentze MW. A dual inhibitory mechanism restricts msl-2 mRNA translation for dosage compensation in Drosophila. Cell 2005; 122 : 529–40. 36. Reimann I, Huth A, Thiele H, Thiele BJ. Suppression of 15-lipoxygenase synthesis by hnRNP E1 is dependent on repetitive nature of LOX mRNA 3’UTR control element DICE. J Mol Biol 2002; 5 : 965–74. 37. Ostareck-Lederer A, Ostareck DH, Cans C, et al. c-Src-mediated phosphorylation of hnRNP K drives translational activation of specifically silenced mRNAs. Mol Cell Biol 2002; 13 : 4535–43. 38. Proud C. Control of the elongation phase of protein synthesis. In : Hershey JWB, Mathews MB, Sonenberg N, eds. Translational control of gene expression. Cold Spring Harbor, New York : Cold Spring Harbor Laboratory Press : 2000 : 719–39. 39. Nédélec S, Trembleau A. Emx2 in axons: translational functions of homeodomain transcription factors. Med Sci (Paris) 2005; 21 : 237–9. |