| |
| Med Sci (Paris). 2006 April; 22(4): 374–380. Published online 2006 April 15. doi: 10.1051/medsci/2006224374.Les datations moléculaires à l’heure de la génomique Emmanuel J.P. Douzery,1* Frédéric Delsuc,1,2 and Hervé Philippe2* 1Laboratoire de paléontologie, phylogénie et paléobiologie-CC064, Institut des sciences de l’évolution UMR 5554/CNRS, Université Montpellier II, place E. Bataillon, 34095 Montpellier Cedex 05, France 2Canadian Institute for Advanced Research, Département de Biochimie, Centre Robert-Cedergren, Université de Montréal, CP 6128-Succursale Centre-ville, Montréal (Québec), H3C 3J7, Canada |
L’horloge moléculaire : des molécules pour remonter le temps 1
En 1965, Emile Zuckerkandl et Linus Pauling ont conçu une astucieuse machine à remonter le temps [
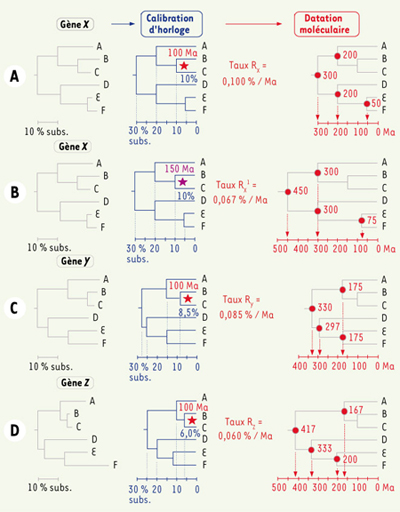
1]. Ils ont remarqué que les changements dans les macromolécules biologiques s’accumulaient de manière relativement constante au cours du temps. Ils eurent l’idée d’utiliser ce phénomène nommé « horloge moléculaire » pour transposer le degré de divergence moléculaire entre deux espèces en l’âge de leur ancêtre commun le plus récent. La datation moléculaire consiste ainsi à coupler un arbre phylogénétique décrivant les relations de parenté entre espèces, reconstruit à partir de séquences d’ADN ou de protéines, avec des points de calibration (ou étalonnages) paléontologiques afin d’estimer des âges absolus de divergence entre organismes. À partir du gène X, un arbre phylogénétique est inféré, et les longueurs de branches sont déduites à partir des changements observés dans les séquences. La quantité d’évolution allant de la racine de l’arbre à chacune des espèces actuelles (égale à la somme des longueurs de branches) est variable (Figure 1A). Cependant, de telles variations sont probablement dues à l’aspect stochastique de l’évolution moléculaire. Si ces différences de vitesse ne sont pas statistiquement significatives, l’arbre peut être converti en une phylogénie où tous les taxons terminaux A-F sont équidistants de la racine de l’arbre. Un tel arbre est dit ultramétrique et correspond à une parfaite horloge moléculaire globale. La paléontologie indiquant que les taxons B et C partagent un ancêtre commun âgé de 100 millions d’années (Ma), nous en déduisons que le taux de substitution nucléotidique du gène X est Rx = 0,100 % / Ma. Connaissant le degré de divergence génétique des taxons A-F de par la comparaison de leurs séquences (échelle violette), cette horloge moléculaire globale RX permet de déduire les âges de tous les nœuds de l’arbre (échelle rouge, exprimée en Ma).
 | Figure 1.
Les forces et les faiblesses de l’horloge moléculaire globale. A. Le principe. Au cours de l’histoire évolutive du gène X, les séquences des taxons A à F ont évolué à une vitesse similaire (à gauche, en gris). Les taxons - ou groupes taxonomiques - sont des groupes d’organismes considérés à un niveau donné de la classification : ce sont, par exemple, des espèces, des familles ou encore des ordres. En contraignant la phylogénie à respecter l’hypothèse d’horloge moléculaire, toutes les séquences évoluent à la même vitesse (au milieu, en violet). Les échelles grises et violettes sont exprimées en pourcentage de substitutions nucléotidiques (% substitutions [subs.]) : 10 % signifie ainsi que, pour 100 sites comparés, 10 substitutions ont été inférées le long de la branche correspondante lors de l’évolution du gène X. Un fossile dont la position phylogénétique et l’âge sont connus fournit maintenant une calibration temporelle. Les taxons B et C partagent un ancêtre commun dont l’âge paléontologique est de 100 Ma (étoile rouge). Sachant que les longueurs de branche conduisant à B et C correspondent chacune à 10 % de substitutions, nous en déduisons que le taux d’évolution absolu est de RX = 0,1 %/ Ma/lignée. La divergence moléculaire entre E et F étant de 5 %, nous en déduisons que ces deux taxons se sont séparés il y a 5/0,1 = 50 Ma, ainsi que tous les autres âges de divergence (à droite, en rouge). Notons que ces âges mesurés sont soit plus vieux (divergence A/B-C à 200 Ma), soit plus récents (divergence E/F à 50 Ma) que l’âge de calibration paléontologique. B. L’erreur paléontologique. Si une erreur paléontologique conduit à dater la divergence B/C à 150 Ma (étoile violette), alors le taux d’horloge devient RX’ = 0,067 %/ Ma/lignée. Tous les âges de divergence sont alors vieillis d’un facteur de 1,5. C. L’erreur stochastique. Le choix d’un locus donné (Y) plutôt qu’un autre (X) dans le génome peut avoir pour conséquence une erreur qualifiée de stochastique. En choisissant la même calibration paléontologique que précédemment (100 Ma), le gène Y conduit à une horloge moléculaire plus lente (RY) et fournit des âges de divergence différents de ceux estimés à partir du gène X. D. Absence d’horloge moléculaire. À un troisième locus, le gène Z présente des différences marquées de taux d’évolution, contrairement aux gènes X et Y. La phylogénie ultramétrique inférée (en violet) présente donc une très forte distorsion par rapport aux longueurs de branches originales, ce qui conduit à une horloge moléculaire artificielle (RZ) et à des âges estimés encore différents. |
L’horloge moléculaire a rapidement suscité un vif intérêt. Elle offre, en effet, la possibilité d’estimer les âges d’apparition des espèces appartenant à des groupes pour lesquels aucun renseignement paléontologique n’était disponible. Tel est le cas, par exemple, de la plupart des micro-organismes. Cependant, l’existence d’une horloge moléculaire, c’est-à-dire d’un taux d’évolution apparemment constant à long terme, n’exclut pas la possibilité de fluctuations à court terme autour d’une valeur moyenne. En fait, les horloges en évolution moléculaire se comportent plus de manière stochastique que selon le rythme régulier de véritables métronomes. |
Les forces et les faiblesses de l’horloge moléculaire Depuis sa formulation, le concept d’horloge moléculaire a été largement appliqué. Dès les années 1960, Sarich et Wilson [
2] suggérèrent que l’homme s’est séparé de ses plus proches parents, le chimpanzé et le gorille, il y a environ 5 Ma, alors que des paléontologues proposaient plutôt un âge de 30 Ma pour la séparation entre la lignée humaine et celles des grands singes. Le consensus fut néanmoins que l’estimation moléculaire était beaucoup plus proche de la date réelle (estimée actuellement à environ 7 Ma) que la date paléontologique proposée à l’époque. Un grand optimisme dans les potentialités de l’horloge moléculaire en a découlé, ce qui s’est traduit par une multitude de résultats. Par exemple, Korber et al. [
3] ont suggéré que l’ancêtre commun le plus récent des principales souches de VIH date des années 1915-1941, c’est-à-dire longtemps avant la pandémie actuelle. Cet optimisme initial a cependant été tempéré par l’observation de désaccords difficilement réconciliables entre âges paléontologiques et moléculaires. L’exemple le plus célèbre concerne probablement le cas des animaux métazoaires. L’examen du registre fossile indique leur subite diversification à la base du Cambrien, soit il y a environ 540 Ma. Cela est généralement interprété comme résultant d’événements rapides de spéciations communément désignés par l’expression « explosion cambrienne » [
4]. De manière nettement moins consensuelle, les estimations moléculaires de l’âge de diversification des métazoaires diffèrent quasiment du simple au double, allant de 582 Ma [
5] à 976 Ma [
6], en passant par 573-656 Ma [
7], 670-736 Ma [
8] ou encore 830 Ma [
9] ! Le fait que les âges moléculaires soient plus anciens que les âges paléontologiques ne constitue pas une surprise. En effet, la découverte du plus vieux fossile d’un groupe taxonomique n’est jamais garantie et la divergence génétique entre espèces précède leur divergence morphologique (deux organismes morphologiquement indiscernables peuvent appartenir à des espèces différentes et donc accumuler des différences génétiques). Cependant, des écarts temporels d’une telle amplitude sont déconcertants. Comme l’illustre la Figure 1, certaines limites propres au principe de l’horloge moléculaire pourraient expliquer ces désaccords. Remarquons que ce sont les fossiles qui fournissent les références géologiques nécessaires pour accéder au temps absolu - généralement exprimé en millions d’années. Si, par suite d’une erreur d’identification du fossile ou de datation de la strate à laquelle il appartient, l’âge de la divergence entre les taxons B et C est porté de 100 à 150 Ma, alors le taux d’évolution du gène X devient RX’ = 0,067% / Ma. Les répercussions sur les âges de divergence sont immédiates : elles vieillissent tous les nœuds d’un facteur de 1,5 (Figure 1B). Dans de nombreuses études de datation moléculaire, une unique référence paléontologique est généralement considérée ; de plus, elle est dépourvue de son inhérente incertitude (par exemple, le célèbre point de calibration 310 ± 0 Ma pour la divergence mammifères/oiseaux [
10]). Les inévitables erreurs sur ces rares points de calibration vont ainsi affecter les âges mesurés [
11]. Le choix des gènes peut, lui aussi, avoir une répercussion importante sur les estimations. Utilisons, par exemple, un second gène, Y. Les gènes X et Y représentant des échantillonnages de sites nucléotidiques tirés de deux emplacements (locus) indépendants du génome, la phylogénie inférée à partir de Y présente des longueurs de branches différentes de celle fondée sur X, principalement pour des raisons stochastiques. Les branches conduisant à B et C ont maintenant une longueur de 8,5 % (Figure 1C). Sur la phylogénie avec horloge moléculaire, le taux d’évolution vaut donc RY = 0,085 % / Ma ; par conséquent, on constate que les âges mesurés par le gène Y varient localement de manière substantielle par rapport au gène X (voir les nœuds au sein du groupe D + E + F). C’est pour réduire l’effet stochastique que certains chercheurs ont choisi d’utiliser un grand nombre de gènes [6] ; malheureusement c’est au détriment du nombre d’espèces considérées et donc du nombre de points de calibration [10]. Considérons enfin le cas d’un troisième gène, Z. Il n’évolue manifestement pas à un taux constant (Figure 1D), ce qui constitue le cas le plus répandu. Afin de le prouver statistiquement, il existe des tests de détection des écarts par rapport à l’hypothèse d’horloge moléculaire. Par exemple, en utilisant un cadre probabiliste, il est possible de savoir si l’arbre avec horloge moléculaire est significativement moins vraisemblable que l’arbre sans horloge. Si ces tests de détection ne sont pas assez performants, ce qui est généralement le cas [
12,
13], la transformation abusive de l’arbre en phylogénie avec horloge moléculaire introduit d’importantes distorsions dans les longueurs de branches. Les courtes branches du groupe B + C conduisent à des longueurs valant 6 % dans l’arbre d’horloge moléculaire et donc à un faible taux d’évolution Rz = 0,060 % / Ma. L’application de ce taux erroné pour la datation introduit d’importantes erreurs dans les âges de divergence mesurés par le gène Z, en particulier pour les plus anciens. |
Les solutions : les horloges moléculaires assouplies Trois écueils majeurs empêchent la réalisation de datations moléculaires fiables : (1) la prise en compte d’un nombre limité d’espèces et de gènes ; (2) l’incorporation de calibrations fossiles isolées et fixées ; (3) l’existence d’hétérogénéités de taux d’évolution entre lignées. Pour ce qui est de l’échantillonnage taxonomique, la prise en compte d’un nombre conséquent d’espèces permet d’obtenir une phylogénie plus fiable [
14] ainsi qu’une meilleure estimation des longueurs de branches, donc des taux d’évolution et des temps de divergence. Pour ce qui est de l’échantillonnage génomique, de multiples gènes ou protéines doivent être considérés afin de ne pas rendre les estimations d’âges de divergence trop dépendantes du choix d’un seul locus et de l’importante erreur stochastique qui lui est associée [
15]. Avec le progrès des méthodes de séquençage, le premier écueil ne constitue plus un problème insurmontable. Les points (2) et (3) requièrent cependant de considérables raffinements méthodologiques ; ils ont été entrepris ces dernières années. Le couplage fossiles/molécules a été amélioré par des méthodes de datation pouvant (a) incorporer simultanément plusieurs calibrations paléontologiques et (b) considérer ces dernières comme des intervalles de temps plutôt que comme des points fixes dépourvus d’incertitude [
16,
17]. Les importantes variations de taux d’évolution observées pour de nombreuses espèces ont conduit au développement de méthodes de datation moléculaire ne faisant pas appel à l’hypothèse, trop forte, d’une horloge moléculaire dite globale, c’est-à-dire appliquée à l’ensemble de la phylogénie considérée. C’est ainsi que la méthode dite des horloges moléculaires locales a été proposée, en supposant qu’il peut y avoir des taux de substitution constants dans une région de l’arbre, malgré des variations de taux à de plus grandes échelles phylogénétiques [
18]. Cette approche se heurte cependant à la difficulté que représente l’identification objective des ensembles de branches qui vont évoluer selon une même horloge moléculaire locale. Plutôt que de rester contraint par l’horloge moléculaire, des chercheurs ont proposé d’assouplir cette hypothèse en modélisant l’évolution des taux d’évolution le long des branches de l’arbre phylogénétique. Sanderson [16] a été le premier à mettre en œuvre un lissage des taux d’évolution dans lequel les écarts entre le taux de la branche descendante et celui de la branche parentale - immédiatement ascendante - sont limités. Par la suite, d’autres modèles de variation des taux d’évolution ont été envisagés sur différentes bases mathématiques [5,
19,
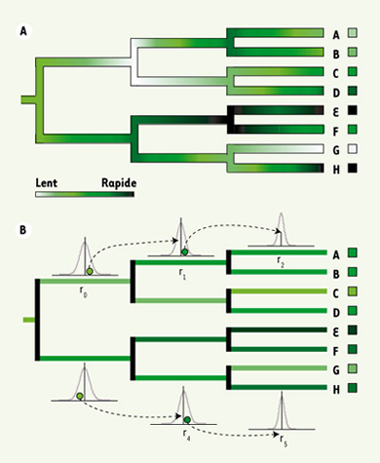
20]. Nous n’allons présenter ici que le modèle d’horloge assouplie qui est actuellement le plus utilisé. Ce dernier se fonde sur l’observation, essentielle, de l’héritabilité du taux d’évolution. En effet, au moment même où deux taxons se séparent par spéciation, leurs taux d’évolution respectifs à un locus donné sont identiques (voir les branches verticales sur la Figure 2A). Par la suite, des différences de taux d’évolution peuvent se propager indépendamment le long des deux branches descendantes : partageant initialement un ancêtre commun à taux intermédiaire, les taxons G et H sont maintenant caractérisés par des taux respectivement lent et rapide. Plus l’échantillonnage taxonomique est dense, meilleure sera la délimitation de ces variations de taux d’évolution le long des branches de l’arbre phylogénétique (Figure 2A). 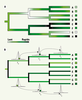 | Figure 2.
Les horloges moléculaires assouplies. A. Les variations graduelles de taux d’évolution le long des branches d’une phylogénie. À partir de la racine de cette phylogénie, différentes trajectoires conduisent aux taux observés chez les taxons A à H symbolisés dans les carrés correspondants. Les taux d’évolution, de lents jusqu’à rapides, varient le long des branches horizontales. Les branches verticales représentent les événements de spéciation. Ces derniers produisent deux espèces descendantes qui, au moment où elles se séparent l’une de l’autre, possèdent le même taux d’évolution hérité de l’espèce ancestrale dont elles sont issues. Trois principaux cas de figure se présentent ensuite : les espèces accélèrent (C et D), ralentissent (A et B) ou bien l’une accélère (H) tandis que l’autre ralentit (G). Enfin, une espèce peut évoluer de manière quasiment constante, ici rapide (E). B. Le modèle des horloges assouplies avec autocorrélation des taux. Les variations graduelles de taux présentées ci-dessus sont modélisées de la manière suivante. Chaque branche est caractérisée par un unique taux, qui est la moyenne de son taux initial et de son taux final. Le taux r1 d’une branche descendante est tiré (disque vert) dans une distribution normale centrée sur le taux r0 de la branche ascendante. Le taux r1 est ici plus rapide que r0. Les autres relations d’ordre illustrées sont : r2 > r1 > r0, r3 > r4, et r5 > r4. Une variance de cette distribution normale des taux respectivement importante ou faible entraîne un écart important ou faible par rapport à l’hypothèse d’horloge moléculaire. |
Le passage de cette observation biologique à la modélisation se fait en considérant que le taux d’évolution le long d’une branche descendant d’un nœud est a priori autocorrélé à celui de la branche ascendante : leurs moyennes sont les mêmes, à une certaine variance près, estimée à partir des données (Figure 2B). Si cette variance est a posteriori proche de zéro, les taux des branches descendantes seront quasiment identiques à celui de la branche ascendante, se rapprochant en cela d’une horloge moléculaire globale. Plus cette variance est importante, plus les taux des branches descendantes sont différents du taux parental. Cela permet de modéliser d’importantes variations de taux se propageant le long de la phylogénie (Figure 2B). En pratique, connaissant les séquences comparées au départ et les calibrations fossiles, la distribution des âges de divergence et des taux d’évolution est estimée de façon à identifier les valeurs qui maximisent la probabilité d’observer l’arbre phylogénétique avec ses longueurs de branches. Ce modèle d’assouplissement de l’hypothèse de l’horloge moléculaire a été appliqué dans un cadre statistique bayésien [17,
21]. De manière importante, les différentes approches d’horloge assouplie produisent des estimations d’âges de divergence couplées à des incertitudes généralement exprimées sous forme d’intervalles de crédibilité. Les incertitudes ont souvent été négligées dans les datations moléculaires classiques, ce qui a fait artificiellement croire à une grande précision des horloges moléculaires [10]. En fait, la connaissance de cette incertitude est essentielle non seulement pour quantifier la précision du signal de datation moléculaire présent dans les données génomiques mais encore pour permettre une meilleure comparaison avec le registre fossile. |
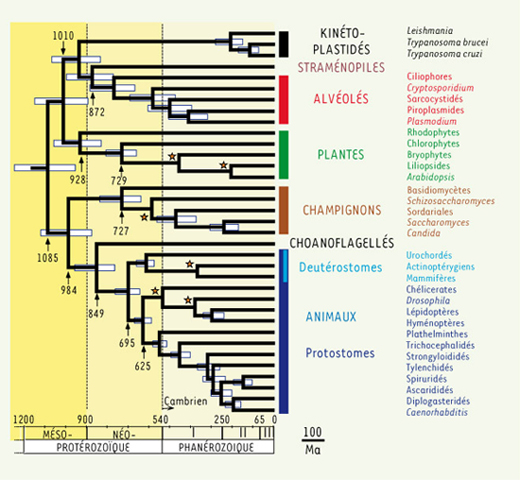
Les âges moléculaires des métazoaires et des mammifères La comparaison de centaines de gènes ou de protéines chez des dizaines d’espèces permet de réduire l’erreur stochastique au niveau moléculaire. En élargissant les possibilités de calibration, elle réduit aussi l’erreur paléontologique. Dans une récente étude [
22], nous avons ainsi tenté d’estimer l’âge de diversification des principaux rameaux d’eucaryotes. Nous avons considéré la concaténation en une super-protéine de 129 protéines nucléaires (participant à la transcription, à la traduction et au métabolisme cellulaire ou encore constitutives du cytosquelette), représentant un total de 30 399 sites d’acides aminés alignés de manière non ambiguë. L’échantillonnage taxonomique comprenait 36 eucaryotes (15 animaux, un choanoflagellé, 5 champignons, 5 plantes et 10 protistes), une raisonnable représentativité des principales lignées eucaryotes - notamment au sein des métazoaires. Six références paléontologiques, empruntées aux animaux, champignons et plantes et réparties en différentes régions de l’arbre ont permis la calibration. L’incertitude paléontologique a été incorporée en considérant les bornes temporelles récentes et anciennes des couches stratigraphiques auxquelles appartenaient les fossiles de référence. D’importantes variations de taux d’évolution existant dans nos données - les trypanosomes et nématodes évoluent par exemple 2 à 3 fois plus rapidement que les mammifères - l’application de l’horloge moléculaire globale est proscrite (Figure 1D) ; nous avons donc utilisé l’approche bayésienne d’assouplissement de l’horloge moléculaire (Figure 2). En tenant compte des intervalles de crédibilité associés aux estimations d’âges de divergence, il y a 95 % de chances que les principales lignées d’eucaryotes se soient diversifiées il y a 950-1259 Ma, que les animaux se soient séparés de leurs plus proches parents, les choanoflagellés, il y a 761-957 Ma, et que l’âge débattu de la diversification des métazoaires puisse se situer entre 642-761 Ma (Figure 3). Cette dernière estimation suggère que les animaux bilatériens aient pu se diversifier environ 100 Ma avant l’explosion cambrienne dont témoigne le registre fossile. Ce décalage pourrait s’expliquer par des lacunes des connaissances paléontologiques, directement causées par le fait que les premiers bilatériens étaient probablement des animaux à corps mou ayant pu échapper aux processus de fossilisation. Il faut pourtant noter que des fossiles vieux de 600 Ma, récemment découverts en Chine, ont été identifiés comme appartenant aux bilatériens [
23]. Un meilleur accord entre les datations moléculaire et paléontologique de l’évolution des métazoaires semble donc se dessiner, bénéficiant des progrès conjoints des horloges moléculaires et des archives fossiles.  | Figure 3.
La datation moléculaire des eucaryotes. Les horloges moléculaires assouplies ont été ici appliquées à 129 protéines nucléaires provenant de 36 eucaryotes ; elles comportent six intervalles de calibration (étoiles) (d’après [ 22]). Les intervalles de crédibilité sur les âges estimés sont délimités par les rectangles horizontaux. Les âges en millions d’années (Ma) sont indiqués pour quelques nœuds représentatifs. |
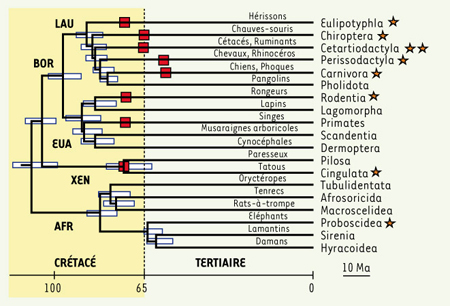
Plus proche de nous, l’âge de la diversification des mammifères placentaires offre un autre exemple de décalage entre estimations paléontologiques et moléculaires. Il a longtemps été considéré que les placentaires ont bénéficié des extinctions de la fin de l’ère secondaire, notamment celle des dinosaures, pour se diversifier au début de l’ère Tertiaire, il y a 65 Ma. Du point de vue moléculaire, Springer et al. [
24] ont estimé les âges de divergence des principaux groupes de mammifères placentaires en tenant compte de manière similaire des problèmes taxonomiques, génomiques, paléontologiques et méthodologiques susmentionnés. Adoptant la même approche bayésienne que précédemment pour calibrer l’horloge moléculaire assouplie, les auteurs ont considéré 42 mammifères, analysant en parallèle 16 kb d’ADN mitochondrial et nucléaire et 9 contraintes paléontologiques. Les résultats suggèrent qu’une diversification des mammifères placentaires est survenue au cours du Crétacé, il y a environ 100 Ma, et indiquent qu’au moment où les dinosaures s’éteignaient, il y a environ 65 Ma, la plupart des ordres de placentaires étaient déjà apparus, sinon diversifiés (Figure 4). L’occupation de niches écologiques laissées vacantes par l’extinction des dinosaures n’aurait donc pas été le facteur déclenchant à l’origine de la radiation évolutive des ordres modernes de mammifères placentaires [
25].  | Figure 4.
La datation moléculaire des mammifères. Les horloges moléculaires assouplies ont été ici appliquées à des données mitochondriales et nucléaires provenant de 42 mammifères ; elles comportent neuf intervalles de calibration choisis au sein de différents ordres (étoiles) (d’après [ 24]). Les intervalles de crédibilité sur les âges estimés sont délimités par les rectangles horizontaux. Les carrés représentent le moment où les ordres correspondants se sont diversifiés en familles. Les ordres modernes de mammifères placentaires apparaissent au Crétacé et se diversifient aussi bien avant qu’après la transition Crétacé/Tertiaire (pointillés verticaux). Les grands groupes de placentaires sont les Afrothériens (AFR), Xénarthres (XEN), Euarchontoglires (EUA), Laurasiathériens (LAU), et Boréoeuthériens (EUA + LAU). Le nom courant de quelques membres des différents ordres de placentaires sont donnés sur les branches. |
Notons pour conclure que les horloges moléculaires assouplies sont utilisées dans des groupes taxonomiques très variés pour répondre à diverses questions de biologie évolutive. Par exemple, il a longtemps été considéré que l’actuelle distribution géographique des espèces de hêtre austral (genre Nothofagus) résulte d’un phénomène de vicariance - c’est-à-dire d’une division de l’aire de répartition ancestrale par une barrière géographique - provoqué ici par la fragmentation du Gondwana, à l’origine notamment de la séparation de l’Australie et de la Nouvelle-Zélande, il y a 80 Ma. Or, les datations moléculaires de Knapp et al. [
26] suggèrent plutôt des âges de divergence entre les hêtres australiens et néozélandais sensiblement plus récents (aux alentours de 30 Ma). La biogéographie des espèces du genre Nothofagus a donc également été gouvernée par des événements de dispersion transocéanique. Ces résultats remettent en cause l’un des principaux exemples d’évolution par vicariance chez les plantes en soulignant l’importance, auparavant insoupçonnée, de la dispersion à longue distance des graines. |
Des allers-retours permanents entre les datations moléculaires et paléontologiques sont plus que jamais nécessaires. En effet, l’étude des fossiles sert à calibrer les horloges moléculaires, qu’elles soient globales, locales ou encore assouplies. Les datations moléculaires qui en découlent permettent alors d’éprouver les hypothèses biologiques, paléontologiques et biogéographiques existantes ; elles conduisent parfois à de nouvelles propositions concernant la chronologie de l’évolution des organismes. En cas de désaccord entre les estimations fondées sur les génomes et sur les fossiles, une analyse critique des données moléculaires et paléontologiques est incontournable. À l’avenir, la disponibilité en données moléculaires va augmenter grâce à la génomique comparative, mais des modèles plus performants de description des variations de taux d’évolution restent à développer. Du côté de la paléontologie, des progrès sont attendus dans l’exploration de gisements inédits et dans l’amélioration des méthodes d’analyse des fossiles, notamment à l’aide de techniques tridimensionnelles. Ces études multidisciplinaires nous aideront à mieux connaître la chronologie des différents événements à l’origine des espèces fossiles et actuelles, et ainsi à mieux comprendre les mécanismes évolutifs, sources de la biodiversité terrestre. |
Nous remercions vivement Vincent Ranwez, Béatrice Roure et deux arbitres anonymes pour leurs commentaires constructifs. Ce travail a bénéficié du soutien de l’ACI Informatique-mathématique-physique en biologie moléculaire [ACI IMP-Bio], de « Génome Québec » et représente la contribution n° 2005-067 de l’Institut des sciences de l’évolution de Montpellier (UMR 5554 - CNRS).
|
Footnotes |
1. Zuckerkandl E, Pauling L. Evolutionary divergence and convergence in proteins. In : Bryson V, Vogel HJ, eds. Evolving genes and proteins. New York : Academic Press, 1965 : 97–166. 2. Sarich VM, Wilson AC. Immunological time scale for hominoid evolution. Science 1967; 158 : 1200–3. 3. Korber B, Muldoon M, Theiler J,et al. Timing the ancestor of the HIV-1 pandemic strains. Science 2000; 288 : 1789–96. 4. Conway Morris S. The Cambrian "explosion" : slow-fuse or megatonnage ? Proc Natl Acad Sci USA 2000; 97 : 4426–9. 5. Aris-Brosou S, Yang Z. Effects of models of rate evolution on estimation of divergence dates with special reference to the metazoan 18S ribosomal RNA phylogeny. Syst Biol 2002; 51 : 703–14. 6. Hedges SB, Blair JE, Venturi ML,et al. A molecular timescale of eukaryote evolution and the rise of complex multicellular life. BMC Evol Biol 2004; 4 : 2. 7. Peterson KJ, Lyons JB, Nowak KS,et al. Estimating metazoan divergence times with a molecular clock. Proc Natl Acad Sci USA 2004;101 : 6536–41. 8. Ayala FJ, Rzhetsky A, Ayala FJ. Origin of the metazoan phyla : molecular clocks confirm paleontological estimates. Proc Natl Acad Sci USA 1998; 95 : 606–11. 9. Gu X. Early metazoan divergence was about 830 million years ago. J Mol Evol 1998; 47 : 369–71. 10. Graur D, Martin W. Reading the entrails of chickens : molecular timescales of evolution and the illusion of precision. Trends Genet 2004; 20 : 80–6. 11. Douzery EJP, Delsuc F, Stanhope MJ,et al. Local molecular clocks in three nuclear genes : divergence times for rodents and other mammals, and incompatibility among fossil calibrations. J Mol Evol 2003; 57 : S201–13. 12. Philippe H, Sörhannus U, Baroin A,et al. Comparison of molecular and paleontological data in diatoms suggests a major gap in the fossil record. J Evol Biol 1994; 7 : 247–65. 13. Bromham L, Penny D, Rambaut A,et al. The power of relative rates tests depends on the data. J Mol Evol 2000; 50 : 296–301. 14. Lecointre G, Philippe H, Lê HLV,et al. Species sampling has a major impact on phylogenetic inference. Mol Phylogenet Evol 1993; 2 : 205–24. 15. Delsuc F, Brinkmann H, Philippe H. Phylogenomics and the reconstruction of the tree of life. Nat Rev Genet 2005; 6 : 361–75. 16. Sanderson MJ. A nonparametric approach to estimating divergence times in the absence of rate constancy. Mol Biol Evol 1997; 14 : 1218–31. 17. Kishino H, Thorne JL, Bruno WJ. Performance of a divergence time estimation method under a probabilistic model of rate evolution. Mol Biol Evol 2001; 18 : 352–61. 18. Yoder AD, Yang Z. Estimation of primate speciation dates using local molecular clocks. Mol Biol Evol 2000; 17 : 1081–90. 19. Huelsenbeck JP, Larget B, Swofford D. A compound poisson process for relaxing the molecular clock. Genetics 2000; 154 : 1879–92. 20. Welch JJ, Bromham L. Molecular dating when rates vary. Trends Ecol Evol 2005; 20 : 320–7. 21. Thorne JL, Kishino H, Painter IS. Estimating the rate of evolution of the rate of molecular evolution. Mol Biol Evol 1998; 15 : 1647–57. 22. Douzery EJP, Snell EA, Bapteste E,et al. The timing of eukaryotic evolution : Does a relaxed molecular clock reconcile proteins and fossils ? Proc Natl Acad Sci USA 2004; 101 : 15386–91. 23. Chen JY, Bottjer DJ, Oliveri P,et al. Small bilaterian fossils from 40 to 55 million years before the Cambrian. Science 2004; 305 : 218–22. 24. Springer MS, Murphy WJ, Eizirik E,et al. Placental mammal diversification and the Cretaceous-Tertiary boundary. Proc Natl Acad Sci USA 2003; 100 : 1056–61. 25. Bromham L, Phillips MJ, Penny D. Growing up with dinosaurs : molecular dates and the mammalian radiation. Trends Ecol Evol 1999; 14 : 113–8. 26. Knapp M, Stöckler K, Havell D,et al. Relaxed molecular clock provides evidence for long-distance dispersal of Nothofagus (Southern beech). PLoS Biol 2005; 3 : e14. |