Classiquement, une mutation stop précoce dans un gène est considérée comme une mutation nulle pour deux raisons : d’une part, le court fragment peptidique produit, correspondant au début de la protéine, n’est en général pas suffisant pour assurer la fonction ; d’autre part, les mutations stop prématurées sont connues pour déclencher la dégradation des ARNm par un système de surveillance des transcrits aberrants, le nonsense mediated decay (NMD) [ 1]. Le NMD a été montré dans le cas des gènes poly-exoniques lorsque la mutation stop se trouve en amont de la dernière jonction exonique. Cependant, une mutation stop précoce n’est pas toujours nulle. En effet, il existe plusieurs mécanismes permettant d’assurer la traduction malgré la présence d’un stop prématuré, comme la ré-initiation traductionnelle. Ce mécanisme permet la synthèse d’un fragment protéique à partir d’un site d’initiation en aval du codon stop précoce. Si le cadre de lecture est conservé, le produit qui en résulte correspond à la protéine délétée de sa région amino-terminale. Il a été montré que ce processus permet de « sauver » l’expression de certains gènes [ 2]. Par ailleurs la ré-initiation traductionnelle a la particularité de diminuer l’efficacité du NMD [ 3].
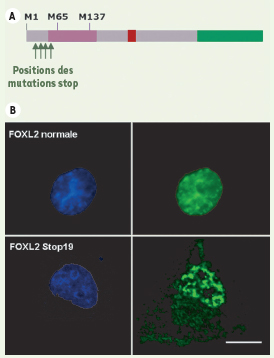
Depuis quelques années, notre équipe s’intéresse au BPES (blepharophimosis ptosis epicanthus inversus syndrome), comportant des malformations cranio-faciales (essentiellement palpébrales) associées dans certains cas à une insuffisance ovarienne prématurée. Il s’agit d’une maladie génétique rare à transmission autosomique dominante causée par les mutations du gène FOXL2 [ 4]. FOXL2 est un gène mono-exonique codant pour un facteur de transcription à domaine forkhead. FOXL2 comporte en outre une polyalanine (14 résidus), de fonction inconnue, qui est retrouvée essentiellement dans des facteurs se liant à l’ADN ou à l’ARN (Figure 1A). Différents types de mutations ont été identifiés dans la séquence codante de FOXL2. L’expansion de la polyalanine de 14 à 24 résidus est la plus fréquente puisqu’elle est retrouvée dans 30 % des cas de BPES [ 5]. Au niveau protéique, l’expansion de la polyalanine entraîne l’agrégation de FOXL2 et sa rétention dans le cytoplasme alors que la protéine sauvage (14 alanines) se localise uniquement dans le noyau de façon diffuse [ 6]. Ce phénomène d’agrégation liée à des expansions de répétitions homopolymériques est bien connu pour les protéines à polyglutamine [ 7] et a récemment été décrit dans d’autres protéines à polyalanine [ 8]. Nous nous sommes récemment intéressés à un autre type de mutations de FOXL2, les mutations stop précoces [ 9]. Étant donné qu’il est dépourvu d’introns, FOXL2 n’est a priori pas soumis au mécanisme de NMD. Nous nous sommes alors demandé si le mécanisme de reprise de la traduction pouvait se produire lorsque FOXL2 contient un codon stop précoce. Pour répondre à cette question, nous avons exprimé dans des cellules de mammifère (COS-7) des formes de FOXL2 contenant des codons stop précoces, en fusion avec la GFP (green fluorescent protein) en position carboxy-terminale. En cas de ré-initiation traductionnelle, la protéine de fusion produirait une fluorescence verte des cellules. Nous avons testé la ré-initiation pour quatre codons stop en position 19, 31, 41 et 53 (cette dernière mutation étant décrite pour un patient BPES) (Figure 1A). L’observation des cellules au microscope nous a permis de voir que, pour toutes les positions, la traduction d’une protéine de fusion a lieu. De façon inattendue, les protéines de fusion observées ne se répartissent pas de façon diffuse et uniquement nucléaire comme la protéine sauvage. En effet, elles sont fortement agrégées dans les noyaux de toutes les cellules qui les expriment (Figure 1B). De plus, 70 % des cellules présentent une localisation cytoplasmique de la protéine. En estimant la taille des protéines de fusion, nous avons montré que, pour les mutations stop 19 et 31, la traduction reprend essentiellement au niveau du codon AUG 65 et dans une moindre mesure en AUG 137. L’efficacité de la ré-initiation a été estimée à environ 25 % dans les deux cas. En revanche, dans le cas des mutations stop 41 et 53, il n’y a pas de ré-initiation en AUG 65 et une faible expression de la protéine démarrant en 137 est détectée. Cette faible expression est également observée avec la forme sauvage de FOXL2 et semble correspondre à un site d’entrée interne des ribosomes. Ces résultats suggèrent que le phénomène de ré-initiation dépend de la position du codon stop précoce. On peut ainsi définir une position critique entre les codons 31 et 41 à partir de laquelle il ne peut plus y avoir de ré-initiation traductionnelle efficace.
| Figure 1.
A. Représentation schématique de la protéine FOXL2. Le cadre violet représente le domaine forkhead de liaison à l’ADN. Le cadre rose représente la polyalanine. Le cadre vert représente la GFP. Les positions 19, 31, 41 et 53 des codons stop sont indiquées par des flèches. M1, M65 et M137 sont les méthionines respectivement codées par les codons AUG 1, AUG 65 et AUG 137. B. Microscopie à fluorescence des cellules COS-7 exprimant les formes de FOXL2 sauvage (en haut) et Stop19 (en bas). En bleu (Hœchst, à gauche), coloration des noyaux. En vert (à droite), protéines de fusion avec la GFP (échelle : 16 µm). |
De façon surprenante, les protéines de fusion dépourvues des 64 premiers acides aminés s’agrègent. Cela suggère que certains de ces acides aminés sont impliqués dans la solubilité de FOXL2. Pour mieux comprendre ce phénomène, nous avons réalisé une série de délétions dans la région amino-terminale et quantifié l’agrégation. Cela nous a permis de montrer que ce sont les 18 premiers acides aminés du domaine forkhead qui seraient impliqués dans la solubilité. Ces 18 acides aminés font partie de la première hélice de ce domaine. Nous avons ensuite cherché à déterminer les régions protéiques responsables de l’agrégation de la protéine dépourvue de sa région amino-terminale. Nous avons d’abord montré que la GFP n’induisait pas l’agrégation. Des expériences de délétions de différentes régions nous ont permis de montrer que : (1) le forkhead seul tronqué de ces 18 premiers acides aminés ne s’agrège pas, ce qui suggère l’implication d’autres régions de la protéine dans l’agrégation ; (2) la polyalanine et la région carboxy-terminale ne sont pas impliquées dans l’agrégation. Il semble donc que c’est la région située entre le domaine forkhead et la polyalanine qui est réellement déterminante pour l’agrégation.
En conclusion, nous avons montré qu’une mutation stop dans FOXL2, en fonction de sa position, peut entraîner un phénomène de ré-initiation traductionnelle et conduire à la synthèse d’une protéine fortement agrégée. De plus, nous avons montré que la protéine ainsi produite est capable de retenir une fraction de la protéine normale dans les agrégats nucléaires, suggérant un possible effet dominant négatif de la protéine tronquée [9]. Pour l’instant, l’absence de mutations stop identifiées en amont du codon 31 chez des patients BPES ne permet pas de vérifier cette hypothèse. Ces résultats élargissent le spectre des mutations pouvant conduire à des agrégats protéiques. L’agrégation avait précédemment été montrée pour des expansions de polyglutamine et de polyalanine ainsi que pour des substitutions d’acides aminés [7, 8]. Par ailleurs, ces résultats pointent l’importance du phénomène de ré-initiation et ses conséquences. En effet, dans certains cas, une reprise de la traduction peut sauver un phénotype. Dans d’autres cas, elle pourrait conduire à la synthèse de protéines toxiques ou ayant un effet dominant négatif. Dans les cas de mutations stop précoces associées à des phénotypes inattendus, il apparaît fondamental d’explorer la possibilité d’une ré-initiation traductionnelle.