| |
| Med Sci (Paris). 2006 January; 22(1): 62–67. Published online 2006 January 15. doi: 10.1051/medsci/200622162.USF : un régulateur essentiel de la transcription Sébastien Corre and Marie-Dominique Galibert* CNRS UMR 6061, Laboratoire de génétique et développement, Faculté de Médecine, Université de Rennes 1, 2, avenue du Professeur Léon Bernard, 35043 Rennes Cedex, France |
Les facteurs de transcription USF (upstream stimulating factors)
La purification biochimique des éléments de régulation en trans du promoteur majeur tardif de l’adénovirus (MLPA) [
1] a permis d’identifier et de caractériser les facteurs de transcription USF-1 et -2 (upstream stimulating factor 1, 2), respectivement de 43 et 44 kDa [
2,
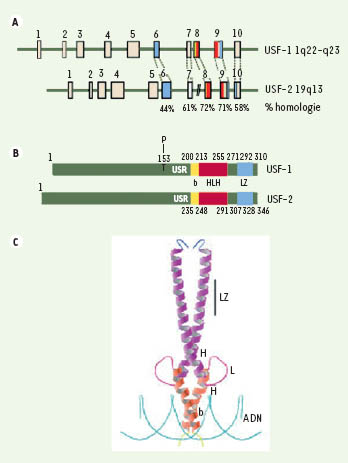
3]. Ces protéines sont codées par deux gènes distincts USF-1 et USF-2, localisés respectivement chez l’homme en 1q22-23 et 19q13 (Figure 1A) [
4,
5]. L’organisation des gènes USF-1 et -2 est comparable, avec 10 exons répartis sur 4 et 10 kb, respectivement (Figure 1A) [
6]. Les facteurs USF-1 et -2 possèdent une homologie de séquence importante au niveau de leurs ADNc et de leurs protéines [
7]. De même, le niveau de conservation des protéines entre espèces (homme, rat, souris) est élevé, pouvant atteindre 99 % d’identité [
8]. De fait, les protéines USF font partie de la super-famille des b-HLH-LZ (basic-helix-loop-helix-leucine zipper) [
9] qui comprend, entre autres, les protéines eucaryotes des sous-familles Myc (Myc, Max, Mad, Mxi1), TFE3 (transcription factor binding to IGHM enhancer 3) (TFE3, TFEB, TFEC) et Mitf (microphtalmia-associated transcription factor). 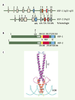 | Figure 1.
Structure des gènes et protéines des facteurs de transcription USF (upstream stimulating factor). A. Les gènes codant pour les facteurs USF-1 (1q22-q23) et USF-2 (19q13) sont composés de 10 exons répartis sur 4 et 10 kb respectivement, et présentent une bonne homologie de séquence, particulièrement dans la région carboxyterminale. B. USF-1 et USF-2 appartiennent à la famille conservée des facteurs de transcription de type b-HLH-LZ. Le domaine basique b (jaune) intervient dans la liaison à l’ADN alors que les domaines hélice-boucle-hélice HLH (rouge) et leucine zipper LZ (bleu) interviennent dans la dimérisation. La présence de l’USR (USF specific region) en amont de la région basique est nécessaire à l’activité transcriptionnelle. Pour être actif, USF-1 est phosphorylé par la kinase p38 sur la thréonine 153. C. Structure cristallographique des complexes b-HLH-LZ/ADN. Les protéines USF-1 et -2 interagissent entre elles formant des homo- ou des hétérodimères. LZ : leucine zipper ; bHLH : basic helix-loop-helix. |
Les protéines b-HLH-LZ se caractérisent par la présence d’un domaine de liaison à l’ADN (région basique) et de deux domaines de dimérisation (régions HLH et LZ) (Figure 1B, 1C). La présence des domaines HLH et LZ contigus distingue ces protéines des facteurs de transcription de type bHLH ou bZip qui ne possèdent qu’un seul domaine de dimérisation. La région basique est impliquée dans l’interaction avec l’ADN au niveau de boîtes E, de séquences consensus CANNTG [
10]. Les structures de dimérisation HLH et LZ permettent l’établissement de liaisons spécifiques entre les membres d’une même famille, même si le domaine LZ participe également à la stabilisation du complexe ADN-protéines. L’établissement de la structure cristallographique des complexes b-HLH-LZ/ADN a permis de mettre en évidence les interactions spécifiques établies entre acides aminés et ADN, nécessaires au maintient du complexe transcriptionnel. Les protéines USF-1 et - 2 interagissent principalement entre elles, conduisant à la formation d’homo- ou d’hétérodimères [
11]. |
Propriétés de liaison à l’ADN La séquence palindromique des boîtes E (CANNTG) constitue le site de liaison spécifique des facteurs de transcription de type b-HLH et b-HLH-LZ. La petite taille du motif (six nucléotides) explique leur grande fréquence à travers le génome. Cependant, seul un nombre limité de motifs E constitue de véritables éléments régulateurs de l’expression des gènes. En effet, la spécificité de reconnaissance et d’interaction des boîtes E avec les dimères protéiques est dépendante de la nature des deux nucléotides centraux qui sont généralement GC ou CG. Ainsi, le facteur de transcription USF-1 se lie au motif CACGTG avec une meilleure affinité qu’à la séquence CATGTG qui correspond au motif de liaison du facteur de transcription Mitf. De même, la variation du nombre de nucléotides entre les deux hémi-sites CA et TG abolit les interactions protéines/ADN. Les nucléotides de part et d’autre de la séquence consensus, CANNTG, affectent également la fixation des facteurs de transcription. Ainsi, la présence d’un résidu thymidine en 5’ du motif E est indispensable à la liaison spécifique du facteur de transcription Mitf, alors que ce même résidu empêche la liaison au motif E du facteur de transcription Myc. Une analyse in silico de la composition nucléotidique des boîtes E des promoteurs de gènes régulés par USF-1, avec, entre autres, le promoteur majeur tardif de l’adénovirus, celui de l’hème oxygénase, de la métallothionéine, du gène APC, de l’ADN topo-isomérase IIIα et de la désoxycytidine kinase, met en évidence la présence constante de résidus AC en position 3’ de la boîte E. Cette observation suggère que le facteur de transcription USF-1 reconnaît spécifiquement les sites CACGTGAC. Enfin, l’affinité de la liaison du facteur de transcription USF-1 au motif E peut varier en présence de SNP (single nucleotide polymorphism) affectant le motif E [
12] ou par méthylation de l’îlot CpG central à la boîte E [
13], modulant ainsi l’activation transcriptionnelle de gènes cibles. |
Régulateurs majeurs de la machinerie de transcription L’épissage différentiel des transcrits USF-1 et -2 engendre différentes formes de protéines à activité transcriptionnelle distincte (présence ou non de domaine de transactivation). L’activité transcriptionnelle des dimères USF varie selon la nature du dimère formé qui est dépendante de la quantité d’isoformes USF présente au sein de chaque cellule [
14] et des modifications post-traductionnelles propres à chaque isoforme. Ainsi, les variations protéiques quantitatives et qualitatives au sein de chaque tissu, induisent vraisemblablement une régulation distincte de l’expression des gènes dépendants de USF. La famille de protéines USF participe ainsi à différents niveaux de la régulation transcriptionnelle avec, premièrement, la reconnaissance et la liaison aux motifs E spécifiques présents au niveau des séquences de régulation de gènes cibles. Deuxièmement, les interactions protéines-protéines coopératives, impliquant les protéines USF-1 et des facteurs de transcription ubiquitaires ou spécifiques de tissu avec, respectivement, les facteurs SP1 [
15], PEA3 [
16] ou MTF1 [
17], permettent de moduler la régulation transcriptionnelle de gènes cibles. Troisièmement, le facteur de transcription USF-1 interagit directement avec le facteur TFIID (transcription factor II) et certains TAF (TATA binding protein-associated factor) [
18] présents dans le complexe de pré-initiation (PIC) de la transcription de gènes « TATA-plus ». De même, dans les cas de promoteur de type « TATA-less », USF-1 est capable d’interagir avec le facteur TFII-I impliqué dans la reconnaissance de la séquence initiatrice (Inr) ou directement avec la séquence Inr. Par ailleurs, les données structurales et biochimiques suggèrent que le facteur USF-1 existe sous forme de tétramère permettant la liaison simultanée de deux sites distincts. Finalement, les protéines USF-1 sont capables de recruter des enzymes (CAF et SET7/9) impliquées dans l’acétylation et la méthylation de certaines histones [
19] à l’origine d’un remodelage de la chromatine, favorisant l’ouverture et l’accès de la machinerie de transcription. De même, la liaison des facteurs USF aux boîtes E du gène de la topo-isomérase III (hTOP3α) permet la régulation de son expression qui entraîne des changements topologiques de l’ADN. Les modifications des « supertours » de la structure favorisent ainsi l’accès des zones de régulation de la transcription par les facteurs de la machinerie de transcription, en permettant le déroulement de la double hélice. Les facteurs USF sont donc largement impliqués dans la machinerie transcriptionnelle, en accord avec une expression ubiquitaire et une large distribution des motifs spécifiques de reconnaissance à travers le génome. Le rôle essentiel des protéines USF est d’ailleurs mis en évidence par le phénotype létal des souris double knock-out USF-1 et USF-2 [
20]. L’activité transcriptionnelle des facteurs USF ne se limite toutefois pas aux gènes du développement embryonnaire ; ils ont été également impliqués dans l’activation transcriptionnelle d’un grand nombre de gènes intervenant dans une large variété de fonctions et systèmes dont certains seront décrits dans cet article. |
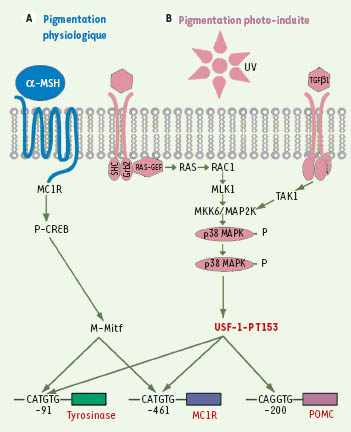
USF-1 dans les mélanocytes et la pigmentation UV-dépendante Les cellules de la peau constituent la première barrière de protection de l’organisme contre les agressions physiques, chimiques ou biologiques de l’environnement. Parmi elles, les rayonnements ultraviolets constituent le plus important de ces dangers, à l’origine de pathologies cutanées. La pigmentation permet la protection de la peau contre les rayonnements UV, la photo-cancérogenèse cutanée et le photo-aging. L’accumulation des pigments constitue une barrière vis-à-vis des effets néfastes provoqués par le soleil (dommages de l’ADN à type de dimères de cyclobutane pyrimidine et 6-4 pyrimidine pyrimidone, mais aussi d’anomalies des protéines). L’acquisition d’un phénotype pigmenté repose sur la coopération entre deux types cellulaires, les kératinocytes et les mélanocytes. Ces derniers sont des cellules dendritiques où siège la synthèse de la mélanine. Ils reposent sur la membrane basale de l’épiderme et interagissent avec les kératinocytes adjacents. Le mélanocyte, isolé au sein d’une population de kératinocytes (environ un pour quarante), intègre les signaux émis par les kératinocytes et permet une protection efficace grâce à la synthèse du pigment photoprotecteur, la mélanine. Cette synthèse se déroule dans les mélanosomes, qui sont des organites spécifiques transférés vers les kératinocytes environnants, permettant ainsi une protection globale de la peau. La production de mélanine est le résultat d’un réseau complexe impliquant des facteurs paracrines sécrétés par les kératinocytes (α-melanocyte specific hormone) ou α-MSH codée par le gène de la pro-opiomélanocortine ou POMC, endothéline et FGF-2) et leurs récepteurs spécifiques (respectivement, MC1R [melanocortin 1 receptor], EDNRB [endothelin receptor type B] et récepteur tyrosine kinase). Ce réseau implique également plusieurs facteurs de transcription (Mitf, USF-1) ainsi que des gènes impliqués directement dans la synthèse du pigment (tyrosinase, TRP-1 et Dct) [
21]. Alors que le facteur de transcription Mitf intervient dans la pigmentation constitutive [
22], le facteur de transcription USF-1, phosphorylé par la kinase p38 en réponse au stress UV, est impliqué dans la réponse pigmentaire [
23,
24]. Le facteur de transcription USF-1 augmente l’expression des gènes de la pigmentation POMC, MC1R, tyrosinase (Figure 2), en réponse à une stimulation par les UV [23, 24]. Ainsi, les cellules MEL USF1−/−
établies à partir des souris knock-out USF-1 ne répondent plus aux stimulations UV. Enfin, différentes boucles d’amplification, faisant intervenir la kinase p38 et le facteur de transcription USF-1, permettent d’augmenter le processus de pigmentation pour une protection efficace de la peau (Figure 2). 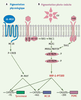 | Figure 2.
Régulation de la pigmentation photo-induite par USF-1. A. La pigmentation constitutive de la peau fait intervenir la voie AMPc activée par la fixation de l’α-MSH (α-melanocyte stimulating hormone) à son récepteur MC1R (melanocortin 1 receptor), et relayée par le facteur de transcription Mitf (microphtalmia-associated transcription factor). B. Le facteur USF-1 est, quant à lui, impliqué dans la réponse pigmentaire induite par les UV. À la suite d’une stimulation par les rayonnements solaires, la phosphorylation d’USF-1 par la kinase p38 est activée, permettant l’induction de l’expression des gènes de la pigmentation (TYR, pro-opiomélanocortine ou POMC, MC1R) par USF-1 après fixation aux boîtes E (CANNTG). TGFβ1 : transforming growth factor β1 ; SHC : Src homology 2 domain containing ; Grb2 : Growth factor receptor-bound protein 2 ; RAS-GEF : protéines Ras-GEF (guanine-nucleotide exchange factor) ; RAC1 : RAS-related C3 botulinum toxin substrate 1 ; MLK1 : mixed lineage kinase 1 ; TAK1 : TGF-β-activated kinase 1 ; MKK6/MAP2K : MAP kinase kinase 2 ; p38 MAPK : p38 mitogen-activated protein kinase ; P-CREB : phosphorylated cAMP-responsive element-binding protein. |
|
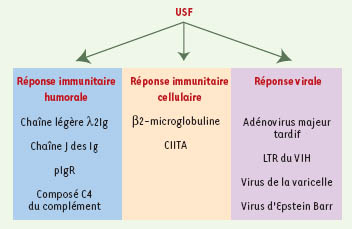
USF-1 et la réponse immunitaire L’implication du facteur USF-1 comme élément de réponse aux stress permet de mieux comprendre son rôle dans la réponse immunitaire. En effet, de nombreux gènes exprimés en réponse à une infection virale ou bactérienne sont régulés par la protéine USF-1. Ainsi, USF-1 participe à la régulation transcriptionnelle de gènes de la réponse humorale et cellulaire (Figure 3). Les boîtes E sont des éléments régulateurs essentiels des promoteurs des chaînes d’immunoglobulines [
25] et USF-1 a été impliqué dans la régulation de la transcription du gène codant la chaîne légère l2 [
26]. De plus, au cours de la réponse immunitaire précoce, la formation de pentamère d’IgM dépend de la synthèse simultanée des chaînes J des immunoglobulines qui permettent l’assemblage des monomères. Ensuite, leur transport à travers l’épithélium muqueux et glandulaire est assuré par le récepteur polymérique des immunoglobulines (pIgR) [
27]. L’expression concomitante des gènes codant l’IgJ et le récepteur pIgR est essentielle pour une défense efficace et est en partie sous le contrôle du facteur USF-1. Enfin, l’élimination des antigènes pathogènes par cytolyse ou phagocytose est obtenue après reconnaissance et liaison spécifique des immunoglobulines. Le facteur USF-1 participe à cette phase de la réponse immunitaire en régulant l’expression d’un gène du système du complément, le C4 [7] et le gène de la β2-microglobuline [
28], qui code la glycoprotéine impliquée dans la présentation de l’antigène, en association avec le complexe majeur d’histocompatibilité (CMH) de classe I. USF-1 active également la transcription du gène CIITA [
29] qui est un intermédiaire obligatoire de l’induction des gènes de CMH de classe II par l’interféron dans la réponse cellulaire. Par ailleurs, une dégradation spécifique d’USF-1 a été décrite en présence du pathogène intracellulaire Chlamydia, abolissant la reconnaissance par les lymphocytes T. Un détournement de la protéine USF-1 au profit de la machinerie transcriptionnelle virale a également été observé avec différents virus dont l’adénovirus majeur tardif, le LTR du VIH, le virus de la varicelle et le virus d’Eptsein-Barr [
30]. Le rôle des protéines USF est donc double, participant à la réponse immunitaire et à la prolifération virale. 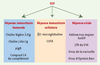 | Figure 3.
Régulation de la réponse immunitaire par USF. Les facteurs de transcription USF permettent la régulation de la réponse immunitaire humorale et cellulaire en régulant l’expression de différents gènes. Par ailleurs, les virus détournent ces facteurs à leur profit pour la transcription de leur génome. pIgR : récepteur polymérique des immunoglobulines ; CIITA : class II transactivator. |
|
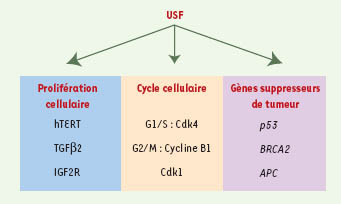
USF et le contrôle de la prolifération cellulaire La progression du cycle cellulaire est régulée de manière précise par les complexes cyclines-Cdk (cyclin-dependent-kinase) pour assurer le transfert de l’intégrité de l’information génétique des cellules mères aux cellules filles [
31]. Ainsi, la régulation de la transition G1/S est sous le contrôle des cyclines D et E, en combinaison avec les Cdk2, 4 et 6, alors que la transition G2/M est dépendante des cyclines A et B en combinaison avec la Cdk1 (Cdc2). Le facteur de transcription USF-1 est un régulateur de l’expression des gènes codant pour la cycline B1 et Cdk1, favorisant ainsi la transition G2/M [
32,
33], et USF-2 régule l’expression du gène Cdk4 (Figure 4). Par ailleurs, la phosphorylation de la protéine recombinante USF-1, en présence des complexes cyclineA2-cdk1 et cyclineB1-cdk1, augmente son affinité pour le motif E et stimule l’activité transcriptionnelle, ce qui suggère la présence d’une boucle d’amplification favorisant la transition G2/M. Le rôle des protéines USF dans la prolifération cellulaire ne se limite pas aux gènes du cycle cellulaire proprement dits. En effet, USF-1 permet de réguler l’expression du gène hTERT (human telomerase reverse transcriptase) impliqué dans le maintien de l’intégrité des extrémités télomériques [
34]. Dans les cellules différenciées où le gène hTERT n’est plus exprimé, la taille des télomères est directement liée au nombre de divisions cellulaires, et donc à la durée de vie de la cellule, à l’inverse des cellules souches totipotentes qui possèdent une activité télomérase. De même, 85 % des cellules cancéreuses se caractérisent par la réactivation de l’expression du gène hTERT. Dans les cellules cancéreuses, USF-1 participerait au maintien de l’expression du gène hTERT sans pour autant être impliqué dans sa réactivation. Bien que USF-1 participe à la prolifération illimitée dans les cellules cancéreuses étudiées, son rôle dans la pathologie cancéreuse est controversé. En effet, une perte d’activité transcriptionnelle de la protéine USF-1 a été observée dans trois lignées de carcinomes mammaires sur six étudiées. Cette modification d’activité, qui est de spécificité cellulaire, semble être dépendante de la perte de cofacteurs (voir plus haut TFIID, TAF), essentiels pour la machinerie transcriptionnelle. De même, l’implication d’USF-1 dans la régulation de l’expression de gènes suppresseur de tumeur (p53, BRCA2, APC) semble en opposition avec la régulation du gène hTERT [
35] (Figure 4). Le débat sur le rôle d’USF-1 dans le processus cancéreux reste donc ouvert. 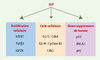 | Figure 4.
Régulation de la prolifération cellulaire et du cycle par USF. Les facteurs de transcription USF régulent positivement l’expression de gènes impliqués dans la prolifération cellulaire. USF intervient également dans les transitions G1/S et G2/M en régulant l’expression de cyclines et de Cdk. Il est également impliqué dans la régulation de gènes suppresseurs de tumeur. |
|
Les facteurs de transcription USF, de la famille des b-HLH-LZ, participent à la régulation d’un réseau de gènes se caractérisant par la présence d’éléments de régulation spécifiques appelés boîte E (CANNTG). Les travaux récents montrent que les gènes régulés par les facteurs USF sont de plus en plus nombreux, permettant d’impliquer USF dans de nouveaux processus cellulaires. Les fonctions des facteurs USF sont dépendantes du contexte cellulaire à l’origine d’interactions protéine/protéine spécifiques, de modifications post-transcriptionnelles et post-traductionnelles distinctes. Ces paramètres, associés aux diverses modifications des motifs E (méthylation, SNP) spécifiques de tissu ou de patient, modulent l’activité transcriptionnelle des facteurs USF. La présence de ces nombreux paramètres rend compte de la réelle complexité et diversité des processus de régulation des gènes dépendants d’USF. |
Footnotes |
1. Sawadogo M, Roeder RG. Interaction of a gene-specific transcription factor with the adenovirus major late promoter upstream of the TATA box region. Cell 1985; 43 : 165–75. 2. Sawadogo M. Multiple forms of the human gene-specific transcription factor USF. II. DNA binding properties and transcriptional activity of the purified HeLa USF. J Biol Chem 1988; 263 : 11994–2001. 3. Sawadogo M, Van Dyke MW, Gregor PD, Roeder RG. Multiple forms of the human gene-specific transcription factor USF. I. Complete purification and identification of USF from HeLa cell nuclei. J Biol Chem 1988; 263 : 11985–93. 4. Shieh BH, Sparkes RS, Gaynor RB, Lusis AJ. Localization of the gene-encoding upstream stimulatory factor (USF) to human chromosome 1q22-q23. Genomics 1993; 16 : 266–8. 5. Groenen PM, Garcia E, Debeer P, et al. Structure, sequence, and chromosome 19 localization of human USF2 and its rearrangement in a patient with multicystic renal dysplasia. Genomics 1996; 38 : 141–8. 6. Lin Q, Luo X, Sawadogo M. Archaic structure of the gene encoding transcription factor USF. J Biol Chem 1994; 269 : 23894–903. 7. Galibert MD, Boucontet L, Goding CR, Meo T. Recognition of the E-C4 element from the C4 complement gene promoter by the upstream stimulatory factor-1 transcription factor. J Immunol 1997; 159 : 6176–83. 8. Henrion AA, Vaulont S, Raymondjean M, Kahn A. Mouse USF1 gene cloning : comparative organization within the c-myc gene family. Mamm Genome 1996; 7 : 803–9. 9. Atchley WR, Fitch WM. A natural classification of the basic helix-loop-helix class of transcription factors. Proc Natl Acad Sci USA 1997; 94 : 5172–6. 10. Baxevanis AD, Vinson CR. Interactions of coiled coils in transcription factors : where is the specificity ? Curr Opin Genet Dev 1993; 3 : 278–85. 11. Sirito M, Walker S, Lin Q, et al. Members of the USF family of helix-loop-helix proteins bind DNA as homo- as well as heterodimers. Gene Expr 1992; 2 : 231–40. 12. Mandola MV, Stoehlmacher J, Muller-Weeks S, et al. A novel single nucleotide polymorphism within the 5’ tandem repeat polymorphism of the thymidylate synthase gene abolishes USF-1 binding and alters transcriptional activity. Cancer Res 2003; 63 : 2898–904. 13. Prendergast GC, Ziff EB. Methylation-sensitive sequence-specific DNA binding by the c-Myc basic region. Science 1991; 251 : 186–9. 14. Viollet B, Lefrancois-Martinez AM, Henrion A, et al. Immunochemical characterization and transacting properties of upstream stimulatory factor isoforms. J Biol Chem 1996; 271 : 1405–15. 15. Liu M, Whetstine JR, Payton SG, et al. Roles of USF, Ikaros and Sp proteins in the transcriptional regulation of the human reduced folate carrier B promoter. Biochem J 2004; 383 : 249–57. 16. Firlej V, Bocquet B, Desbiens X, et al. Pea3 Transcription factor cooperates with USF-1 in regulation of the murine bax transcription without binding to an Ets-binding site. J Biol Chem 2005; 280 : 887–98. 17. Andrews GK, Lee DK, Ravindra R, et al. The transcription factors MTF-1 and USF1 cooperate to regulate mouse metallothionein-I expression in response to the essential metal zinc in visceral endoderm cells during early development. EMBO J 2001; 20 : 1114–22. 18. Chiang CM, Roeder RG. Cloning of an intrinsic human TFIID subunit that interacts with multiple transcriptional activators. Science 1995; 267 : 531–6. 19. West AG, Huang S, Gaszner M, et al. Recruitment of histone modifications by USF proteins at a vertebrate barrier element. Mol Cell 2004; 16 : 453–63. 20. Casado M, Vallet VS, Kahn A, Vaulont S. Essential role in vivo of upstream stimulatory factors for a normal dietary response of the fatty acid synthase gene in the liver. J Biol Chem 1999 274 : 2009–13. 21. Kadekaro AL, Kavanagh RJ, Wakamatsu K, et al. Cutaneous photobiology. The melanocyte versus the sun : who will win the final round ? Pigment Cell Res 2003; 16 : 434–47. 22. Busca R, Ballotti R. Cyclic AMP a key messenger in the regulation of skin pigmentation. Pigment Cell Res 2000; 13 : 60–9. 23. Galibert MD, Carreira S, Goding CR. The Usf-1 transcription factor is a novel target for the stress-responsive p38 kinase and mediates UV-induced tyrosinase expression. EMBO J 2001; 20 : 5022–31. 24. Corre S, Primot A, Sviderskaya E, et al. UV-induced expression of key component of the tanning process, the POMC and MC1R genes, is dependent on the p-38 activated upstream stimulating factor-1 (USF-1). J Biol Chem 2004; 279 : 51226–33. 25. Ephrussi A, Church GM, Tonegawa S, Gilbert W. B lineage : specific interactions of an immunoglobulin enhancer with cellular factors in vivo. Science 1985; 227 : 134–40. 26. Chang LA, Smith T, Pognonec P, et al. Identification of USF as the ubiquitous murine factor that binds to and stimulates transcription from the immunoglobulin lambda 2-chain promoter. Nucleic Acids Res 1992; 20 : 287–93. 27. Wallin JJ, Rinkenberger JL, Rao S, et al. B cell-specific activator protein prevents two activator factors from binding to the immunoglobulin J chain promoter until the antigen-driven stages of B cell development. J Biol Chem 1999; 274 : 15959–65. 28. Gobin SJ, Biesta P, Van den Elsen PJ. Regulation of human beta 2-microglobulin transactivation in hematopoietic cells. Blood 2003; 101 : 3058–64. 29. Muhlethaler-Mottet A, Di Berardino W, Otten LA, Mach B. Activation of the MHC class II transactivator CIITA by interferon-gamma requires cooperative interaction between Stat1 and USF-1. Immunity 1998; 8 : 157–66. 30. Liu C, Sista ND, Pagano JS. Activation of the Epstein-Barr virus DNA polymerase promoter by the BRLF1 immediate-early protein is mediated through USF and E2F. J Virol 1996; 70 : 2545–55. 31. Hunt T. Cyclins and their partners : from a simple idea to complicated reality. Semin Cell Biol 1991; 2 : 213–22. 32. Cogswell JP, Godlevski MM, Bonham M, et al. Upstream stimulatory factor regulates expression of the cell cycle-dependent cyclin B1 gene promoter. Mol Cell Biol 1995; 15 : 2782–90. 33. North S, Espanel X, Bantignies F, et al. Regulation of cdc2 gene expression by the upstream stimulatory factors (USFs). Oncogene 1999; 18 : 1945–55. 34. Goueli BS, Janknecht R. Regulation of telomerase reverse transcriptase gene activity by upstream stimulatory factor. Oncogene 2003; 22 : 8042–7. 35. Reisman D, Rotter V. The helix-loop-helix containing transcription factor USF binds to and transactivates the promoter of the p53 tumor suppressor gene. Nucleic Acids Res 1993; 21 : 345–50. |