| |
| Med Sci (Paris). 2005 December; 21(12): 1025–1027. Published online 2005 December 15. doi: 10.1051/medsci/200521121025.Dynamique d’assemblage des machineries nucléolaires après la mitose, en temps réel et en cellules vivantes Danièle Hernandez-Verdun* Institut Jacques Monod, 2, place Jussieu, 75251 Paris Cedex 05, France MeSH keywords: Protéines nucléaires, Organisateur nucléolaire, Cartographie d'interactions entre protéines, ARN ribosomique |
Le noyau des cellules est réorganisé après chaque mitose. Ce qui implique la reformation de l’enveloppe nucléaire, la décondensation de la chromatine et la réorganisation des machineries nucléaires. Il faut réadresser les machineries au bon endroit au bon moment pour rassembler un noyau fonctionnel.
 | Figure 1.
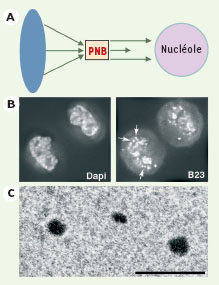
Les PNB (prenucleolar bodies). A. Schéma représentant la formation des PNB en sortie de mitose. Les protéines nucléolaires à la périphérie des chromosomes (ellipse bleue) pendant la mitose forment des PNB en télophase. En début d’interphase, les protéines des PNB sont progressivement recrutées dans le nucléole. B. Protéine nucléolaire B23 localisée par immunofluorescence en fin de télophase dans une cellule HeLa. Le Dapi (coloration spécifique de l’ADN) montre la compaction des chromosomes dans les noyaux des deux cellules filles. Les flèches indiquent les PNB contenant B23. C. Structure des PNB en microscopie électronique. On note la structure fibro-granulaire compacte de ces structures (© Photo J. Gebranne-Younès). Barre : 0,1 µm. |
 | Figure 2.
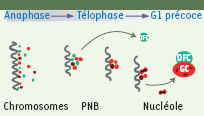
Trafic et interaction des protéines nucléolaires pendant l’assemblage du nucléole (d’après [
6]). |
La re-formation du nucléole, site de la biogenèse des ribosomes, a été choisi comme système modèle [
1,
2]. Nous cherchons à comprendre comment les machineries de maturation des ARN ribosomiques (ARNr) sont recrutées sur les sites de transcription des gènes ribosomiques. Pendant la mitose, les machineries de maturation nucléolaires sont localisées à la périphérie des chromosomes et donc à distance des gènes ribosomiques. On sait que le recrutement des protéines de maturation n’est pas direct mais fait intervenir une étape intermédiaire : la formation de corps appelés PNB (prenucleolar body) [
3–
5]. Dans un travail récent, nous avons comparé la dynamique de formation des PNB, le tri des protéines en fonction du temps et les interactions protéines-protéines dans les PNB [6]. Notre but est de comprendre le rôle des PNB au cours de l’assemblage du nucléole. |
Nous avons utilisé la vidéo microscopie pour comparer la dynamique des protéines avec des étiquettes fluorescentes vertes (GFP) ou rouge (DsRed). Nous avons comparé les concentrations des protéines de maturation précoces ou tardives dans les PNB. Enfin nous avons utilisé une méthode de FRET (fluorescence resonance energy transfer) fondé sur la décroissance de la durée de vie de la GFP (donneur) en présence de DsRed (accepteur) [
7]. Cette analyse non invasive, est réalisée pendant l’assemblage du nucléole et mesure la distance entre les deux étiquettes fluorescentes. La distance entre la GFP et la DsRed est inférieure à 7 nm s’il y a FRET ce qui correspond à la distance d’interaction des molécules. |
La dynamique et le tri des protéines Les protéines de maturation précoce (fibrillarine) et de maturation tardive (Bop1, B23 et Nop52) sont co-localisées en anaphase à la surface des chromosomes et forment plusieurs dizaines de PNB par noyaux en télophase. Nous avions montré préalablement en vidéomicroscopie [
8] que la présence de la fibrillarine dans les PNB était en moyenne de 15 minutes avant son recrutement dans le nucléole, tandis qu’elle était de 80 minutes pour Nop52. Nous en avions conclu qu’il y avait deux types de PNB correspondant respectivement à la maturation précoce et à la maturation tardive et à la cinétique de recrutement des protéines dans le nucléole. Dans ce travail, nous avons comparé dans la même cellule la dynamique des deux types de protéines. Nous avons co-exprimé la GFP-fibrillarine et la DsRed-B23, et suivi la localisation respective des deux protéines. Les images montrent la superposition des deux signaux dans les mêmes PNB. La quantification des deux signaux dans les PNB et en dehors des PNB indique : (1) que les deux protéines passent par les mêmes PNB ; (2) que la concentration dans les PNB est de 3 à 6 fois celle des protéines diffuses ; et (3) que la fibrillarine quitte plus vite les PNB que la protéine B23. Ainsi, dans les PNB, des protéines des machineries de maturation précoce et tardive sont co-localisées, mais leur temps de résidence n’est pas le même. Il y a donc un mécanisme de tri pour adresser ces protéines dans le nucléole. La même démarche a été suivie pour comparer la dynamique des protéines tardives entre elles. Nous avons constaté que les protéines Nop52 et B23 étaient ensemble dans les PNB et que leur concentration variaient simultanément dans les PNB et dans le nucléole. Cette observation peut s’expliquer par une interaction entre ces deux protéines avant leur recrutement dans le nucléole. Les interactions protéiques dans les PNB Pour mettre en évidence l’interaction des protéines Nop52 et B23, nous avons tout d’abord montré un FRET positif dans le nucléole. La durée de vie de la GFP-Nop52 est réduite en présence de DsRed-B23. En revanche, la durée de vie de la GFP-fibrillarine et de GFP-Bop1 n’est pas diminuée en présence de DsRed-B23. Il n’y a donc pas d’interaction détectable entre ces protéines. L’absence de FRET est attendue dans le cas du couple fibrillarine-B23 puisque les deux protéines ne sont pas dans le même compartiment nucléolaire (contrôle négatif). Par ailleurs, Bop1 et B23 sont co-localisés dans le même composant nucléolaire et participent à la même étape de maturation des ARNr. Cela est un bon contrôle pour montrer la spécificité de la détection de FRET. La mesure du FRET a été réalisée par cette approche entre GFP-Nop52 et DsRed-B23 pendant deux heures à partir de l’anaphase. Pendant cette période, les protéines forment des PNB puis les protéines sont recrutées dans le nucléole. L’enregistrement de la durée de vie de la GFP est réalisé sur l’ensemble du champ, ensuite les structures d’intérêts sont sélectionnées et le FRET est mesuré au cours du temps dans ces structures. À la périphérie des chromosomes en anaphase, le FRET n’a jamais été détecté. Le FRET est détecté dans 20 % des PNB quand les cellules entrent en télophase, mais dans 40 % en fin de télophase. Ce pourcentage est de 55 % dans les PNB en début de G1. Ces résultats indiquent que les interactions entre GFP-Nop52 et DsRed-B23 ont lieu dans les PNB avant leur recrutement dans le nucléole. |
Le recrutement des protéines de maturation dans le nucléole n’est pas direct en début d’interphase. Il y a, entre la périphérie des chromosomes et le nucléole une étape intermédiaire, étape correspondant à la formation de nombreuses petites structures : les PNB. En analysant la dynamique des protéines de maturation précoce et tardive, nous avons montré que ces deux classes de protéines passent par les mêmes PNB. Cette observation semble indiquer qu’il y aurait des sites de formation des PNB, puis un tri des protéines pour un recrutement précoce ou tardif dans le nucléole. Le mécanisme de tri n’est pas connu pour le moment. La mesure de FRET pendant l’assemblage du nucléole a permis de découvrir une interaction entre des protéines de maturation tardive au niveau des PNB. Ce signal de FRET augmente au cours du temps et suggère un rôle de plate-forme d’assemblage pour les PNB. Les nouvelles techniques d’observation et de mesure en cellules vivantes, et en temps réel [
9,
10], montrent leur potentiel pour comprendre des phénomènes complexes comme l’assemblage du nucléole. |
1. Strouboulis J, Wolffe AP. Functional compartmentalization of the nucleus. J Cell Sci 1996; 109 : 1991–2000. 2. Hernandez-Verdun D, Louvet E. Le nucléole : structure, fonctions et maladies associées. Med Sci (Paris) 2004; 20 : 37–44. 3. Ochs RL, Lischwe MA, Shen E, et al. Nucleologenesis : composition and fate of prenucleolar bodies. Chromosoma 1985; 92 : 330–6. 4. Dundr M, Misteli T, Olson MOJ. The dynamics of postmitotic reassembly of the nucleolus. J Cell Biol 2000; 150 : 433–46. 5. Hernandez-Verdun D, Roussel P, Gébranne-Younès J. Emerging concepts of nucleolar assembly. J Cell Sci 2002; 115 : 2265–70. 6. Angelier N, Tramier M, Louvet E, et al. Tracking the interactions of rRNA processing proteins during nucleolar assembly in living cells. Mol Biol Cell 2005; 16 : 2862–71. 7. Tramier M, Gautier I, Piolot T, et al. Picosecond-Hetero-FRET microscopy to probe protein-protein interactions in live cells. Biophys J 2002; 83 : 3570–7. 8. Savino TM, Gébrane-Younès J, De Mey J, et al. Nucleolar assembly of the rRNA processing machinery in living cells. J Cell Biol 2001; 153 : 1097–110. 9. Misteli T. Protein dynamics : implications for nuclear architecture and gene expression. Science 2001; 291 : 843–7. 10. Janicki SM, Spector DL. Nuclear choreography : interpretations from living cells. Curr Opin Cell Biol 2003; 15 : 149–57. |