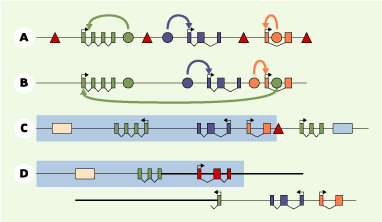
Un gène est généralement décrit comme un ensemble composé d’une unité transcrite et d’éléments régulateurs (promoteurs, enhancer et silencer) qui agissent de concert pour déterminer de manière précise l’expression du gène en temps, place et quantité voulus. Pour échapper à l’effet des enhancers ou silencers de ses voisins, un gène est flanqué d’éléments « isolants » qui définissent des frontières à l’action des enhancer/silencers (Figure 1A). Les gènes sont alors essentiellement exprimés de manière autonome même s’il existe des cas de chevauchement physique entre plusieurs gènes (Figure 1B). Ce modèle d’un génome structuré gène par gène vient d’être remis en cause par plusieurs études qui ont comparé de manière globale les profils d’expression des gènes en fonction de leur localisation génomique [ 1– 3]. Chez le nématode, la mouche et l’homme, les gènes adjacents ont tendance à avoir le « même » profil d’expression. Les régions de co-expression ainsi définies contiennent plusieurs gènes distincts (jusqu’à 20) et ne correspondent pas aux classiques complexes multigéniques produits par la duplication d’un gène ancestral (qui partagent souvent les séquences régulatrices héritées de leur ancêtre commun). Selon les auteurs de ces travaux, le génome serait donc composé de vastes domaines de co-expression comprenant plusieurs gènes (Figure 1C). Mais quels sont les mécanismes et les éventuelles fonctions associés à ces domaines d’expression globale ?
| Figure 1.
Organisation des gènes au sein du génome. A. Un génome structuré en gènes autonomes séparés par des éléments frontières (triangles rouges). B. Dans certains cas, gènes et éléments régulateurs sont physiquement enchevêtrés. Dans ce cas, la spécificité des enhancer (cercles) pour un gène est déterminée par des interactions préférentielles entre enhancer et promoteurs (rectangles). C. Le génome est composé de domaines de co-régulation (zone tramée bleu clair) définis par des éléments régulateurs qui imposent à tous les gènes situés dans ces domaines un profil d’expression commun (outre leur profil propre). Les limites de ces domaines peuvent être fixées par des éléments de type frontière. D. Un réarrangement chromosomique intervenant dans un domaine de régulation a des conséquences qui ne se limitent pas à la mutation du gène (vert) situé au point de cassure : perte d’expression pour les gènes qui sortent du domaine de co-régulation (bleu et orange), gain d’expression pour les gènes (rouge) qui se retrouvent maintenant dans la zone d’action de l’élément global. |
Les gènes HOX sont regroupés en complexes multigéniques, cette organisation étant liée à leur fonction d’ « architectes » du développement de l’embryon le long de son axe antéro-postérieur [ 4]. Au cours de l’évolution, les gènes situés dans la partie 5’ du complexe HoxD ont été recrutés pour participer à la formation des membres : ils sont notamment tous exprimés dans la partie distale qui formera les doigts. Plusieurs travaux de notre laboratoire ont montré que cette co-expression dépendait de l’action globale et non spécifique d’un enhancer commun. Nous avons récemment identifié cet enhancer et montré qu’il définissait en fait une région semblable aux domaines de co-expression décrits dans les expériences d’analyse globale d’expression génique [ 5]. Les deux gènes adjacents au complexe HoxD, Evx2 (à 8 kb de Hoxd13) et Lunapark (Lnp, à 90 kb), sont exprimés dans la partie distale des membres, avec un profil et une séquence d’activation semblables à ceux de Hoxd13. De plus, Evx2 et Lnp sont co-exprimés dans certaines régions du système nerveux. Une étude fonctionnelle et systématique par transgenèse des 700 kb autour de ce locus a identifié en amont de Lnp (à 250 kb de Hoxd13) une séquence capable d’activer un gène rapporteur dans les membres et le système nerveux, lieux d’expression de Hoxd13 ou Evx2 et Lnp. La perte d’expression d’Evx2 et de Hoxd13 dans le cas de la mutation Ulnaless - une inversion chromosomique qui sépare Evx2 et HoxD de cet enhancer - montre que celui-ci est effectivement impliqué dans l’expression de ces gènes. Cette global control region (GCR) définit un domaine de co-expression qui contient les gènes Lnp, Evx2 et Hoxd13 à Hoxd10, et tout gène inséré dans cette région tombe sous l’influence du GCR et adopte le profil d’expression correspondant [ 6].
L’introduction de gènes supplémentaires dans la zone d’action du GCR se traduit par une diminution du niveau d’expression des gènes les plus éloignés [ 7] : ce phénomène de compétition suggère qu’un domaine de co-expression ne se limite pas à une région de chromatine ouverte [ 2], favorisant passivement l’expression des gènes situés à l’intérieur, mais qu’il doit y avoir interaction entre les promoteurs des gènes et des éléments enhancer du GCR. Cependant, l’extrême conservation du GCR chez différents mammifères (> 95 % sur plusieurs kb) et plus généralement chez les vertébrés, va bien au-delà de ce que l’on s’attendrait à voir pour un enhancer classique [5]. Et certaines des régions les plus conservées ne semblent pas avoir d’effet activateur direct. Leur rôle pourrait donc être d’organiser (au niveau de la chromatine ? d’une localisation particulière dans le noyau ?) le locus autour du GCR pour permettre aux enhancer présents d’agir à distance sur plusieurs gènes.
Qu’en est-il de l’importance fonctionnelle de ces domaines ? Une vision extrême consisterait à y voir un « choix » évolutif permettant d’organiser le génome en de vastes opérons pour assurer la co-expression de protéines participant à la même fonction. Cependant, contrairement au gène Hoxd13, ni Lnp, ni Evx2 ne semblent avoir de fonction importante dans les membres. Alors pourquoi ces deux gènes ont-ils conservé cette expression au cours de l’évolution ? Pour activer Hoxd13 dans les membres, le GCR doit être capable d’agir sur l’intervalle entier ; il active donc de façon « ectopique » Lnp et Evx2, qui doivent rester proches du GCR, car celui-ci est nécessaire à leur expression dans le système nerveux où ces gènes sont importants. De telles expressions « ectopiques » n’auraient le plus souvent pas d’effet et les éléments frontières (insulator) ne seraient présents que dans les quelques cas où l’activation d’un gène par les éléments voisins serait trop délétère. Les régions de co-régulation seraient donc une conséquence indirecte de l’enchevêtrement inextricable entre les gènes et leurs éléments régulateurs dispersés : un seul gène serait fonctionnellement important et l’expression de ses voisins ne serait qu’une conséquence « passive » (le contrôle de l’expression des gènes ne serait donc pas nécessairement aussi strict que ce que l’on pourrait penser). Il est aussi possible que l’expression de Lnp et Evx2 ait une fonction dans les doigts, non pas via les protéines correspondantes, mais parce qu’elle titrerait l’effet enhancer du GCR sur le gène Hoxd13. Cet ajustement du niveau d’expression d’un gène précis pourrait expliquer le maintien des domaines de co-expression au cours de l’évolution ; à l’inverse, les éléments comme le GCR pourraient permettre à un gène d’acquérir « facilement » de nouveaux domaines d’expression où exercer potentiellement de nouvelles fonctions.
Le profil d’expression d’un gène va ainsi dépendre autant de sa fonction propre que de celle de ses voisins, du fait notamment de l’action globale sur de vastes domaines chromosomiques d’éléments de type GCR mais aussi de la simple présence de voisins susceptibles de moduler son niveau d’expression. Cette interdépendance entre gènes faisant partie d’un domaine de co-régulation doit nous amener à reconsidérer ce qu’est un gène, ou tout au moins la façon dont nous nous le représentons physiquement, et à absolument tenir compte du contexte génomique de manière plus vaste, que ce soit pour comprendre son mode de fonctionnement ou les effets d’une anomalie génétique (Figure 1D).