Le développement d’un arsenal croissant d’antibiotiques depuis la commercialisation de la pénicilline a donné l’illusion que les maladies infectieuses n’étaient plus une menace pour la santé publique. L’utilisation d’antibiotiques, et ce quelle que soit leur nature, est devenue tellement courante, voire abusive et synonyme d’efficacité à toute épreuve, que la notion d’organismes réfractaires à ces antibiotiques est désormais quasi inexistante.
Néanmoins, bien avant l’usage de la pénicilline en clinique, Abraham et Chain [1] avaient mis en évidence l’existence de souches de Staphylococcus aureus (staphylocoque doré) capables d’inactiver le pouvoir antimicrobien de cet antibiotique. Depuis, le pourcentage des bactéries résistantes à un ou plusieurs antibiotiques n’a cessé d’augmenter. C’est ainsi que 40-60 % des souches cliniques de S. aureus, l’agent responsable de nombreuses infections nosocomiales, sont actuellement résistantes à tous les antibiotiques à l’exclusion de la vancomycine [2], considéré comme l’antibiotique de dernier recours contre les infections causées par des bactéries à Gram positif. S. aureus est un pathogène remarquable, dont le pouvoir pathogène va d’un simple abcès à la septicémie, et dont certaines complications (endocardites, méningites ou ostéomyélites), parfois mortelles, sont souvent source de séquelles.
Malheureusement, les entérocoques, bactéries proches des staphylocoques, et elles aussi responsables de maladies nosocomiales surtout chez des patients immunodéprimés, ont acquis la capacité de résister à l’action bactéricide de la vancomycine [3]. Cette résistance à la vancomycine est transmissible via des éléments génétiques mobiles. In vitro, le transfert de ces déterminants génétiques de résistance des entérocoques vers S. aureus a été accompli avec succès [4] soulevant une inquiétude légitime quant à une possible transmission in vivo. Ce transfert in vivo a déjà été mis en évidence des entérocoques aux streptocoques du groupe B [5], et un transfert identique à S. aureus serait catastrophique d’un point de vue clinique.
La vancomycine est le chef de file de la classe des antibiotiques glycopeptidiques. Les glycopeptides, comme les ß-lactamines, inhibent la synthèse du peptidoglycane des bactéries. Le peptidoglycane est un hétéropolymère présent exclusivement chez les bactéries, comparable à une cotte de maille entourant la bactérie. Ainsi, le peptidoglycane contribue-t-il au maintien de l’intégrité cellulaire et, en son absence, il y aurait une lyse des bactéries entraînant la mort cellulaire. Il est donc tout à fait justifié de privilégier la voie de synthèse de ce peptidoglycane comme cible thérapeutique.
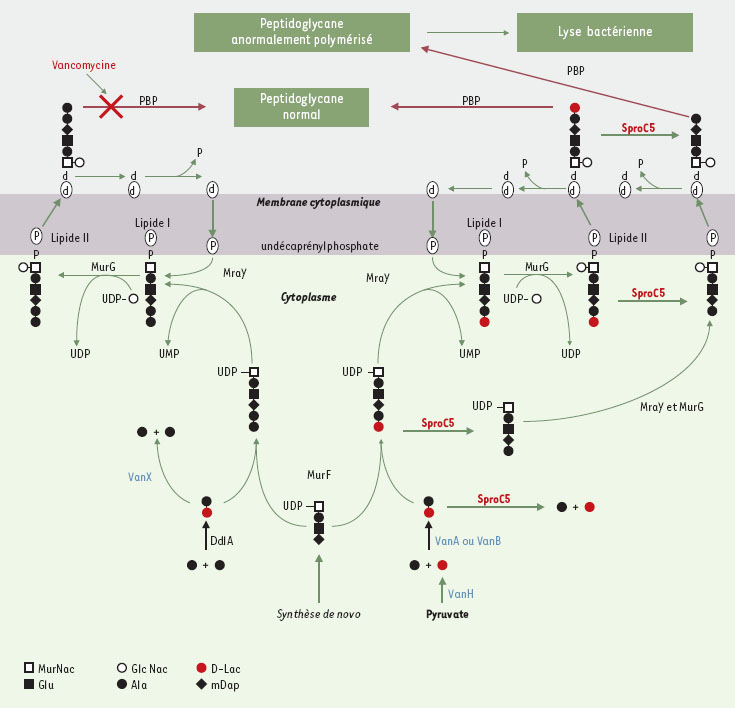
La synthèse du peptidoglycane fait intervenir trois étapes majeures (Figure 1) : une étape cytoplasmique de synthèse du précurseur UDP-acide-N-acétylmuramique-(MurNAc)-pentapeptide ; puis une étape de translocation des précurseurs à travers la membrane cytoplasmique; enfin, une dernière étape de polymérisation du peptidoglycane à partir de ces précurseurs lipidiques. La grande majorité des bactéries synthétisent des précurseurs qui ont une terminaison carboxylique D-Ala-D-Ala, et c’est cette terminaison qui est reconnue par la vancomycine. La vancomycine, en se liant très fortement à la terminaison D-Ala-D-Ala, bloque les précurseurs au stade lipidique [6], et inhibe par voie de conséquence la synthèse du peptidoglycane, entraînant inévitablement la mort cellulaire.
| Figure 1 Voie de synthèse du peptidoglycane. Des souches sensibles à la vancomycine synthétisent exclusivement des précurseurs se terminant par le motif D-Ala-D-Ala. La vancomycine inhibe la synthèse du peptidoglycane, en se fixant fortement sur l’extrémité des précurseurs. Dans des souches résistantes, l’extrémité normale (D-Ala-D-Lac) est remplacée par une extrémité modifiée D-Ala-D-Lac (précurseurs représentés par des boules rouges). Pour ce remplacement, les protéines VanA (ou VanB), VanH et VanX sont essentielles (en bleu). Les entérocoques n’ont pas d’activité pyruvate déshydrogénase endogène, d’où le caractère essentiel de VanH. VanA (ou VanB) catalyse la formation de la liaison ester D-Ala-D-Lac que la protéine endogène DdlA est incapable de catalyser. Finalement, VanX code pour une D-Ala-D-Ala dipeptidase essentielle pour enrichir la bactérie en précurseurs D-Ala-D-Lac. Notre stratégie est analogue à celle que la bactérie utilise pour devenir résistante. SProC5 peut agir à différentes étapes de la voie métabolique alternative. En hydrolysant le depsipeptide D-Ala-D-Lac, SProC5 agit de façon analogue à VanX. La diminution de la concentration de D-Ala-D-Lac nous paraît le moyen le plus efficace pour contrer le mécanisme de résistance puisqu’il touche directement au rapport précurseurs normaux/ précurseurs modifiés. Néanmoins, cela suppose que SProC5 diffuse librement à travers la membrane cyto-plasmique. SProC5 peut également hydrolyser la terminaison D-Ala-D-Lac une fois incorporé dans la structure pentapeptidique entraînant l’accumulation de précurseurs tétrapeptidiques. Ces précurseurs peuvent être utilisés exclusivement en tant que donneurs lors de la polymérisation du peptidoglycane par les PBP (protéines liant la pénicilline ou penicillin-binding proteins). Ceci entraînera inévitablement une lyse cellulaire par manque de précurseurs accepteurs : seuls des précurseurs pentapeptidiques peuvent être utilisés comme accepteurs. |
La grande majorité (95%) des entérocoques résistants à la vancomycine isolés en clinique sont porteurs des déterminants génétiques vanA ou vanB [3]. Ces déterminants sont souvent associés à des éléments génétiques mobiles (transposons) présents sur des plasmides conjugables. Les souches vanA ou vanB se distinguent des autres par une terminaison carboxylique des précurseurs de type D-Ala-D-Lac au lieu de D-Ala-D-Ala. Le peptidoglycane synthétisé par ces bactéries résistantes a une structure identique à celle des souches sensibles. En revanche, l’affinité de la vancomycine pour cette extrémité altérée D-Ala-D-Lac est diminuée d’un facteur 1000 [7]. Le niveau de résistance des souches est directement proportionnel au pourcentage de précurseurs possédant une extrémité D-Ala-D-Lac [8].
De nombreux efforts ont été entrepris pour trouver de nouveaux antimicrobiens qui pourraient contrer ce mécanisme de résistance à la vancomy-cine [9–11], soit par modification de la vancomycine pour augmenter son affinité pour la terminaison D-Ala-D-Lac, soit via l’inhibition de la voie de synthèse de D-Ala-D-Lac (Figure 1). Ces stratégies ont un désavantage majeur. Commel’expérience clinique l’a maintes fois démontré, les bactéries sont très versatiles et aptes à trouver de nouveaux mécanismes de résistance. Le développement de nouveaux antibiotiques dans le sens traditionnel du terme (une cible thérapeutique - un antibiotique) résultera donc à moyen ou à long terme en l’apparition de souches résistantes. Nous avons donc envisagé une stratégie différente, en cherchant des molécules qui seraient capables d’hydrolyser laliaison ester D-Ala-D-Lac, dans l’espoir de pouvoir transformer des bactéries résistantes en bactéries à nouveau sensibles à la vancomycine [12].
Pour identifier des molécules capables de cliver la liaison D-Ala-D-Lac, nous avons synthétisé un analogue du D-Ala-D-Lac marqué avec un chromogène (Disperse Red Dye 1). Cette sonde a été utilisée pour cribler des bibliothèques combinatoires aléatoires (un total de 300 000 tripeptides/tétra-peptides différents) couplées à des billes. Dans des solvants polaires, les molécules capables de cliver la liaison ester restaient liées de façon covalente à la bille la rendant rouge. Les billes rouges ont toutes été recueillies et la séquence peptidique analysée après photolyse par chromatographie gazeuse. Il s’est averé que toutes les séquences peptidiques actives avaient une sérine en position amino-terminale. Une bibliothèque tripeptidique de 50 000 composés a permis d’identifier trois séquences actives : X-L-Lys-L-Ser diméthylurée (avec X variable), X-D-Lys-D-Ser diméthylurée et L-Lys-D-Pro-L-Ser diméthylurée. Toutes les séquences avaient un groupe nucléophile (Ser amino-terminal), un électrophile (Lys, Cu2+, NH) et un donneur d’électrons (diméthylurée) pour activer le nucléophile (groupe hydroxyle de la sérine). Tous ces éléments avaient besoin d’une organisation, d’une orientation optimale et d’une certaine rigidité conformationelle pour permettre l’attaque nucléophile de la sérine, ce qui était suggéré par la prévalence de la proline dans les molécules actives, puis confirmé par des études de modélisation de structure.
La molécule BnNH-L-Lys-D-Pro-L-Ser diméthylurée (peptide 4αc) a été choisie pour confirmer nos présomptions fondées sur les informations structurales des molécules actives, en étudiant son activité d’hydrolyse du D-Ala-D-Lac in vitro. Le peptide 4(X a été comparé à des structures proches (peptide 4(X avec le groupe hydroxyle de la sérine bloqué par un groupement butyle, peptide 7) ou ses sous-unités (BnNH-D-Pro-L-Ser diméthylurée, peptide 6, et N-acétyl-BnNH-L-Lys, peptide 8). Dans des conditions physiologiques, le peptide 40C avait une activité modeste (hydrolyse de 20 % de D-Ala-D-Lac en 24 heures) mais très spécifique puisque aucune des molécules témoins 6, 7, 8 et 6+8 n’a montré une activité significative comparée au tampon. Ces résultats confirmaient la nécessité d’une structure complète et bien assemblée puisque les deux sous-unités ajoutées simultanément (6+8) n’avaient aucune activité. L’énantiomère1 du peptide 4(α (BnNH-D-Lys-L-Pro-D-Ser diméthylurée) était deux fois moins actif, suggérant qu’une complémentarité structurale (définie par une chiralité des molécules) entre la molécule active et la terminaison D-Ala-D-Lac est nécessaire pour une activité optimale. Finalement, pour prouver que l’hydrolyse de la liaison ester D-Ala-D-Lac par le peptide 4(α se produit en réponse à une attaque nucléophile du groupe hydroxyle de la sérine, nous avons purifié l’intermédiaire réactionnel par chromatographie liquide à haute pression (HPLC) et identifié sa structure par spectroscopie de masse et résonance magnétique nucléaire.
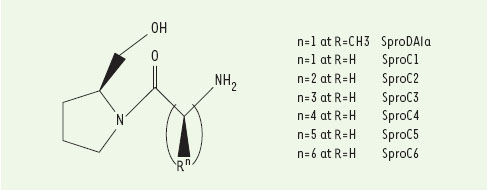
Ayant établi les caractéristiques structurales nécessaires pour avoir une molécule active et après avoir démontré le mécanisme réactionnel essentiel à leur activité, nous avons recherché des molécules non peptidiques qui pourraient incorporer ces mêmes propriétés. Nous nous sommes intéressés surtout à des dérivés prolinol N-acétylés puisque ce sont des molécules intrinsèquement chirales, leur alcool primaire est un bon donneur d’électron via des liaisons hydrogène intramoléculaires, et elles permettent le couplage d’un groupe électrophile (NH) via différents linkers (Figure 2). Nous avons trouvé que la molécule SProC5 était la plus active (deux fois plus efficace que le peptide 4(α: hydrolyse de 50 % du D-Ala-D-Lac en 24 heures). Sa plus grande activité est due à une nature plus nucléophile du groupe hydroxyle de SProC5, confirmée par des études de RMN et de modélisation structurale.
La nature peptidique de la molécule 4(α était un handicap pour des études d’activité biologique. Au contraire, SProC5 étant non peptidique, nous avons pu tester son activité contre une souche d’Enterococcus faecium très résistante à la vancomycine. Cette souche EF228 est vanA positive et a une CMI (concentration minimaleinhibitrice) pour la vancomycine de 1000 µg/ml. En combinant la vancomycine avec 50 mM de SProC5, nous pouvions inhiber la croissance de la souche avec 62,5 µg/ml de vancomycine. L’activité bactéricide de la combinaison vancomycine-SProC5 a été confirmée en dénombrant le nombre de colonies viables. En combinant 62,5 µg/ml de vancomycine et 50 mM de SProC5, nous avions diminué de 3 ordres de magnitude la viabilité celluaire par rapport à la vancomycine (250 µg/ml) ou SProC5 (50 mM) utilisés seuls. Cet effet a été encore plus prononcé en combinant 250 µg/ml de vancomycine et 50 mM de SProC5 avec une diminution de la viabilité de 4 ordres de magnitude. La synergie entre la vancomycine et SProC5 est dose-dépendante et, seul SProC5, est incapable d’inhiber la souche EF228.
L’activité biologique de SProC5 aurait pu être un fait du hasard. Pour corréler son activité biologique avec son activité d’hydrolyse du D-Ala-D-Lac, nous avons comparé l’activité de SProC5 soit à celle d’un autre dérivé prolinol Nacétylé SProC2 moins efficace in vitro, soit à celle de molécules témoins (RProC5, son énantiomère, et le linker C5, sous-unité de SProC5).
Effectivement, SProC2 en combinaison avec la vancomycine était moins efficace que SProC5. De plus, les molécules témoins n’avaient aucune activité synergique avec la vancomycine, suggérant que c’est bien sa capacité d’hydroyser la liaison D-Ala-D-Lac qui explique l’efficacité de SProC5. Finalement, nous avons testé toutes ces molécules en présence d’une souche bactérienne sensible à la vancomycine, et dont les précurseurs du peptidoglycane ont une extrémité D-Ala-D-Ala. Cette souche s’est avérée complètement insensible à leur combinaison avec la vancomycine, indiquant la spécificité d’action de SProC5 vis-à-vis de souches résistantes synthétisant des précurseurs avec une extrémité D-Ala-D-Lac.
Le clivage sélectif de la liaison D-Ala-D-Lac présente exclusivement dans des souches résistantes représente une nouvelle stratégie thérapeutique. Celle-ci a l’avantage de réduire les moyens de pression sélective - la vancomycine reste le seul vrai antibiotique - l’utilisation de SProC5 n’engendre pas une pression sélective puisque les souches sensibles sont indifférentes à sa présence. Dans les souches résistantes, SProC5 agit sur un substrat contrairement à d’autres antibiotiques qui inhibent une activité biochimique, d’où un risque diminué de développement de résistance. Des études plus poussées sur des ana-ogues de SProC5 plus actifs in vitro et contre des souches résistantes, permettraient d’envisager des études dans des modèles animaux, et, éventuellement, de valider cette nouvelle approche thérapeutique.