| |
| Med Sci (Paris). 2003 June; 19(6-7): 725–734. Published online 2003 June 15. doi: 10.1051/medsci/20031967725.
C. elegans : des neurones et des gènes Christelle Gally and Jean-Louis Bessereau* Biologie Cellulaire de la Synapse, Inserm U.497, École Normale Supérieure, 46, rue d’Ulm, 75230 Paris Cedex 05, France |
Le cerveau humain est un organe complexe qui contient 100 milliards de neurones et probablement mille fois plus de synapses. Pour identifier les mécanismes biologiques qui sous-tendent l’activité d’un tel système, les disciplines qui se sont individualisées au sein des neurosciences portent leur analyse sur des objets de différentes complexités, de la genèse du langage au fonctionnement des canaux ioniques. Relier ces niveaux de complexité reste néanmoins particulièrement difficile. Une approche complémentaire repose sur l’utilisation d’organismes simples et manipulables qui permettent de passer rapidement de la molécule au comportement. À la fin des années 1960, S. Brenner introduisit le nématode Caenorhabditis elegans comme système modèle dans le but d’identifier par une approche génétique les règles de fonctionnement du système nerveux [
1]. Les résultats obtenus depuis le temps des pionniers montrent que le ver peut être utilisé de façon pertinente pour identifier de nouveaux gènes ou de nouvelles fonctions qui sont conservés chez les vertébrés dans des processus neurobiologiques, physiologiques ou pathologiques. |
Pourquoi étudier le système nerveux d’un ver ?
C. elegans est un nématode non segmenté mesurant 1 mm à l’âge adulte, vivant dans la terre à l’état naturel. Plusieurs caractéristiques de sa physiologie en font un organisme particulièrement adapté à la neurobiologie: (1) il possède un système nerveux simple, constitué de 302 neurones dont le nombre et la position sont constants d’un individu à l’autre (Figure 1). Les neurones de C. elegans ont une morphologie généralement simple, avec un ou deux prolongements neuritiques. À partir de coupes sériées examinées en microscopie électronique, J. White et al. ont reconstitué l’ensemble de la connectivité du réseau neuronal [
2]. Ce réseau est très reproductible d’un individu à l’autre bien qu’il puisse exister des variations interindividuelles du nombre des connexions formées entre les partenaires neuronaux. Le système nerveux de C. elegans contient environ 7000synapses chimiques, dont 2000 jonctions neuromusculaires et 600 jonctions communicantes (gap junctions). A titre de comparaison, une seule cellule pyramidale du cortex de mammifère forme plus de 10000 synapses; (2) C. elegans est transparent durant la totalité de son développement. Chaque neurone peut être identifié individuellement en microscopie optique d’après la position de son noyau dans l’animal. Avec un laser, il est ensuite possible de procéder à la photo-ablation d’un neurone ou d’un groupe de neurones pour évaluer leur fonction dans un comportement précis; (3) de profondes altérations du système nerveux ne sont généralement pas incompatibles avec le développement de C. elegans en conditions de laboratoire. Pour se nourrir, C. elegans aspire les bactéries présentes dans le milieu extérieur grâce à une pompe musculaire, le pharynx. Celuici est doué d’une activité myogénique intrinsèque et fonctionne en absence d’innervation. Pour se reproduire, C. elegans existe sous deux formes sexuées mâle et hermaphrodite. L’hermaphrodite peut s’autoféconder, ce qui permet à des animaux de donner une descendance même s’ils sont incapables de se déplacer et de s’accoupler. Ainsi, sur les 302 neurones présents chez C. elegans, seuls 3 sont absolument nécessaires à la survie de l’individu en condition de laboratoire: le neurone M4 qui permet la relaxation du pharynx, et la paire de neurones CANL et CANR qui contrôlent l’osmolarité du milieu intérieur. De ce fait, des mutations affectant profondément le développement ou le fonctionnement du système nerveux de C. elegans sont viables et analysables chez l’adulte.
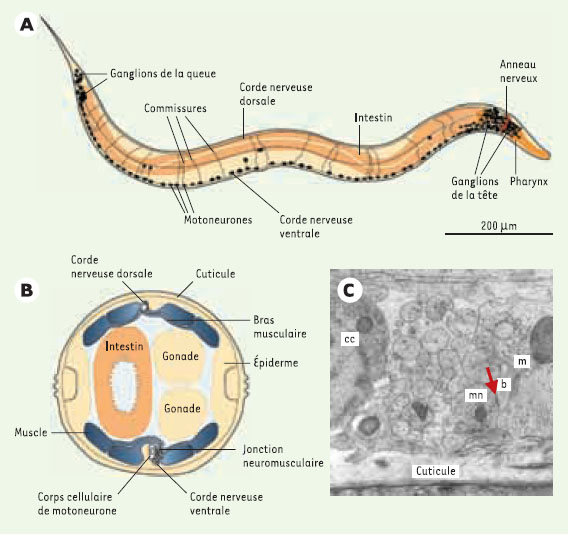
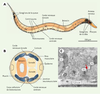 | Figure 1.
Le système nerveux de C. elegans adulte. A. Les 302 neurones du nématode se répartissent pour la plupart entre les ganglions de la tête, de la queue et le long de la corde nerveuse ventrale. Tous les corps cellulaires (cc) des motoneurones ne sont pas représentés sur ce schéma. Un faisceau de neurites majoritairement issues des neurones contenus dans les ganglions de la tête constitue « l’anneau nerveux » qui entoure le pharynx. Les motoneurones sont situés dans la région ventrale de l’animal. Ils possèdent des extensions neuritiques ventrales qui circulent selon un axe antéro-postérieur dans la corde ventrale. Certains motoneurones envoient des projections dorsales via des commissures circonférentielles. Les cellules musculaires qui permettent le déplacement du ver sont réparties en quatre quadrants ventraux et dorsaux. B. Coupe transversale schématique d’un hermaphrodite adulte. Les cellules musculaires envoient des extensions cytoplasmiques, les « bras musculaires », à destination des axones moteurs contenus dans les cordes ventrale ou dorsale. C. Une coupe transversale de la corde ventrale observée en microscopie électronique permet de visualiser une jonction neuromusculaire (flèche) entre un motoneurone (mn) et un bras musculaire (b). cc: corps cellulaire; m: cellule musculaire (photographie réalisée par P. Rostaing, Inserm U.497). |
Le séquençage du génome de C. elegans en 1998 a confirmé le haut degré de conservation qui existe à l’échelle moléculaire entre le système nerveux d’un nématode et celui d’un vertébré [
3,
4]. Hormis le canal sodium sensible au potentiel, l’ensemble des familles de récepteurs et de canaux ioniques est détecté dans le génome de C. elegans. Pour de multiples gènes impliqués dans la neurotransmission tels que unc-25 qui code pour la glutamic acid decarboxylase (GAD, enzyme de biosynthèse du GABA), le génome de C. elegans ne contient qu’un seul gène alors qu’il existe plusieurs paralogues chez les mammifères [
5]. Cette caractéristique permet de s’affranchir des problèmes de redondance et facilite l’analyse fonctionnelle des mutants. À l’opposé, quelques familles géniques semblent avoir été amplifiées durant l’évolution. Par exemple, le génome de C. elegans contient probablement une quarantaine de gènes codant des sous-unités de récepteurs nicotiniques de l’acétylcholine alors que les mammifères n’en possèdent que 16! La raison d’une telle diversité de récepteurs au regard du faible nombre de neurones n’est pas comprise à l’heure actuelle. |
Analyse de la croissance axonale : apport des cribles visuels Dès que les neurones commencent à se différencier, des expansions neuritiques se développent et naviguent sur de longues distances pour atteindre les cibles à innerver. La simplicité et la reproductibilité du réseau neuronal alliées à la transparence de l’organisme font de C. elegans un système particulièrement adapté à l’analyse des mécanismes de la croissance axonale. Même si la tâche est d’une complexité très supérieure chez le mammifère par rapport au nématode, les composants élémentaires utilisés dans chacun des systèmes sont très conservés. Les premiers mutants de croissance axonale ont été identifiés par S. Brenner en 1974 sur la base d’anomalies de la locomotion (mutants Unc: uncoordinated) [1]. Par exemple, chez les mutants unc-5, unc-6 et unc-40, les migrations selon l’axe dorso-ventral sont anormales [
6]. UNC-6 est une protéine de la famille des laminines qui est secrétée par des cellules ventrales. Le gradient de concentration ventro-dorsal est interprété via les récepteurs transmembranaires UNC-40 [
7] et UNC-5 [
8]. Les axones exprimant UNC-40 sont attirés ventralement alors que les axones exprimant UNC-5, seul ou en combinaison avec UNC-40, sont repoussés en direction dorsale. Dans le laboratoire de M. Teissier- Lavigne, les nétrines, qui sont les homologues de UNC-6, ont été identifiées chez le poulet par une approche biochimique [
9]. DCC (deleted in colorectal cancer), l’orthologue de unc-40, était connu mais sa fonction dans le guidage axonal n’a été mise en évidence qu’après avoir démontré qu’il constituait un récepteur de la nétrine, sur la base des données génétiques obtenues chez C. elegans [
10]. Les homologues de unc-5 ont été clonés chez le vertébré sur la base de similarités de séquence. Le système de signalisation par la nétrine s’est avéré particulièrement important chez le vertébré pour l’établissement des croisements de la ligne médiane par les axones commissuraux. Si la perte de fonction de certains gènes est responsable de déficits massifs de la formation du câblage axonal qui s’accompagnent de phénotypes comportementaux facilement identifiables, d’autres gènes contribuent de façon plus fine à la mise en place de la connectivité axonale et leur mutation peut être phénotypiquement silencieuse. D’autres stratégies sont utilisées pour identifier de tels gènes. L’existence d’interactions génétiques entre les locus impliqués dans le même processus biologique peut être mise à profit. Par exemple, le groupe de J. Culotti a imaginé un crible de suppression pour identifier de nouveaux gènes impliqués dans les processus de migration dépendant de la nétrine. Le récepteur UNC-5 a été exprimé dans les neurones mécanosensoriels qui envoient normalement des axones latéraux ou ventraux. L’expression ectopique de UNC-5 dans ces neurones redirige leurs axones vers la corde dorsale. Un crible a été conduit pour identifier des mutants supprimant le phénotype lié à l’expression ectopique de unc-5 [
11]. Cette approche a permis d’isoler un nouveau gène, unc-129, qui code pour une protéine de la famille du transforming growth factor β (TGF β) [
12]. unc-129 est exprimé dans les muscles dorsaux et détermine probablement un gradient de concentration dorso-ventral qui se superpose au gradient ventro-dorsal de nétrine. Au cours du développement, les axones qui migrent dorsalement sont donc à la fois repoussés par la nétrine UNC-6 et attirés par UNC-129 (Figure 2).
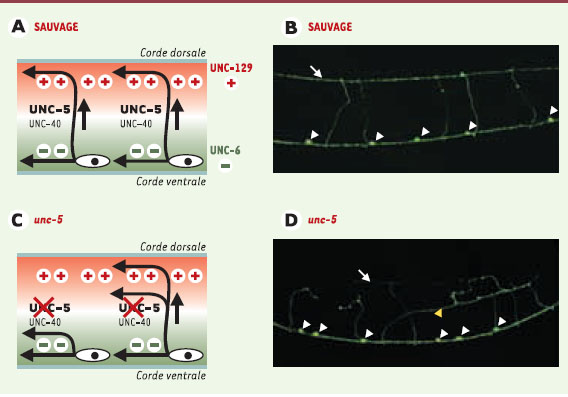
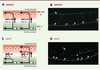 | Figure 2.
Le gène unc-5 est impliqué dans la croissance axonale ventro-dorsale. Plusieurs systèmes de signalisation agissent en parallèle dans les processus de croissance axonale circonférentielle à destination dorsale. Premièrement, la nétrine UNC-6, une protéine de la famille des laminines, est sécrétée par des cellules ventrales. La nétrine repousse en direction dorsale les neurites qui expriment le récepteur transmembranaire UNC-5, en interaction ou non avec le récepteur NUC-40. Deuxièmement, la protéine UNC-129 de la famille des transforming growth factors β est exprimée par les muscles dorsaux et joue un rôle attracteur sur les cônes de croissance qui migrent de la corde ventrale en direction de la corde dorsale (A). L’expression de la GFP (green fluorescent protein) sous le contrôle d’un promoteur spécifique des neurones GABA permet de visualiser les motoneurones GABAergiques chez l’animal vivant (B et D, pointes de flèches blanches) ainsi que leurs expansions neuritiques. En contexte sauvage, les axones des motoneurones dorsaux rejoignent la corde dorsale (flèche blanche) via des commissures circonférentielles. En contexte mutant unc-5, la nétrine perd son action répulsive (C) et les axones circulent en position ectopique latérale (D, pointe de flèche jaune). |
La transparence de C. elegans permet également de visualiser in vivo les processus neuritiques en exprimant la green fluorescent protein (GFP) dans des neurones à partir d’un transgène [
13] (Figure 2). Après mutagenèse, il est possible d’isoler des mutants chez lesquels la morphologie des axones est anormale et de cloner le gène muté, indépendamment de tout phénotype comportemental. Cette stratégie a récemment permis d’identifier le gène max-1 (motoneuron axon guidance) et son homologue humain hMAX-1. Malgré des anomalies de migration dorsale qui affectent 20 % des motoneurones, le phénotype fonctionnel ne se traduit que par des anomalies discrètes du déplacement vers l’arrière [
14]. Ce gène pourrait moduler la voie de signalisation qui dépend du récepteur UNC-5. À côté de ces approches de génétique « directe », il est possible d’invalider un gène d’intérêt pour évaluer sa fonction dans la mise en place du réseau axonal. Cette stratégie a été utilisée pour analyser la fonction de slt- 1, l’homologue du gène de drosophile slit [
15]. Le gène slit code pour le ligand des récepteurs robo (roundabout) dont l’expression permet aux cônes de croissance de sélectionner un trajet longitudinal spécifique plus ou moins éloigné de la source de la protéine Slit exprimée par les cellules de la ligne médiane. Contrairement à la drosophile et aux vertébrés, C. elegans ne contient qu’un seul gène de la famille robo appelé sax-3 (sensory axon guidance). SLT-1 est fortement exprimé par les muscles dorsaux et repousse les axones qui expriment SAX-3. Ce gradient répulsif dorso-ventral est utilisé parallèlement au gradient attractif de nétrine lu par les neurones exprimant le récepteur UNC-40. SLT-1 contrôle également des processus de migration antéro-postérieure et de croisement de la ligne médiane. De façon inattendue, les mutants sax-3 ont des défauts de migration plus importants que les mutants slt-1, suggérant l’existence d’un deuxième ligand pour sax-3/robo ou le fonctionnement de SAX-3/robo indépendamment de son ligand. La mise en place du réseau neuronal repose donc sur l’existence de gradients morphogènes qui sont lus différentiellement par les cônes de croissance en fonction des répertoires de récepteurs exprimés par chaque neurone. Pour traduire les signaux positionnels en modifications directionnelles, un groupe de gènes contrôle la dynamique du cytosquelette au niveau du cône de croissance. Enfin, des protéines d’adhérence permettent la formation et le maintien des faisceaux neuronaux. La mutation de ces gènes peut se traduire chez l’animal adulte par des anomalies morphologiques du réseau (pour revue, voir [
16,
17]). Il est possible de filmer le comportement des cônes de croissance au cours du développement [
18]. Ces techniques de vidéomicroscopie in vivo, alliées à la génétique et à la biochimie, devraient permettre de mieux comprendre les mécanismes de la croissance neuritique. |
Visualiser la synapse in vivo : l’accès à la génétique d’une structure subcellulaire Quand l’axone a atteint la cellule qu’il doit innerver, les synapses se différencient pour connecter le neurone et sa cible. Le petit nombre de synapses et la reproductibilité interindividuelle de la connectivité permettent d’analyser la synaptogenèse chez C. elegans avec la résolution d’une synapse unique. Ces caractéristiques ont permis d’identifier certains des mécanismes du contrôle génétique de la synaptogenèse et d’isoler de nouveaux gènes participant à la construction des appareils pré- et post-synaptiques. Une modification de la synaptogenèse peut se traduire, au niveau de l’animal entier, par des anomalies comportementales, en particulier locomotrices. Cette stratégie a permis d’identifier les mutants unc-4 : les vers peuvent se mouvoir vers l’avant mais ne peuvent reculer. Chez C. elegans, le système moteur est subdivisé en deux modules « marche avant » et « marche arrière » (Figure 3). Chaque module est constitué de « neurones de commande » qui innervent une classe spécifique de motoneurones permettant un mouvement soit vers l’avant, soit vers l’arrière. Grâce à la microscopie électronique, il a été montré que chez les mutants unc-4, les motoneurones VA normalement impliqués dans la « marche arrière » ont une structure normale mais sont innervés par les interneurones qui commandent la « marche avant » [
19]. unc-4 code pour un facteur de transcription à homéodomaine. Il est exprimé dans les motoneurones VA où il réprime l’expression de gènes propres aux neurones de la « marche avant » VB [
20,
21].
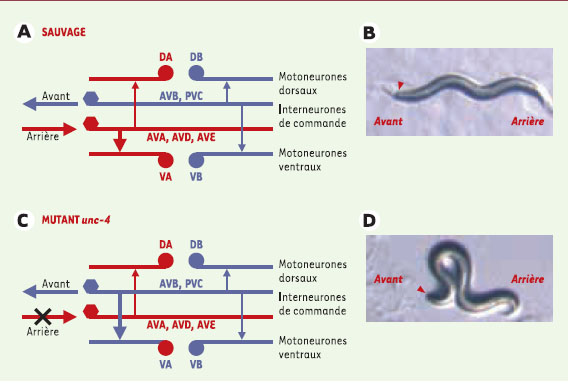
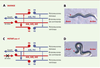 | Figure 3.
Le déficit locomoteur du mutant unc-4 est lié à des anomalies de la connectivité du circuit moteur. A. Chez C. elegans, le système moteur est subdivisé en deux modules: les interneurones de commande AVB et PVC ont des projections sur les motoneurones cholinergiques de classe B qui permettent le déplacement vers l’avant alors que les interneurones AVA, AVD et AVE ont des projections sur les motoneurones de classe A qui sont nécessaires à la marche vers l’arrière. Les motoneurones dits « ventraux » innervent les muscles ventraux et sont notés V, les motoneurones dits « dorsaux » innervent les muscles dorsaux et sont notés D. B. En réponse à une stimulation mécanique au niveau la tête, un individu sauvage recule avec un mouvement sinusoïdal. C. L’identité des motoneurones ventraux de classe A dépend de l’expression du facteur de transcription UNC-4. Chez un mutant unc-4, les motoneurones VA impliqués dans la marche arrière acquièrent l’identité des neurones VB et reçoivent leur innervation des interneurones de commande de la marche vers l’avant. D. Les animaux mutants unc-4 ne peuvent plus reculer et lors d’une stimulation au niveau de la tête (indiquée par la pointe de flèche), l’animal stoppe et s’enroule sur lui-même. |
Lorsque unc-4 est inactivé, les neurones de la « marche arrière » expriment un répertoire de gènes normalement réservé aux motoneurones de la « marche avant ». Il est probable que l’expression aberrante d’une ou de plusieurs molécules de surface induise la formation de connections inappropriées avec les interneurones de commande de la marche avant. Ces signaux restent, pour l’heure, à caractériser. Plus récemment, la visualisation de synapses individuelles in vivo a permis d’identifier de nouveaux gènes directement impliqués dans la construction des synapses. Les expérimentateurs ont fusionné la GFP à la synaptobrévine SNB-1, une protéine intrinsèque des vésicules synaptiques. Ce marqueur permet de visualiser sous la forme d’un point fluorescent l’agrégat de vésicules synaptiques formé au niveau de chaque varicosité (Figure 4) [
22]. En identifiant des mutants chez lesquels la distribution de la fluorescence est anormale, il a été possible d’identifier plusieurs gènes impliqués dans l’organisation des zones présynaptiques (pour revue, voir [
23]). Ces gènes sont exprimés dans l’ensemble du système nerveux et leurs mutations affectent la structure des synapses de façon plus ou moins profonde en fonction des neurones considérés. Il est à noter que malgré ces altérations morphologiques, les mutants identifiés ont un comportement normal ou proche de la normale. Ces cribles visuels ne sont pas saturés et devraient permettre l’identification de nouveaux constituants de la synapse. En utilisant des variants de couleur de la GFP, il est possible de visualiser chez un même animal les zones pré- et post-synaptiques (Figure 4). Ces nouveaux outils sont utilisés pour isoler des gènes impliqués dans la reconnaissance entre les partenaires synaptiques et la formation des domaines postsynaptiques [
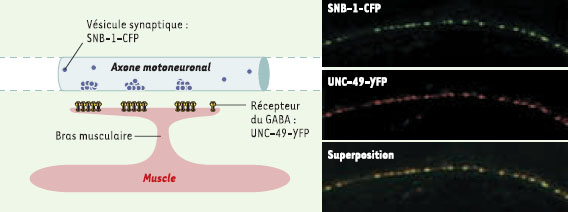
24].  | Figure 4.
Visualisation des jonctions neuromusculaires GABAergiques chez un animal vivant. Les synapses peuvent être observées in vivo de manière individuelle grâce à des protéines de fusion fluorescentes. En utilisant des variants de la GFP (green fluorescent protein), il nous a été possible de visualiser chez le même individu les différenciations pré- et postsynaptiques des jonctions neuromusculaires GABAergiques. La CFP (cyan fluorescent protein) a été fusionnée à SNB-1, une protéine intrinsèque des vésicules synaptiques, et exprimée dans les motoneurones GABAergiques. Au niveau de chaque varicosité synaptique GABAergique, les vésicules forment un agrégat visualisable sous la forme d’un point fluorescent (canal vert). Dans le muscle, une fusion entre le récepteur du GABA (UNC-49) et la YFP (yellow fluorescent protein) permet de visualiser les zones postsynaptiques (canal rouge). La superposition des deux images montre que chaque agrégat de récepteurs fait face à une différenciation présynaptique GABAergique. |
|
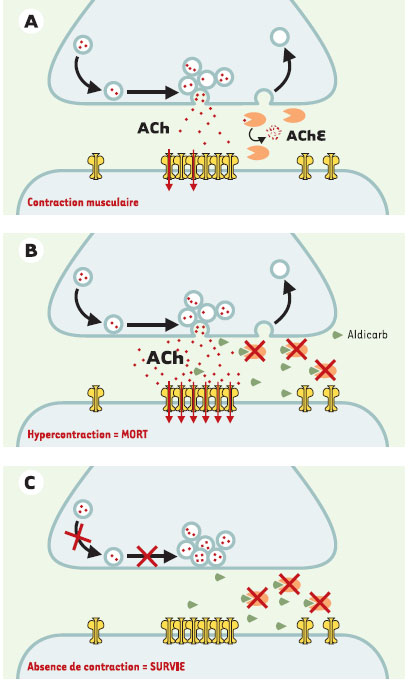
Quand les gaz de combat démasquent les dysfonctionnements de la transmission synaptique du nématode Une fois différenciée, la synapse chimique va permettre le transfert de l’information entre deux cellules excitables. Au niveau présynaptique, la dépolarisation de la membrane axonale provoque l’ouverture de canaux calciques sensibles au potentiel. L’influx local de calcium provoque la fusion des vésicules synaptiques avec la membrane plasmique au niveau de la zone active et la libération du neurotransmetteur dans la fente synaptique. Par diffusion, le neurotransmetteur atteint la membrane de la cellule post-synaptique où il active des récepteurs spécifiques. Les vésicules présynaptiques sont recyclées localement par endocytose et rechargées en neurotransmetteur (Figure 5A). De nombreuses protéines impliquées dans la dynamique des vésicules ont été identifiées chez la levure ou chez les vertébrés par des approches biochimiques. C. elegans permet d’identifier de nouvelles protéines impliquées dans ces processus et d’analyser in vivo la fonction des composants isolés dans d’autres systèmes expérimentaux. 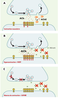 | Figure 5.
Identification de mutants de la transmission synaptique dans un crible de résistance à l’aldicarb. A. Au niveau des jonctions neuromusculaires excitatrices, les motoneurones cholinergiques libèrent de l’acétylcholine (ACh). L’acétylcholine diffuse dans la fente synaptique et se lie à des récepteurs canaux présents sur la membrane musculaire. La liaison de l’acétylcholine aux récepteurs provoque l’ouverture de canaux cationiques qui provoque une dépolarisation membranaire suivie de la contraction de la cellule musculaire. L’acétylcholine est rapidement hydrolysée par l’acétylcholinestérase (AChE). La dégradation de l’acétylcholine limite l’intensité et la durée de la stimulation des récepteurs. B. L’aldicarb est un inhibiteur de l’acétylcholinestérase. En présence d’aldicarb, l’acétylcholine s’accumule dans la fente synaptique, entraînant une hypercontraction musculaire, létale pour le ver. C. Si la transmission synaptique est altérée, la quantité d’acétylcholine libérée dans la fente synaptique est réduite et le blocage de l’acétylcholinestérase n’est plus toxique. L’aldicarb permet ainsi de sélectionner des mutants variés de la transmission synaptique ayant des défauts dans la maturation, l’exocytose ou le recyclage des vésicules synaptiques. |
Des anomalies de la libération synaptique provoquent une perturbation globale du système nerveux et en particulier du système locomoteur. C’est sur la base d’un phénotype Uncoordinated qu’ont été identifiés unc-13 [
25] et unc-18 [
26] dont la perte de fonction entraîne une paralysie du ver. Les gènes de nématode ont permis le clonage de leurs homologues chez le vertébré, les gènes Munc. Il a été montré récemment que l’invalidation de Munc18-1 ou de Munc13-1 et -2 abolit totalement la libération des vésicules synaptiques chez la souris. L’analyse de mutants Unc permet donc d’identifier des composants essentiels de la libération synaptique mais, comme nous l’avons vu, de très nombreuses mutations peuvent être à l’origine de déficits locomoteurs. Pour identifier de façon plus ciblée des gènes impliqués dans la libération synaptique des neurotransmetteurs, plusieurs laboratoires ont utilisé avec succès un crible de résistance à un neurotoxique, l’aldicarb. Cette drogue est un inhibiteur de l’acétylcholinestérase. Quand les neurones cholinergiques sont stimulés, ils libèrent de l’acétylcholine qui est très rapidement hydrolysée par l’acétylcholinestérase. Quand cette enzyme est inhibée, l’acétylcholine s’accumule dans la fente synaptique et l’hyperactivation des récepteurs de l’acétylcholine est létale (Figure 5B). Cette propriété est utilisée dans certains gaz de combat. En revanche, si la libération synaptique est inefficace, très peu d’acétylcholine est libéré par le neurone. Dans ce cas, le ver survit à l’inhibition de l’acétylcholinestérase car les quantités d’acétylcholine libérées dans la fente synaptique ne permettent pas d’atteindre des concentrations toxiques (Figure 5C). L’utilisation de l’aldicarb pour identifier des mutants de la transmission synaptique est très puissante car il s’agit de sélectionner les rares animaux qui survivent en présence de la drogue alors que le reste de la population meurt. Cette approche a permis d’identifier de nombreux gènes impliqués dans la neurotransmission [
27–
29]. Depuis 1999, l’analyse électrophysiologique de la neurotransmission chez C. elegans a été rendue possible grâce au développement d’une nouvelle préparation permettant l’enregistrement de la transmission synaptique au niveau des jonctions neuromusculaires [
30] (Figure 6). L’électrophysiologie permet de quantifier les anomalies de la neurotransmission chez les animaux mutés et de caractériser la nature du déficit. En combinant les mutations et en exprimant des protéines modifiées à partir de transgènes, il est possible d’analyser les interactions entre les constituants de la machinerie de libération synaptique et de tester in vivo les modèles échafaudés à partir de données biochimiques [
31]. La combinaison de la génétique et de la physiologie permet également de caractériser les récepteurs et les canaux présents dans la cellule enregistrée. Par exemple, l’enregistrement des cellules musculaires a révélé l’existence de deux types distincts de récepteurs de l’acétylcholine aux jonctions neuromusculaires. Néanmoins, la réalisation d’enregistrements in vivo reste techniquement délicate et n’est pas adaptée à tous les neurones. Récemment, il a été possible de réaliser des cultures primaires de cellules embryonnaires de C. elegans qui se différencient in vitro en neurones et en cellules musculaires [
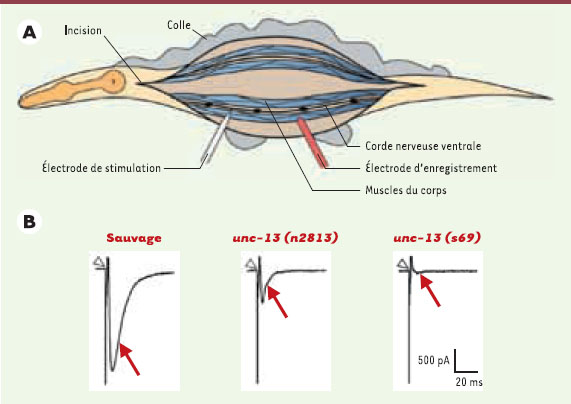
32]. En utilisant des embryons transgéniques qui expriment la GFP dans une classe de neurones particuliers, il est possible d’identifier ces mêmes neurones au sein de la culture grâce à la fluorescence de la GFP. L’enregistrement électrophysiologique permet d’analyser l’expression et la pharmacologie des canaux et récepteurs présents dans ces cellules. Les cultures à partir d’embryons mutants permettront ensuite d’analyser l’effet fonctionnel d’une mutation. 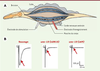 | Figure 6.
Analyse électrophysiologique d’un mutant de transmission synaptique. En 1999, J. Richmond et E. Jorgensen [30] ont mis au point une préparation de C. elegans qui permet d’enregistrer la transmission synaptique au niveau des jonctions neuromusculaires. A. Le ver est fixé sur un support avec une colle chirurgicale. Une incision latérale de la cuticule permet d’exposer les cellules musculaires et la corde nerveuse ventrale après aspiration du contenu de la cavité pseudocoelomique. La stimulation des motoneurones par une électrode placée au voisinage de la corde ventrale provoque la libération de neurotransmetteur. B. La stimulation des récepteurs de l’acétylcholine et du GABA présents aux jonctions neuromusculaires produit un courant synaptique enregistrable au niveau de la cellule musculaire (flèche rouge); la pointe du triangle indique l’artéfact de stimulation. La protéine UNC-13 joue un rôle clé dans l’exocytose des vésicules synaptiques. Chez des mutants unc-13, partiel (n2813) ou nul (s69), le courant post-synaptique est respectivement diminué ou aboli [38]. |
|
Du comportement aux molécules Dans le sol, C. elegans se trouve dans un milieu complexe et changeant où il doit trouver sa nourriture, fuir les parasites et les toxiques, rester à des températures compatibles avec son développement et éviter de pondre dans un environnement qui ne sera pas propice à la croissance de la nouvelle génération. Des comportements complexes ont été mis en place pour répondre à ces contraintes environnementales. Cependant, la simplicité du système nerveux de C. elegans permet d’identifier les cellules impliquées dans des processus intégrés et, au sein de ces cellules, d’extraire les composants critiques des voies de signalisation mises en jeu. Par exemple, C. elegans a un répertoire chémosensoriel très important. Il détecte plusieurs centaines de substances chimiques dont au moins une soixantaine de produits volatiles. Certaines de ces substances odorantes telles que le diacétyl ou la pyrazine sont attractives, d’autres comme la 2-nonanone sont répulsives. En utilisant un laser, il est possible de tuer spécifiquement un ou plusieurs neurones dans un embryon ou une larve. C. Bargmann et al. ont ainsi montré que les neurones sensoriels AWA et AWC sont responsables de la détection des odeurs attractives alors que les neurones AWB détectent les odeurs répulsives (pour revue, voir [
33]). Un crible pour des vers ayant perdu la capacité de sentir le diacétyl mais toujours capables de sentir la pyrazine a permis d’identifier odr-10 (odor defective). Ce gène code pour un récepteur à sept hélices transmembranaires couplé aux protéines G, uniquement exprimé dans le cil de la paire de neurones AWA [
34]. Un mutant odr-10 chez lequel on exprime la forme fonctionnelle de ODR-10 dans les neurones AWB n’est plus attiré mais repoussé par le diacétyl. La nature attractive ou répulsive de l’odeur semble donc codée par le type de neurone stimulé plutôt que par la nature moléculaire du récepteur activé. L’exposition au diacétyl de cellules de mammifères en culture exprimant ODR-10 entraîne une augmentation transitoire du calcium intracellulaire [
35]. ODR-10 est le premier chémorécepteur lié aux protéines G dont le ligand ait été déterminé. L’analyse de mutants de l’olfaction a permis d’identifier, selon la même logique, les composants principaux de la cascade de signalisation intracellulaire situés en aval de ODR-10. Les comportements de chémosensations ou de thermosensation sont plastiques. C. elegans est par exemple capable d’associer une température spécifique à la présence de nourriture (pour revue, voir [
36]). Les animaux sont normalement cultivés en présence de nourriture à une température entre 15 et 25 °C. Transférés sur des boîtes sans nourriture où un gradient de température a été établi, les vers vont migrer et se déplacer sur l’isotherme correspondant à la température à laquelle ils avaient été cultivés en présence de nourriture. En revanche, si les animaux ont été antérieurement affamés, ils vont fuir la température correspondante. Il faut deux à quatre heures pour acclimater les vers à une nouvelle température. Les neurones sensoriels AFD et les deux paires d’interneurones AIY et AIZ sont nécessaires à la perception et au traitement de l’information thermique. ncs-1 (neural calcium sensor) code pour une protéine intracellulaire liant le calcium, conservée de la levure aux mammifères. L’inactivation de ncs-1 ou sa surexpression modifie l’association du stimulus thermique à la présence de nourriture [
37]. C. elegans constitue donc un système simple qui permet d’analyser le traitement de l’information avec des outils génétiques. Le développement de nouveaux marqueurs de l’activité neuronale permettra peut-être à terme de visualiser en temps réel l’état du réseau au cours du traitement de l’information et de la production de comportements. |
Malgré la distance évolutive qui sépare C. elegans et les mammifères, nous constatons qu’aux échelles moléculaire et cellulaire, la physiologie du neurone est très conservée entre ces organismes. Les outils génétiques disponibles chez C. elegans permettent d’extraire les éléments critiques des réseaux d’interactions mis en jeu dans des processus cellulaires ou comportementaux. La très grande simplicité de son système nerveux permet donc d’utiliser le ver comme un « tube à essai vivant » pour identifier de nouveaux gènes, analyser les voies moléculaires de signalisation et explorer la biologie cellulaire du neurone in vivo.

|
1. Brenner S. The genetics of Caenorhabditis elegans. Genetics 1974; 77: 71–94. 2. White JG, Southgate E, Thomson JN, Brenner S. The structure of the nervous system of Caenorhabditis elegans. Philos Trans R Soc Lond B Biol Sci 1984; 314: 1–340. 3. Bargmann CI. Neurobiology of the Caenorhabditis elegans genome. Science 1998; 282: 2028–33. 4. Genome sequence of the nematode C. elegans: A platform for investigating biology. The C. elegans Sequencing Consortium. Science 1998; 282: 2012–8. 5. Jin Y, Jorgensen E, Hartwieg E, Horvitz HR. The Caenorhabditis elegans gene unc-25 encodes glutamic acid decarboxylase and is required for synaptic transmission but not synaptic development. J Neurosci 1999; 19: 539–48. 6. Hedgecock EM, Culotti JG, Hall DH. The unc-5, unc-6, and unc-40 genes guide circumferential migrations of pioneer axons and mesodermal cells on the epidermis in C. elegans. Neuron 1990; 4: 61–85. 7. Chan SS, Zheng H, Su MW, et al. UNC-40, a C. elegans homolog of DCC (Deleted in Colorectal Cancer), is required in motile cells responding to UNC-6 netrin cues. Cell 1996; 87: 187–95. 8. Leung-Hagesteijn C, Spence AM, Stern BD, et al. UNC-5, a transmembrane protein with immunoglobulin and thrombospondin type 1 domains, guides cell and pioneer axon migrations in C. elegans. Cell 1992; 71: 289–99. 9. Serafini T, Kennedy TE, Galko MJ, Mirzayan C, Jessell TM, Tessier-Lavigne M. The netrins define a family of axon outgrowthpromoting proteins homologous to C. elegans UNC-6. Cell 1994; 78: 409–24. 10. Keino-Masu K, Masu M, Hinck L, et al. Deleted in Colorectal Cancer (DCC) encodes a netrin receptor. Cell 1996; 87: 175–85. 11. Colavita A, Culotti JG. Suppressors of ectopic UNC-5 growth cone steering identify eight genes involved in axon guidance in Caenorhabditis elegans. Dev Biol 1998; 194: 72–85. 12. Colavita A, Krishna S, Zheng H, Padgett R W, Culotti JG. Pioneer axon guidance by UNC-129, a C. elegans TGF-beta. Science 1998; 281: 706–9. 13. Chalfie M, Tu Y, Euskirchen G, Ward WW, Prasher DC. Green fluorescent protein as a marker for gene expression. Science 1994; 263: 802–5. 14. Huang X, Cheng HJ, Tessier- Lavigne M, Jin Y. MAX-1, a novel PH/MyTH4/FERM domain cytoplasmic protein implicated in netrin-mediated axon repulsion. Neuron 2002; 34: 563–76. 15. Hao JC, Yu TW, Fujisawa K, et al. C. elegans slit acts in midline, dorsal-ventral, and anterior-posterior guidance via the SAX- 3/Robo receptor. Neuron 2001; 32: 25–38. 16. Yu TW, Bargmann CI. Dynamic regulation of axon guidance. Nat Neurosci 2001; 4 Suppl: 1169–76. 17. Branda CS, Stern MJ. Cell migration and axon growth cone guidance in Caenorhabditis elegans. Curr Opin Genet Dev 1999; 9: 479–84. 18. Knobel KM, Jorgensen EM, Bastiani MJ. Growth cones stall and collapse during axon outgrowth in Caenorhabditis elegans. Development 1999; 126: 4489–98. 19. White JG, Southgate E, Thomson JN. Mutations in the Caenorhabditis elegans unc-4 gene alter the synaptic input to ventral cord motor neurons. Nature 1992; 355: 838–41. 20. Miller DM, Shen MM, Shamu CE, et al. C. elegans unc-4 gene encodes a homeodomain protein that determines the pattern of synaptic input to specific motor neurons. Nature 1992; 355: 841–5. 21. Winnier AR, Meir JY, Ross JM, et al. UNC-4/UNC-37- dependent repression of motor neuron-specific genes controls synaptic choice in Caenorhabditis elegans. Genes Dev 1999; 13: 2774–86. 22. Nonet ML. Visualization of synaptic specializations in live C. elegans with synaptic vesicle protein- GFP fusions. J Neurosci Methods 1999; 89: 33–40. 23. Jin Y. Synaptogenesis: insights from worm and fly. Curr Opin Neurobiol 2002; 12: 71–9. 24. Gally C, Bessereau JL. GABA is dispensable for the formation of junctional GABA receptor clusters in Caenorhabditis elegans. J Neurosci 2003; 23: 2591–9. 25. Maruyama IN, Brenner S. A phorbol ester/diacylglycerolbinding protein encoded by the unc-13 gene of Caenorhabditis elegans. Proc Natl Acad Sci U S A 1991; 88: 5729–33. 26. Gengyo-Ando K, Kamiya Y, Yamakawa A, et al. The C. elegans unc-18 gene encodes a protein expressed in motor neurons. Neuron 1993; 11: 703–11. 27. Miller KG, Alfonso A, Nguyen M, Crowell JA, Johnson CD, Rand JB. A genetic selection for Caenorhabditis elegans synaptic transmission mutants. Proc Natl Acad Sci U S A 1996; 93: 12593–8. 28. Nonet ML, Saifee O, Zhao H, Rand JB, Wei L. Synaptic transmission deficits in Caenorhabditis elegans synaptobrevin mutants. J Neurosci 1998; 18: 70–80. 29. Harris TW, Hartwieg E, Horvitz HR, Jorgensen EM. Mutations in synaptojanin disrupt synaptic vesicle recycling. J Cell Biol 2000; 150: 589–600. 30. Richmond JE, Jorgensen EM. One GABA and two acetylcholine receptors function at the C. elegans neuromuscular junction. Nat Neurosci 1999; 2: 791–7. 31. Richmond JE, Weimer RM, Jorgensen EM. An open form of syntaxin bypasses the requirement for UNC-13 in vesicle priming. Nature 2001; 412: 338–41. 32. Christensen M, Estevez A, Yin X, et al. A primary culture system for functional analysis of C. elegans neurons and muscle cells. Neuron 2002; 33: 503–14. 33. Bargmann CI, Kaplan JM. Signal transduction in the Caenorhabditis elegans nervous system. Annu Rev Neurosci 1998; 21: 279–308. 34. Sengupta P, Chou JH, Bargmann CI. odr-10 encodes a seven transmembrane domain olfactory receptor required for responses to the odorant diacetyl. Cell 1996; 84: 899–909. 35. Zhang Y, Chou JH, Bradley J, Bargmann CI, Zinn K. The Caenorhabditis elegans seven-transmembrane protein ODR-10 functions as an odorant receptor in mammalian cells. Proc Natl Acad Sci U S A 1997; 94: 12162–7. 36. Mori I. Genetics of chemotaxis and thermotaxis in the nematode Caenorhabditis elegans. Annu Rev Genet 1999; 33: 399–422. 37. Gomez M, De Castro E, Guarin E, et al. Ca2+ signaling via the neuronal calcium sensor-1 regulates associative learning and memory in C. elegans. Neuron 2001; 30: 241–8. 38. Richmond JE, Davis WS, Jorgensen EM. UNC-13 is required for synaptic vesicle fusion in C. elegans. Nat Neurosci 1999; 2: 959–64. |