La drosophile a largement fait ses preuves comme système modèle pour l’étude des voies de signalisation contrôlant les divers aspects du fonctionnement cellulaire. Cependant, la contribution de ce modèle à l’étude des processus de transformation cancéreuse est restée, jusqu’à récemment, très limitée [ 1].
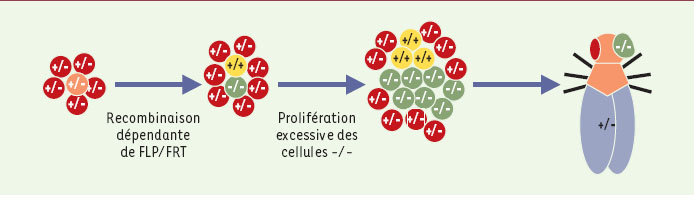
L’apparition de nouveaux outils génétiques devrait permettre de combler rapidement ce retard. Par recombinaison au cours de la mitose, une cellule hétérozygote pour une mutation donnée peut engendrer une cellule homozygote mutante et une cellule homozygote de type sauvage (Figure 1). La mise à profit du système de recombinaison inductible FLP/FRT (flippase, flippase recognition targets) chez la drosophile permet de déclencher à volonté ce type de recombinaison au cours du développement [1] (→). On peut ainsi produire des clones de cellules mutantes au sein d’un organisme hétérozygote et évaluer leur potentiel prolifératif dans le contexte d’un organisme vivant (Figure 1). Le laboratoire d’Iswar Hariharan au MGH Cancer Center de Boston (MA, USA) a utilisé ce principe pour rechercher des mutations induisant une prolifération cellulaire incontrôlée au niveau de l’œil [ 2]. Au cours de ce crible génétique, 23 groupes de mutations ont été isolés sur environ 300000 mouches testées. Certaines mutations touchaient des homologues de suppresseurs de tumeurs de mammifères (dPTEN, tuberous sclerosis complexe [Tsc1/2]), d’autres ont permis d’identifier des gènes inconnus, dont le gène salvador (sav) qui a fait l’objet d’une récente publication dans Cell [ 3]. Les cellules mutantes pour sav prolifèrent anormalement et augmentent la taille de l’œil (Figure 2). Cet excès de cellules mutantes provient de la combinaison de deux effets: un défaut de sortie du cycle cellulaire et un défaut de déclenchement de la mort cellulaire programmée ou apoptose.
| Figure 1.
Utilisation du système FLP/FRT pour identifier de nouveaux régulateurs de la prolifération. Dans un organisme hétérozygote pour une mutation donnée (+/-), le système FLP/FRT permet d’induire la recombinaison mitotique de façon à produire une cellule homozygote mutante (-/-) et une cellule homozygote sauvage pour cette mutation (+/+). Si la mutation confère un avantage prolifératif, la descendance de la cellule mutante envahit le tissu. Dans le crible génétique effectué [2], on provoque la recombinaison dans l’œil de façon massive. Si la mutation testée confère un avantage prolifératif, l’œil sera composé essentiellement des cellules homozygotes mutantes et deviendra éventuellement plus volumineux (voir Figure 2). |
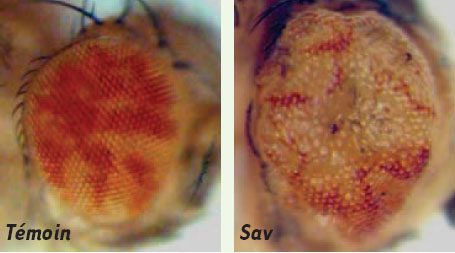
| Figure 2.
La mutation salvador confère un avantage prolifératif aux cellules mutantes. La mutation salvador confère un avantage prolifératif aux cellules mutantes (en blanc) par rapport au cellules saines (en rouge). Dans l’œil témoin, on observe une représentation égale des populations blanches et rouges. (Reproduit d’après [3], avec la permission de Elsevier Science). |
(→) m/s 1995, n° 5, p. 735
Le défaut de sortie du cycle cellulaire conduit les cellules mutantes à effectuer un à deux cycles supplémentaires par rapport au programme normal de division. L’arrêt de la prolifération à certaines étapes du développement de la drosophile nécessite l’élimination de l’activité cycline E/Cdk2. Dans les clones de cellules mutantes pour sav, on observe une persistance de l’expression du messager codant pour la cycline E à une étape où il a disparu dans les cellules sauvages [3]. Cela pourrait expliquer, du moins en partie, le retard avec lequel les cellules quittent le cycle cellulaire.
Les mutations sav affectent également l’apoptose. Des signaux pro-apoptotiques interviennent normalement dans le développement de l’œil pour éliminer les cellules excédentaires et dessiner ainsi l’arrangement régulier des unités qui composent l’œil des ommatidies [ 4]. Les cellules mutantes pour sav ne répondent pas à ces signaux et ne sont donc pas éliminées. L’activation de la cascade apoptotique dépend des protéines proapoptotiques Reaper, Hid et Grim (→).
(→) m/s 2002, n° 8-9, p. 831
Ces protéines agissent sur DIAP1 (Drosophila inhibitor of apoptosis) en réduisant son expression et en le séquestrant par interaction directe. La fonction normale de DIAP1 est de réprimer l’activation des caspases, les protéases à cystéine qui « exécutent » le programme apoptotique. En présence de Hid, Grim ou Reaper, la fonction de DIAP1 est bloquée, et les caspases ainsi déréprimées entraînent la mort des cellules. Où se place sav dans ce schéma ? Dans les cellules mutantes pour sav, on observe que l’activation des caspases résultant de la surexpression de Hid ou de Reaper est bloquée. Cela est consécutif à une élévation générale de l’expression de DIAP1 dans ces cellules bien que le niveau d’expression du messager codant pour DIAP1 soit normal. Il semble donc que Sav soit normalement impliquée dans la répression de l’expression de DIAP1 par un mécanisme posttranscriptionnel [3].
(→) m/s 1999, n° 8-9, p. 1057
Le gène salvador a été cloné. Il code pour une protéine de 608 acides aminés contenant deux domaines WW et une région coiled-coil mais aucun domaine catalytique connu. Les domaines WW interagissent avec les motifs Pro-Pro-XTyr (PY), et les domaines coiled-coil ont également été impliqués dans des interactions entre protéines, suggérant que Sav interagit avec différents partenaires dans la cellule [ 5]. La protéine kinase Lats (large tumor suppressor)/Warts, qui avait été identifiée dans un crible similaire, pourrait être l’un des partenaires de Sav [ 6] (→). En effet, le phénotype des mutants lats est très proche de celui des mutants sav: retard de l’arrêt de la prolifération et réduction de l’apoptose [3]. De plus, Lats possède cinq motifs PY et pourrait donc interagir directement avec Sav. Cette interaction a été démontrée par un test in vitro et un test génétique [3]. Sav et Lats agiraient donc de concert dans une voie de signalisation responsable du contrôle coordonné de la prolifération et de l’apoptose. Cet aspect est particulièrement intéressant car peu de protéines capables de combiner le contrôle de ces deux voies cruciales dans la carcinogenèse ont été décrites.
Plusieurs questions se posent concernant les fonctions cellulaires de Sav et de son partenaire Lats. Comment la protéine Sav contrôle-t-elle la transcription du gène de la cycline E et l’expression de DIAP1 ? Salvador pourrait moduler la stabilité de DIAP1, protéine très instable dont la quantité est contrôlée de façon très précise à la fois par un contrôle général de la traduction impliquant Reaper et Grim et par dégradation. Celle-ci est dépendante de l’ubiquitinylation de DIAP1, à laquelle Hid, Grim, Reaper et DIAP1 elle-même participent, ainsi que les ubiquitine ligases Morgue et UbcD1 [ 7]. Il sera intéressant de savoir où Sav intervient dans ce contexte. Par ailleurs, la voie Ras/MAPK a également été impliquée dans un contrôle coordonné de la prolifération et de l’apoptose chez la drosophile et les mammifères [ 8]. Existe-il des liens entre Sav et cette voie de signalisation? Même si les mutations sav ne semblent pas affecter l’activation des MAPK [3], il sera intéressant de savoir si, à l’inverse, Sav est une cible de la voie Ras.
La découverte, chez la drosophile, de Salvador et de son interaction avec Lats, ouvre de nouvelles perspectives dans le domaine de la cancérologie. En effet, ces deux protéines ont des homologues chez l’homme : hWW45 pour Sav, LATS1 et LATS2 pour Lats [ 9, 10]. Plusieurs arguments indiquent que ces homologues fonctionnent probablement comme des suppresseurs de tumeurs chez les mammifères. En effet, la transfection du gène LATS1 dans différentes lignées tumorales humaines induit soit l’apoptose, soit l’arrêt du cycle cellulaire et supprime ainsi leur capacité de former des tumeurs chez des souris immunodéprimées [ 11]. Par ailleurs, des souris dont le gène LATS1 a été invalidé sont hypersensibles aux carcinogènes et développent toutes des tumeurs bénignes de l’ovaire, ainsi que, plus rarement, des sarcomes métastatiques des tissus mous [10] (→). Ce phénotype n’est pas aussi spectaculaire que celui des mutants de drosophile, probablement parce que LATS2 peut suppléer l’absence de LATS1, mais il met néanmoins clairement en lumière un rôle suppresseur de tumeur pour cette protéine.
Le gène hWW45, quant à lui, est situé dans une région du génome humain 14q13-14q23 caractérisée par une perte d’allélisme dans différents types de cancers, notamment des cancers du rein [3]. De plus, l’analyse d’une cinquantaine de lignées dérivées de tumeurs humaines a montré que le gène était délété dans deux lignées indépendantes dérivées de cancers du rein dans lesquelles l’allèle normal était absent.
L’ensemble de ces données révèle le fort potentiel de ces gènes pour des applications diagnostiques ou thérapeutiques. Cependant, il reste à comprendre leur rôle exact dans le choix pour la cellule entre cesser de proliférer ou mourir. Étant donné les parallèles dressés avec les gènes mammifères, la contribution du modèle drosophile nous sera encore précieuse.