| |
| Med Sci (Paris). 2003 January; 19(1): 85–91. Published online 2003 January 15. doi: 10.1051/medsci/200319185.Les homologues de MutS et de MutL au cours de la méiose chez les mammifères Sabine Santucci-Darmanin and Véronique Paquis-Flucklinger* Équipe M3R, LRC CEA n° 32-V, UMR Cnrs UNSA 6549, Faculté de Médecine, avenue de Valombrose, 06107 Nice Cedex 2, France |
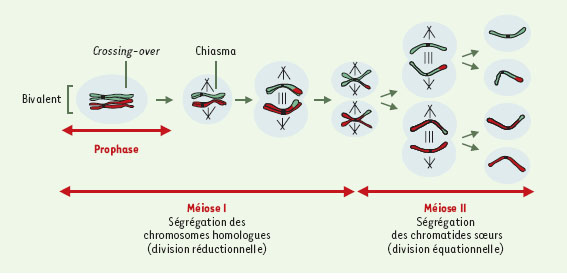
La méiose est un type particulier de division cellulaire qui assure la production de gamètes haploïdes à partir de cellules parentales diploïdes. Lors de la méiose, une seule réplication de l’ADN est suivie de deux divisions cellulaires successives (Figure 1). Alors que la méiose II est semblable à une mitose puisqu’elle permet la ségrégation de chromatides sœurs, la méiose I (dite division réductionnelle) présente certaines particularités. Notamment, au cours de la prophase de méiose I, chaque chromosome dupliqué s’apparie avec son homologue formant ainsi une structure appelée « bivalent » qui contient alors quatre chromatides, deux maternelles et deux paternelles (Figure 1). Lors de la méiose I, ce sont les chromosomes homologues qui se séparent et non les chromatides sœurs.
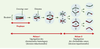 | Figure 1. Les deux divisions méiotiques. Une cellule diploïde donne naissance à quatre cellules filles haploïdes après deux divisions successives. La méiose I est une division réductionnelle au cours de laquelle les chromosomes homologues ségrègent; la méiose II est une division équationnelle au cours de laquelle des chromatides sœurs ségrègent |
|
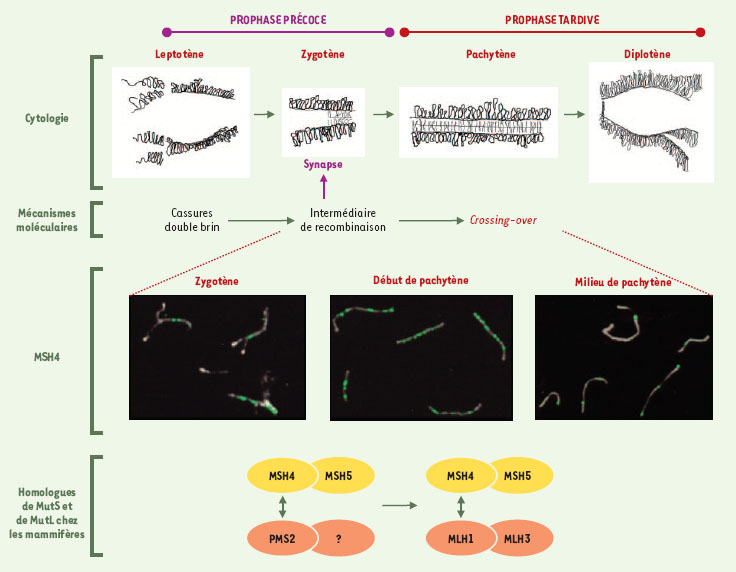
Recombinaison méiotique et ségrégation des chromosomes homologues Au cours de la prophase de méiose I, parallèlement à l’appariement des chromosomes homologues, se déroulent les mécanismes de recombinaison méiotique qui aboutiront à des échanges réciproques de matériel génétique (crossing-over) entre les chromatides maternelles et paternelles. Ces échanges sont indispensables à la diversité génétique d’une part, et à la ségrégation correcte des chromosomes homologues lors de la première division de méiose, d’autre part. En fin de prophase, les chromosomes homologues sont connectés l’un à l’autre par l’intermédiaire d’au moins un crossing-over. Ces connexions, visibles en cytologie, sont appelées chiasmas. Elles sont nécessaires pour une orientation correcte des paires de chromosomes sur le fuseau achromatique lors de la métaphase. Un déficit en crossing-over entraîne une diminution du nombre de chiasmas et une ségrégation incorrecte (phénomène de non-disjonction) des chromosomes homologues. Les cellules filles possèdent alors un nombre anormal de chromosomes. La prophase de méiose I est divisée, au cours du temps, en cinq stades définis par des modifications morphologiques des chromosomes (Figure 2). La relation entre ces modifications morphologiques et les différentes étapes de la recombinaison méiotique n’est pas clairement déterminée. Toutefois, les travaux réalisés chez la levure [1] et, plus récemment, chez les mammifères [2] indiquent que les mécanismes de recombinaison méiotique débutent dès le stade leptotène par des cassures double-brins de l’ADN, se poursuivent au stade zygotène au cours duquel ils sont requis pour l’appariement (ou synapse) des chromosomes homologues, et aboutissent à la formation des crossing-over au stade pachytène, stade au cours duquel les chromosomes sont appariés sur toute leur longueur.
 | Figure 2.Immunolocalisation de la protéine MSH4 sur les chromosomes au cours des différents stades de la prophase de méiose. Au stade leptotène, les chromosomes se rapprochent et se condensent. La synapse débute au zygotène et est complète au pachytène. Au diplotène, les homologues commencent à se séparer. D’un point de vue moléculaire, les mécanismes de recombinaison méiotique débutent au leptotène par des cassures double brin de l’ADN, se poursuivent au zygotène, où ils sont indispensables à la synapse, et se terminent au pachytène où ils aboutissent à la formation des crossingover. L’immunolocalisation de MSH4 (visible en vert) sur les chromosomes méiotiques (visibles en blanc) obtenus à partir d’étalements de spermatocytes de souris, révèle que cette protéine s’associe aux chromosomes appariés dès le stade zygotène et jusqu’en milieu de pachytène [ 15]. Chez les mammifères, au fil de la prophase, la protéine MSH4 change de partenaire. En prophase précoce, lors de la synapse, elle interagit avec la protéine MSH5 et peut-être avec la protéine PMS2. Le second homologue de MutL présent à ce stade reste à identifier. Différents travaux suggèrent qu’en prophase tardive, lors de la formation des crossing-over, MSH4 est associée à l’hétérodimère MLH1-MLH3. Bien que les données disponibles chez la levure soient en faveur d’un hétérodimère de type MSH4/MSH5, la présence de MSH5 à ce stade n’est pas démontrée chez les mammifères. |
|
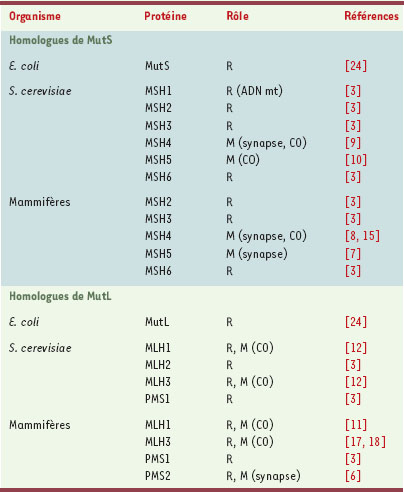
Homologues de MutS et de MutL, réparation des mésappariements de l’ADN et recombinaison méiotique Chez Escherichia coli, MutS, MutL et MutH sont des facteurs essentiels du système de réparation post-réplicatif des mésappariements de l’ADN (système MMR, mutation mismatch repair) [3]. Au cours de l’évolution, ce système a été très conservé et s’est spécialisé. Chez la levure Saccharomyces cerevisiae, six homologues de MutS (protéines MSH, MutS homolog) et quatre homologues de MutL (protéines MLH, MutL homolog et protéines PMS, post-meiotic segregation) sont présents [3] (Tableau I). L’implication de ces protéines dans la réparation des mésappariements de l’ADN a été rapidement démontrée. La reconnaissance d’un mésappariement, chez la levure comme chez l’homme, se fait par un hétérodimère de protéines MSH. Une fois fixées à l’ADN, elles interagissent avec un hétérodimère constitué de deux homologues de MutL. L’interaction entre ces deux complexes protéiques est indispensable à la mise en route du processus de réparation [3]. Chez l’homme, des mutations dans certains gènes MSH et MLH sont responsables du développement de cancers héréditaires de type HNPCC (hereditary non polyposis colon cancer) mais aussi de cancers sporadiques [4]. La mise en évidence d’un défaut du système MMR dans des cancers humains explique que la fonction de réparation des protéines MSH et MLH a été très étudiée au cours des dix dernières années.
 | Tableau I. Homologues de MutS et de MutL. Les homologues de MutS et de MutL interviennent dans la réparation des mésappariements de l’ADN (R) et/ou au cours de la recombinaison méiotique (M). Dans ce cas, ils jouent un rôle dans la formation des synapses (synapse) et/ou des crossing-over (CO). mt: mitochondrial. |
Plusieurs homologues de MutS et de MutL interviennent également au cours de la méiose (Tableau I). Certains sont nécessaires à la réparation des mésappariements présents au niveau des ADN hétéroduplex formés lors de la recombinaison. Cela a été démontré en particulier pour la protéine MSH2 chez S. cerevisiae [5]. Par ailleurs, il a été montré que plusieurs homologues de MutS et MutL sont nécessaires pour l’appariement correct des chromosomes homologues en début de prophase de méiose [6– 8]. Néanmoins, les mécanismes moléculaires impliqués dans ce cas ne sont pas connus. Enfin, certaines protéines MSH et MLH sont indispensables à la formation des crossing-over [9–12]. |
Protéines MSH, MLH et recombinaison méiotique Chez la levure Dès 1994, les travaux des groupes de Roeder et Hollingsworth révèlent que deux des protéines de la famille MSH identifiées chez la levure, MSH4 et MSH5, sont exprimées uniquement au cours de la méiose et sont toutes deux impliquées dans la recombinaison méiotique [ 9, 10]. Un déficit en protéines MSH4 ou MSH5 induit une diminution de la fréquence de crossing-over accompagnée d’un phénomène de non-disjonction. En revanche, l’absence de phénotype mutateur chez ces levures révèle que MSH4 et MSH5 ne participent pas à la réparation des bases non (ou mal) appariées [ 9, 10]. Parmi les protéines MSH connues chez la levure, MSH4 et MSH5 fonctionnent sous forme d’hétérodimère et sont les seules nécessaires à la formation des crossing-over [ 13]. Les deux homologues de MutL, MLH1 et MLH3, initialement étudiés pour leur rôle dans la réparation, sont également requis pour obtenir un nombre correct de crossing-over [ 12]. Ces deux protéines, qui fonctionnent sous forme d’hétérodimère, sont vraisemblablement engagées dans la même voie moléculaire que les protéines MSH4 et MSH5. Chez les mammifères Dès 1995, Baker et ses collaborateurs montrent que la situation chez les mammifères est plus complexe [ 6]. En effet, certains homologues de MutS et de MutL sont requis non seulement pour la formation des crossingover au stade pachytène, mais interviennent également à un stade plus précoce, lors de la synapse des chromosomes. Les homologues de MutS La protéine MSH4 est uniquement exprimée au cours de la prophase de méiose I chez l’homme [ 14, 15]. Contrairement aux protéines MSH2, MSH3 et MSH6, elle n’intervient pas dans la réparation des mésappariements de l’ADN. Dans les cellules germinales, la protéine MSH4 est localisée sous forme de foyers répartis le long des chromosomes méiotiques [ 15]. Ces foyers MSH4 sont associés aux chromosomes dès que ceux-ci commencent à s’apparier au stade zygotène, leur nombre augmente au début du stade pachytène puis décroît jusqu’à leur disparition totale en fin de pachytène (Figure 2). Ces résultats suggèrent que MSH4 intervient lors des étapes précoces de recombinaison nécessaires à la synapse des chromosomes, et plus tard au cours du stade pachytène lors de la formation des crossing-over. Kneitz et al. ont montré que des souris msh4−/−, chez lesquelles le gène MSH4 a été inactivé à l’état homozygote, présentent un défaut de synapse des chromosomes homologues entraînant un blocage de la gamétogenèse et une stérilité [ 8]. Ce défaut, qui n’est pas sans rappeler le phénotype observé chez certains patients stériles ayant un blocage précoce de la spermatogenèse, confirme que MSH4 intervient dès le stade zygotène chez les mammifères. MSH5 est également indispensable au déroulement correct de la prophase I de méiose. En effet, des souris déficitaires en protéine MSH5 (souris msh5−/−) présentent, de la même façon que les souris msh4−/−, une stérilité associée à un défaut d’appariement des chromosomes [7]. Chez l’homme, les protéines MSH4 et MSH5 sont capables d’interagir in vitro, suggérant qu’elles fonctionnent sous forme d’hétérodimère. Les homologues de MutL PMS2 est l’un des homologues de la protéine MutL, essentiellement étudié pour son rôle dans la réparation des mésappariements de l’ADN. Néanmoins, l’analyse phénotypique de souris mâles pms2−/− a révélé que cette protéine était également essentielle pour l’appariement des chromosomes homologues au cours de la méiose. Ces souris sont stériles et présentent un défaut d’appariement des chromosomes [ 6], similaire à celui des souris msh4−/− et msh5−/− [ 8, 7]. Cette observation suggère que MSH4, MSH5 et PMS2 appartiennent à un même complexe protéique impliqué dans les étapes précoces de la recombinaison méiotique, complexe nécessaire à l’appariement des chromosomes homologues au stade zygotène (Figure 2). Cette hypothèse est confortée par la mise en évidence d’une interaction in vitro entre les protéines PMS2 et MSH4. La protéine MLH1 participe à la fois à la réparation des mésappariements de l’ADN et au processus de recombinaison méiotique. Néanmoins, contrairement à PMS2, MLH1 ne joue aucun rôle au moment de la synapse des chromosomes [11]. En revanche, elle est associée aux chromosomes méiotiques au moment de la formation des crossing-over, formation pour laquelle MLH1 est indispensable [11]. À ce stade de la prophase, il existe des arguments suggérant que MLH1 et MSH4 fonctionnent en association (Figure 2). En effet, ces deux protéines sont co-localisées sur les chromosomes méiotiques et il a été montré qu’elles interagissent in vitro [15]. Par analogie avec la levure, il est vraisemblable que les homologues de MutL fonctionnent sous forme d’hétérodimères au cours du processus de recombinaison méiotique chez les mammifères. Dans cette hypothèse, le second homologue de MutL associé à PMS2 au moment de l’appariement des chromosomes reste à identifier. En revanche, différents travaux permettent de proposer aujourd’hui que l’homologue de MutL impliqué avec MLH1 dans la formation des crossing-over, est la protéine MLH3 (Figure 2). Cette protéine, récemment identifiée chez l’homme [16], était un candidat intéressant dans la mesure où, chez Saccharomyces cerevisiae, elle participe aux mécanismes de recombinaison méiotique [12]. Il a été récemment montré que le gène MLH3 est exprimé dans les cellules méiotiques de souris et dans le testicule chez l’homme [17]. La protéine MLH3 est co-immunoprécipitée avec MSH4 dans des extraits de spermatocytes de souris et ces deux protéines sont également capables d’interagir in vitro [17]. L’analyse ultérieure de souris mlh3−/− a permis de confirmer l’implication de la protéine MLH3 dans la méiose [18]. Chez ces souris, de même que chez les souris mlh1−/−, on observe un appariement correct des chromosomes homologues mais un défaut dans la formation des chiasmas. Il en résulte un blocage de la gamétogenèse et une stérilité. Par ailleurs, il a été montré que les protéines MLH1 et MLH3 interagissent [16] et qu’elles sont co-localisées sur les chromosomes méiotiques en fin de prophase. L’ensemble de ces résultats suggère que MSH4, en fin de prophase, est associé à l’hétérodimère MLH1-MLH3, qui est impliqué dans la formation des crossing-over. En conclusion, il semblerait donc que des tétramères composés de protéines MSH et MLH interviennent successivement lors de la synapse et lors de la formation des crossing-over au cours de la méiose chez les mammifères. Un hétérodimère MSH4/MSH5 est vraisemblablement présent aux cours de ces deux étapes, alors que les homologues de MutL sont différents (Figure 2). Ces résultats laissent supposer que la spécificité des fonctions « précoces » et « tardives » de l’hétérodimère MSH4/MSH5 pourrait être en partie liée à la nature des homologues de MutL avec lequel il interagit. |
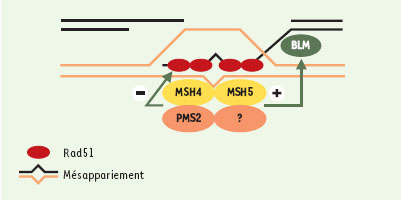
Protéines MSH et MLH et machinerie précoce de recombinaison méiotique Les analyses génétiques faites chez la levure ont permis de caractériser de nombreuses protéines impliquées dans les différentes étapes des mécanismes de recombinaison méiotique. Les homologues de certaines d’entre elles ont été identifiés chez les mammifères, parmi lesquels Rad51 et Dmc1, qui sont deux enzymes essentielles de la machinerie précoce de recombinaison méiotique [19]. Toutes deux sont des homologues de la protéine bactérienne RecA. Rad51 est exprimée dans les cellules somatiques et les cellules germinales, alors que Dmc1 a une expression restreinte à la méiose. In vitro, Rad51 se fixe sur l’ADN simple brin, formant un filament nucléoprotéique capable « d’envahir » une molécule d’ADN double brin homologue (on parle de réaction d’échange de brin ou d’activité « recombinase ») [19]. Dmc1 catalyse également cette réaction d’invasion in vitro. Rad51 et Dmc1 interagissent et sont co-localisées au niveau des chromosomes en prophase de méiose [20]. Après la formation des cassures double brin de l’ADN, ces deux « recombinases » agissent vraisemblablement de concert in vivo pour catalyser la formation d’intermédiaires de recombinaison entre chromosomes homologues. Différents arguments suggèrent que certains des homologues de MutS et de MutL et les recombinases interviennent dans les mécanismes moléculaires requis pour la synapse. D’une part, l’inactivation homozygote du gène Dmc1 chez la souris entraîne un défaut d’appariement des chromosomes homologues [19] proche de celui des souris msh4−/− et msh5−/−. D’autre part, des travaux récents montrent que MSH4 et Rad51 sont capables d’interagir in vitro et que ces deux protéines sont co-localisées sur les chromosomes méiotiques au stade zygotène. De plus, il a été montré que PMS2 est capable d’interagir avec Rad51 in vitro. L’ensemble de ces observations suggère que MSH4, associée à MSH5 et potentiellement à PMS2, interviendrait lors des étapes précoces de la recombinaison méiotique. Quelle pourrait-être alors la fonction « précoce » de MSH4, MSH5 et PMS2? Diverses études indiquent qu’un des rôles, conservé au cours de l’évolution, de certaines protéines des familles MSH et MLH est d’inhiber la recombinaison entre des ADN non homologues [21]. Worth et al. ont démontré que les protéines MutS et MutL peuvent inhiber in vitro l’activité « recombinase » de RecA, lorsque cet échange de brin concerne deux molécules d’ADN non identiques [22]. De la même façon, on peut supposer que l’hétérodimère MSH4/MSH5, associé vraisemblablement à un hétérodimère d’homologues de MutL comprenant la protéine PMS2, pourrait être présent au niveau des intermédiaires de recombinaison nouvellement formés afin de détecter les éventuels mésappariements et d’inhiber la recombinaison entre ADN non homologues. Cette fonction d’inhibition de la recombinaison pourrait s’exercer de différentes façons: (1) on peut imaginer que le complexe MSH4/MSH5 agisse directement sur Rad51 afin de bloquer son activité « recombinase » (Figure 3); (2) l’hétérodimère MSH4/MSH5 pourrait agir de concert avec des enzymes de la recombinaison de façon à inverser le processus de recombinaison. Pour cela, le complexe MSH4/MSH5 pourrait stimuler la fonction « anti-recombinase » de la protéine BLM. BLM est une hélicase de type RecQ codée par un gène qui est muté chez les patients atteints du syndrome de Bloom (→). Ce syndrome est associé notamment à une prédisposition à différents types de cancers et, à l’échelle moléculaire, à une très forte instabilité génétique (cassures chromosomiques et hyper-recombinaison). Des travaux récents semblent indiquer que BLM exerce une fonction qualifiée d’« antirecombinase » qui catalyse la migration inverse des jonctions de Holliday (jonction à quatre brins d’ADN formée au cours du processus de recombinaison) [23]. Par ailleurs, cette protéine est localisée sur les chromosomes méiotiques. La fonction de BLM au cours de la méiose reste à déterminer. Une hypothèse intéressante est, qu’en prophase précoce, les homologues de MutS et de MutL en association avec BLM jouent un rôle d’« antirecombinaison » afin d’éviter des échanges illégitimes entre ADN non homologues au cours de la méiose.
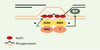 | Figure 3. Hypothèse concernant la fonction des homologues de MutS et de MutL en prophase précoce de méiose. L’hétérodimère MSH4/MSH5 associé à un hétérodimère d’homologues de MutL, comprenant PMS2, pourrait se retrouver au niveau de l’hétéroduplex formé après l’invasion de brin afin de vérifier l’homologie de l’intermédiaire de recombinaison. Dans le cas où des mésappariements seraient détectés, ces protéines pourraient stopper le processus de recombinaison en bloquant l’activité recombinase de Rad51 ou inverser le processus en stimulant la protéine BLM. |
(→) m/s 2002, n°12, p. 1178
|
Une grande partie des connaissances portant sur les mécanismes de recombinaison méiotique reposent sur des analyses génétiques faites chez Saccharomyces cerevisiae. Bien que ces mécanismes et les protéines y participant semblent très conservés au cours de l’évolution, des données récentes de la littérature soulignent des différences notables en fonction de l’organisme étudié. Pour améliorer notre connaissance de la recombinaison méiotique chez les mammifères, il paraît donc indispensable de caractériser la fonction des protéines participant directement à ce processus moléculaire. Les modèles murins suggèrent qu’un défaut d’expression des protéines MSH4, MSH5, PMS2, MLH1 ou MLH3 pourrait être responsable de certains cas de stérilité chez l’homme. Si des progrès ont été réalisés dans le domaine de la procréation médicalement assistée (PMA), il semble maintenant essentiel de mieux cerner les causes des stérilités, notamment en recherchant des mutations dans les homologues de MutS et de MutL chez des patients stériles. L’implication avérée de gènes tels que MLH1 ou PMS2 dans des prédispositions à des cancers soulève une fois de plus la notion de risques génétiques potentiels liés à l’utilisation de méthodes de PMA telles que l’ICSI (intra-cytoplasmic sperm injection). |
1. Roeder GS. Meiotic chromosomes: it takes two to tango. Genes Dev 1997; 11: 2600–21. 2. Mahadevaiah SK, Turner JM, Baudat F, et al. Recombinational DNA double-strand breaks in mice precede synapsis. Nat Genet 2001; 27: 271–6. 3. Kolodner RD, Marsischky GT. Eukaryotic DNA mismatch repair. Cur Opin Genet Dev 1999; 9: 89–96. 4. Peltomaki P. Deficient DNA mismatch repair: a common etiologic factor for colon cancer. Hum Mol Genet 2001; 7: 735–40. 5. Borts HR, Chambers SR, Abdullah MMF. The many faces of mismatch repair in meiosis. Mut Res 2000; 451: 129–50. 6. Baker SM., Bronner CE, Zhang I, et al. Male mice defective in the DNA mismatch repair gene PMS2 exhibit abnormal chromosome synapsis in meiosis. Cell 1995; 82: 309–20. 7. De Vries SS, Baart EB, Dekker M, et al. Mouse MutS-like protein MSH5 is required for proper chromosome synapsis in male and female meiosis. Genes Dev 1999; 13: 523–31. 8. Kneitz B, Cohen PE, Avdievich E, et al. MutS homolog 4 localization to meiotic chromosomes is required for chromosome pairing during meiosis in male and female mice. Genes Dev 2000; 14: 1085–97. 9. Ross-Macdonald P, Roeder GS. Mutation of a meiosisspecific MutS homolog decreases crossing over but not mismatch correction. Cell 1994; 79: 1069–80. 10. Hollingsworth NM, Ponte L, Halsey C. MSH5, a novel MutS homolog, facilitates meiotic reciprocal recombinaison between homologs in Saccharomyces cervisiae but not in mismatch repair. Genes Dev 1995; 9: 1728–39. 11. Baker SM, Plug AW, Prolla TA, et al. Involvement of mouse Mlh1 in DNA mismatch repair and meiotic crossing over. Nat Genet 1996; 13: 336–42. 12. Wang TF, Kleckner N, Hunter N. Functional specificity of MutL homologs in yeast: evidence for three MLH1- based heterocomplexes with distinct roles during meiosis in recombination and mismatch correction. Proc Natl Acad Sci USA 1999; 96: 13914–9. 13. Pochart P, Woltering D, Hollingsworth NM. Conserved properties between functionally distinct MutS homologs in yeast. J Biol Chem 1997; 272: 30345–9. 14. Paquis-Flucklinger V, Santucci-Darmanin S, Paul R, Saunières A, Turc-Carel C, Desnuelle C. Cloning and expression analysis of a meiosis-specific MutS homolog: the human MSH4 gene. Genomics 1997; 44: 188–94. 15. Santucci-Darmanin S, Walpita D, Lespinasse F, Desnuelle C, Ashley T, Paquis-Flucklinger V. MSH4 acts in conjunction with MLH1 during mammalian meiosis. FASEB J 2000; 14: 1539–47. 16. Lipkin SM, Wang V, Jacoby R, et al. MLH3: a DNA mismatch repair gene associated with mammalian microsatellite instability. Nat Genet 2000; 24: 27–35 17. Santucci-Darmanin S, Neyton S, Lespinasse F, Saunières A, Gaudray P, Paquis-Flucklinger V. The DNA mismatch-repair MLH3 protein interacts with MSH4 in meiotic cells, supporting a role for this MutL homolog in mammalian meiotic recombination. Hum Mol Genet 2002; 11 : 1–10. 18. Lipkin SM, Moens PB, Wang V, et al. Meiotic arrest and aneuploidy in MLH3- deficient mice. Nat Genet 2002; 31: 385–90. 19. Shinohara A, Ogawa T. Rad51/RecA families and the associated proteins in eukaryotes. Mutat Res 1999; 435: 13–21. 20. Tarsounas M, Morita T, Pearlman RE, Moens PB. RAD51 and DMC1 form mixed complexes associated with mouse meiotic chromosome cores and synaptonemal complexes. J Cell Biol 1999; 147: 207–19. 21. Radman M. Mismatch repair and the fidelity of genetic recombination. Genome 1989; 31: 68–73. 22. Worth LJR, Clark S, Radman M, Modrich P. Mismatch repair proteins MutS and MutL inhibit RecAcatalyzed strand transfer between diverged DNAs. Proc Natl Acad Sci USA 1994; 91: 3238–41. 23. Karow JK, Constantinou A, Li JL, West SC, Hickson ID. The Bloom’s syndrome gene product promotes branch migration of Holliday junctions. Proc Natl Acad Sci USA 2000; 97: 65504–8. 24. Schofield MJ, Nayak S, Scott TH, Du C, Hsieh P. Interaction of Escherichia coli MutS and MutL at a DNA mismatch. J Biol Chem 2001; 276: 28291–9. |