| |
| Med Sci (Paris). 41(1): 53–61. doi: 10.1051/medsci/2024190.Les humains ont-ils coévolué avec le microbiote intestinal ? Florent Mazel1* 1Département de microbiologie fondamentale, université de Lausanne
,
Lausanne
,
Suisse |
Vignette (© Philippe Sansonetti).
Le riche écosystème de microbes (bactéries, archées, micro-eucaryotes et virus) de notre système digestif, aussi appelé microbiote intestinal, joue un rôle physiologique important. Des bactéries du genre
Roseburia
colonisent notre côlon et produisent, à partir des fibres alimentaires, des acides gras à chaîne courte, comme le butyrate, qui fournissent en énergie les cellules de la paroi intestinale. Plus généralement, la structure écologique du microbiote (sa diversité et sa composition en espèces, et ses fonctions) est souvent associée à la santé, et une dysbiose
1
du microbiote est parfois liée à certaines maladies, notamment immunitaires ou métaboliques.
Les origines de ces « déséquilibres » microbiens sont probablement multiples. Par exemple, le changement brutal de régime alimentaire ou la prise élevée d’antibiotiques sont souvent incriminés. Afin de mieux saisir l’ampleur et l’origine de ces modifications de structure microbienne, il convient de replacer la diversité du microbiote humain dans un contexte plus général en le comparant à nos plus proches parents : les autres espèces de primates, mais aussi à des populations humaines anciennes (
Encadré 1
et
Figure 1
). Ces comparaisons suggèrent que la structure du microbiote humain a subi de grands changements assez récemment, notamment dans les populations dites « industrialisées
2
» et probablement en lien avec la modification du mode de vie au cours de l’anthropocène (
Encadré 1
).
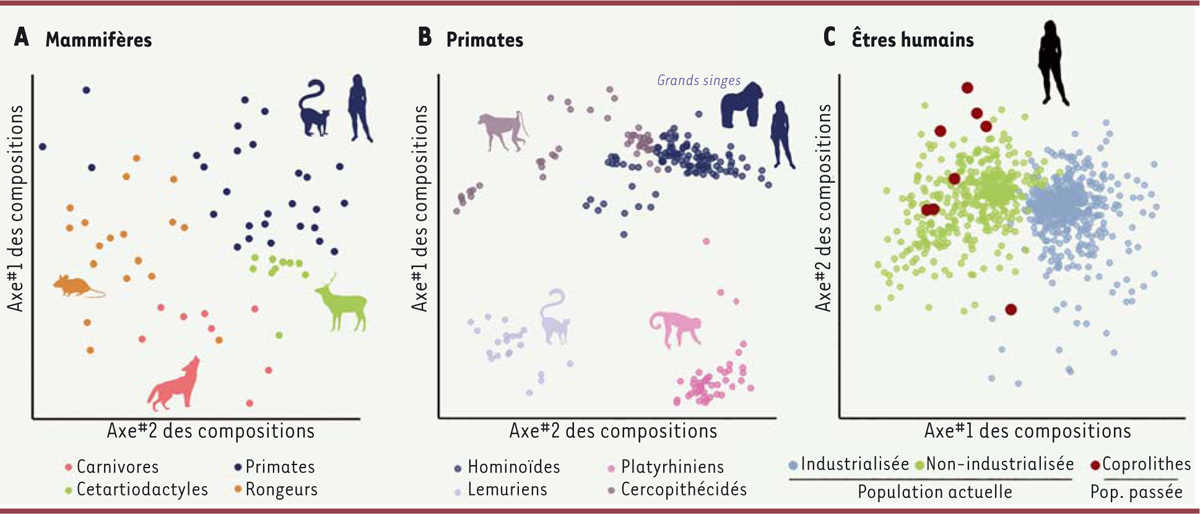
 | Figure 1.
La diversité du microbiote humain dans son contexte évolutif
.
Les différents panneaux sont des représentations multidimensionnelles de la composition des microbiotes. Chaque point représente un échantillon fécal pour lequel l’ADN est extrait, amplifié et séquencé (gène de l’ARN ribosomal 16S ou ADN total) afin d’inférer la composition en espèces bactériennes du microbiote. Les compositions sont ensuite comparées entre échantillons à l’aide d’une analyse de béta-diversité (indice de Jaccard). Sur les graphes, deux échantillons sont d’autant plus proches que les deux microbiotes qu’ils représentent partagent un grand nombre d’espèces bactériennes. (Sources des données 16S : panneau A [
3
,
4
], panneau B [
5
]. Silhouettes tirées de phylopic.org. Source des données métagénomiques : panneau C [
6
]. Code informatique et données pour reproduire le résultat disponible à
https://github.com/FloMazel/Human_microbiome_evolution
).
|
|
La diversité du microbiote humain au sein des mammifères
Bien que la diversité du microbiote intestinal varie au cours de la vie et entre individus d’une même espèce animale, cette variabilité reste faible comparée à celle observée entre espèces, l’être humain ne faisant pas exception [
3
]. C’est ainsi que le microbiote humain est différent de celui des autres primates (
Figure 1B
), eux-mêmes bien différents des autres mammifères (
Figure 1A
) [
8
] (
→
).
(→) Voir la Nouvelle de M. Groussin et F. Mazel,
m/s
n° 12, décembre 2017, page 1038
Pour inférer les changements de diversité au cours de l’évolution humaine (par exemple perte/gain d’espèces bactériennes), deux méthodes sont possibles : observer directement les microbiotes du passé en étudiant des fossiles ou en comparant les microbiotes de différentes populations ou espèces contemporaines.
Approche comparative
: La comparaison du microbiote de plusieurs hominidés (chimpanzés, bonobos, diverses populations humaines, gorilles) suggère que la composition du microbiote humain, et notamment celui des populations dites « industrialisées » a beaucoup changé depuis notre divergence avec le chimpanzé il y a environ 7-8 millions d’années [
9
,
10
]. Par exemple, des archées du genre
Methanobrevibacter
qui sont connues pour dégrader les polysaccharides complexes d’origine végétale, semblent avoir diminué en abondance. À l’inverse, d’autres ont pu augmenter, comme certaines bactéries du genre
Bacteroides
, qui sont parfois associées à un régime alimentaire riche en graisses et protéines animales. Par ailleurs, il est possible que l’être humain ait récemment acquis des bactéries capables de digérer la cellulose et provenant d’autres primates et de ruminants (par exemple, certaines espèces du genre
Ruminococccus
) mais qu’elles aient ensuite été perdues dans les populations industrialisées [
11
].
Approche archéologique
: La façon la plus directe de sonder notre passé microbien est d’étudier les fossiles bactériens et leur ADN ancien, comme ceux présents dans les coprolithes (fossiles de selles) vieux de 1 000 à 2 000 ans retrouvés dans le sud des États-Unis et du Mexique [
6
]. Cette approche a montré que les populations natives de cette région du monde hébergeaient un microbiote plus similaire à celui des populations non-industrialisées contemporaines qu’aux populations contemporaines (
Figure 1C
).
Ainsi, il semblerait que la composition du microbiote soit relativement « plastique » au cours des temps géologiques, avec des gains et des pertes de membres spécifiques et de fonction du microbiote. Ces changements de composition pourraient se faire en réponse à des changements environnementaux, notamment une modification du régime alimentaire.
L’évolution bactérienne : définition et processus élémentaires L’évolution est un phénomène de transformation des caractéristiques héréditaires des populations biologiques sur plusieurs générations. Elle est due à une survie et une reproduction différentes entre les individus d’une population diverse (abritant des individus aux caractères variés). Cela a pour conséquence un changement progressif du caractère moyen de la population au cours des générations parce que certains caractères augmentent en fréquence (se « répandent ») au cours de générations. La diversité initiale des caractéristiques (héréditaires) de la population naît de mutations aléatoires du patrimoine génétique tandis que la survie et la reproduction différentielles des individus peuvent être dictées par des facteurs extérieurs (on parle alors de sélection) ou être aléatoire (on parle de dérive génétique). Par ailleurs, la migration de nouveaux mutants peut également influencer les dynamiques évolutives tout comme le transfert horizontal de gènes, très présent chez les bactéries. On peut estimer le nombre de mutations qui apparaissent chaque jour dans les bactéries intestinales d’un seul individu humain selon le calcul suivant : Nombre de mutations / jour = taille de population × taux de mutation × taux de réplication × taille du génome
On suppose que le nombre de bactéries dans l’intestin (toutes espèces confondues) est d’environ 10
12
– 10
14
, que le taux de mutation est d’environ 10
-9
– 10
-10
mutations/nucléotide/réplication, que le nombre de réplication par jour se situe entre 1 et 10 et que la taille d’un génome bactérien est d’environ 10
6
nucléotides. En effectuant une estimation conservatrice pour le nombre de mutants par jour, on obtient :
10
12
× 10
-10
× 1 × 10
6
= 100 millions de mutations / jour [
12
].
|
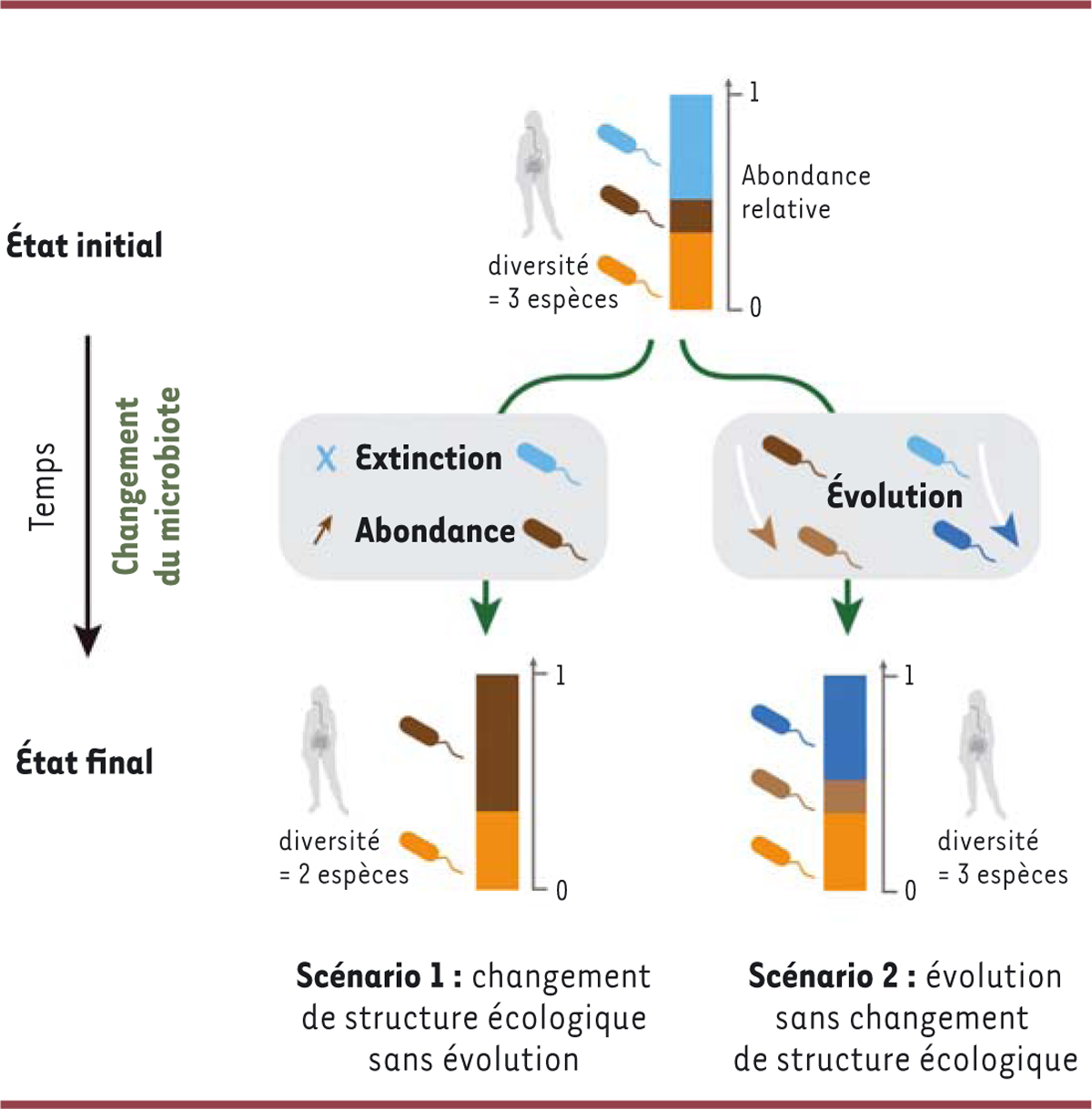
La compréhension de ces changements de structure est importante mais elle reste néanmoins incomplète. La notion de changement de structure écologique considère uniquement les modifications d’abondance, de composition ou de richesse du microbiote, par exemple, la perte ou la diminution en abondance de certaines espèces de bactéries (scénario 1 de la
Figure 2
), mais elle ignore cependant une propriété essentielle du vivant : l’évolution des espèces (
Encadré 2
, scénario 2 de la
Figure 2
). L’évolution de certaines bactéries ne modifie pas nécessairement la structure écologique du microbiote (pas de changement de diversité, par exemple) tout en ayant des conséquences importantes pour l’hôte (
Figure 2
). Par exemple, l’évolution de la résistance aux antibiotiques des bactéries pathogènes est un sujet de préoccupation majeur en santé publique [
1
] (
→
).
(→) Voir l’Éditorial de L. Gutmann et O. Lortholary,
m/s
n° 11, novembre 2010, page 895
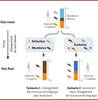 | Figure 2.
Deux scénarios de changement du microbiote
.
La Figure présente un microbiote hypothétique initial qui évolue vers un état final au fil du temps selon deux scénarios distincts : changement d’abondance de bactéries déjà existantes ou évolution de nouvelles bactéries. Les deux processus ne sont pas mutuellement exclusifs et peuvent interagir [
7
].
|
Même s’il est bien établi que les bactéries ne sont pas des entités statiques mais dynamiques, qui peuvent s’adapter très rapidement à des changements environnementaux, cette capacité d’adaptation reste peu étudiée pour nos bactéries intestinales. À quelle vitesse ces bactéries évoluent-elles ? Cela a-til une importance pour notre santé ? Un calcul simple (
Encadré 2
) permet d’estimer que, chaque jour, chez chacun d’entre nous, plusieurs millions de nouvelles mutations apparaissent dans le microbiote intestinal. Si même une infime proportion de ces mutations confère aux microbes qui les portent assez d’avantages en termes de survie et/ou de reproduction, une évolution rapide des populations est attendue. Mais qu’en est-il ?
Dans cet article, nous présentons les preuves de l’évolution des bactéries intestinales, ainsi que ses causes et ses conséquences. L’évolution très récente (microévolution, au sein d’un seul individu) (
Figure 2A
) sera distinguée de l’évolution plus ancienne (macroévolution, de nos ancêtres partagés avec les autres grands singes et les populations humaines) (
Figure 2B
). Les problématiques suivantes guideront cette synthèse :
-
Microévolution :
À une échelle de temps restreinte (par exemple, quelques semaines), est-il possible que les bactéries intestinales évoluent génétiquement et s’adaptent au sein même de chaque individu ? Quelles en sont les conséquences ?
-
Macroévolution :
À une échelle de temps plus longue, est-il possible que les bactéries aient évolué de façon « fidèle » avec les primates et les êtres humains depuis des millénaires (c’est-à-dire en se diversifiant en parallèle de leurs hôtes) ? Cela a-t-il contribué à notre propre adaptation et a notre évolution (coévolution
3
[
2
])?
|
Évolution rapide du microbiote intestinal au sein des individus Les preuves de l’évolution du microbiote intestinal
L’observation en temps réel de l’évolution représente la preuve la plus directe et la plus convaincante de son existence. Des expériences d’évolution expérimentale
4
de microbiotes simplifiés chez la souris (ne comportant qu’une seule espèce de bactérie) ont montré que certaines espèces bactériennes évoluent rapidement au sein même d’un seul individu : c’est par exemple le cas
d’Escherichia coli
ou de
Bacteroides thetaiotaomicron
[
13
-
15
]. Dans ces expériences, certaines nouvelles mutations se répandent rapidement dans les populations (en quelques jours/semaines, soit 10 à 1 000 générations bactériennes) et modifient les caractères des populations en remplaçant parfois les souches ancestrales (
Figure 3A
). L’évolution n’est pas restreinte à ces cas simplifiés, mais elle s’observe aussi lorsque plusieurs espèces bactériennes coexistent au sein d’une même souris de laboratoire [
16
]. De nouvelles techniques de «
barcoding
» moléculaire en cours de développement permettent de suivre précisément des populations au sein d’un même microbiote. Elles apporteront sans doute, dans un futur proche, de précieuses indications sur la vitesse de ces évolutions [
15
].
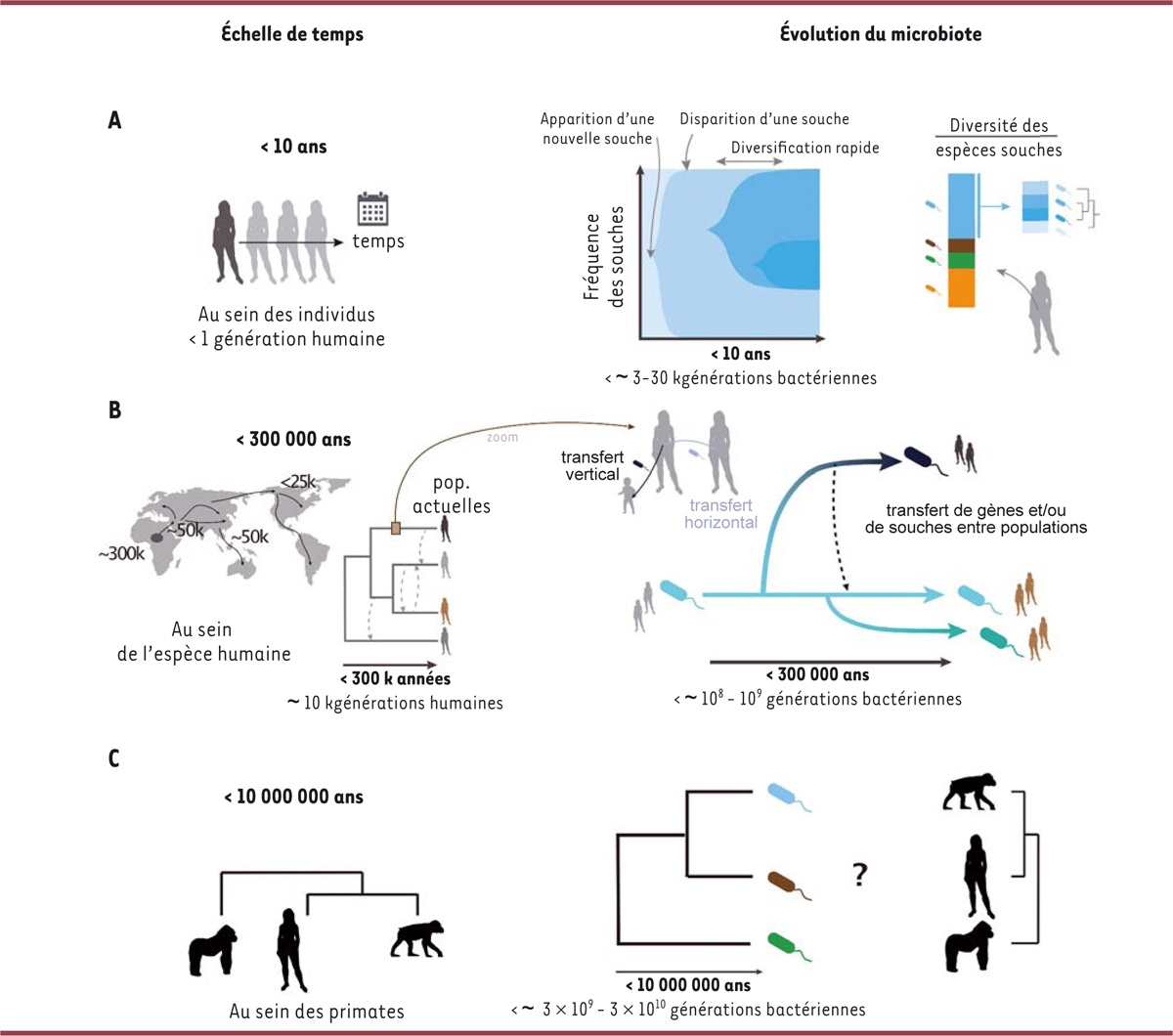
 | Figure 3.
Les différentes échelles de temps de l’évolution du microbiote
.
La Figure décompose l’évolution du microbiote en 3 échelles de temps (lignes). L’échelle de temps est présentée à gauche et l’évolution du microbiote correspondante à droite. Les dates indiquées donnent un ordre de grandeur mais ne sont pas à prendre à la lettre. La représentation des migrations des populations humaines (panneau B) est hautement simplifiée. Le lecteur pourra se référer à des travaux spécialisés pour plus de précisions. La représentation en phylogénie (avec mélange des populations représenté par les flèches grises) des populations humaines est illustrative. On suppose que le nombre de génération des bactéries intestinales se situe entre 1 et 10 par jour.
|
L’évolution d’une espèce de bactérie intestinale au sein d’un seul individu humain est plus compliquée à démontrer car il est difficile d’exclure que l’apparition d’une nouvelle souche génétique n’est pas due à l’immigration à partir d’autres individus. Cependant, en cultivant et en séquençant le génome de 602 souches de
Bacteroides fragilis
de 12 individus humains durant plusieurs années, une étude pionnière a montré qu’à partir d’une seule population initiale, les populations de
Bacteroides fragilis
ont évolué et se sont diversifiées au sein des individus [
12
]. Ces observations semblent se confirmer pour d’autres espèces, notamment certaines Bifidobactéries [
17
,
18
].
Cette évolution est-elle adaptative (
Encadré 2
) ? En d’autres termes, permet-elle aux populations bactériennes de s’adapter à des changements environnementaux
via
la sélection naturelle ? Deux types de données nous permettent de dire que c’est le cas pour certaines bactéries.
Premièrement, certaines des mutations, retrouvées dans les études chez l’homme ou en évolution expérimentale chez la souris, présentent trois caractéristiques qui suggèrent une évolution adaptative et non aléatoire (
Encadré 2
) :
-
Leur position
: elles sont retrouvées indépendamment dans les mêmes gènes de différentes lignées bactériennes (convergence ou parallélisme évolutif),
-
Leur dynamique
: elles augmentent très rapidement en fréquence dans les populations (alors qu’un modèle neutre prédit une augmentation beaucoup plus lente),
-
Leur nature
: elles tendent à modifier les protéines qui dérivent des gènes dans lesquels elles se trouvent.
Deuxièmement, les études d’évolution expérimentale montrent que les souches prélevées à la fin de l’expérience (qui ont « évolué ») surpassent les souches prélevées au début de l’expérience et conservées au congélateur (qui sont « ancestrales ») quand elles sont co-implantées chez les souris. Si l’évolution était non adaptative, les souches évoluées ne devraient pas présenter d’avantages compétitifs.
Face à quelle(s) contrainte(s) s’adaptent les bactéries intestinales ? En d’autres termes, quelles sont les causes, et notamment la nature, des pressions de sélection
5
de cette évolution adaptative ? Les fonctions des gènes mutés dans les études précédentes suggèrent que le régime alimentaire, la pression du système immunitaire ou la pression d’infection par des virus (bactériophages) ont pu jouer un rôle important. Cependant, des expériences plus contrôlées, notamment chez la souris, sont nécessaires pour mieux distinguer et quantifier ces pressions de sélection.
Causes de l’évolution rapide du microbiote intestinal Pressions environnementales
Alors que le régime alimentaire est connu pour moduler la diversité et la structure du microbiote, son impact sur l’évolution génétique des espèces bactériennes est moins clair. En comparant des populations de l’espèce
Bacteroides thetaiotaomicron
dans des souris soumises à deux régimes alimentaires distincts (un régime « standard » et un régime de type « occidental », riche en sucres simples), une étude a récemment montré que le régime alimentaire peut contrôler l’évolution des bactéries intestinales [
19
]. Les populations soumises à un régime « standard » ont, par exemple, accumulé des mutations qui pourraient promouvoir la digestion de fibres alimentaires, tandis que celles soumises à un régime « occidental » ont accumulé des mutations liées à la digestion du mucus produit par la souris. En mettant en compétition dans les mêmes souris les populations « évoluées » et les populations « ancestrales », il apparaît que ces mutations sont adaptatives et permettent aux lignées qui les portent d’augmenter en fréquence dans les populations.
Il est également probable que l’administration d’antibiotiques, au-delà de leurs profonds effets sur la structure du microbiote (notamment une forte diminution de la diversité), promeut l’évolution de la résistance aux antibiotiques au sein du microbiote intestinal [
20
-
22
]. D’autres pressions sélectives, comme le contexte métabolique et les autres membres du microbiote, pourraient aussi jouer un rôle mais sont encore peu explorées [
23
].
Transferts horizontaux de gènes
Jusqu’à présent, seule l’apparition de nouveautés génétiques,
via
les mutations spontanées de nucléotides le long des génomes, a été considérée. Cependant, un autre mécanisme fondamental chez les bactéries peut promouvoir la nouveauté génétique : le transfert horizontal de gènes
6
au sein et entre espèces bactériennes. Ce mécanisme est très répandu chez les bactéries et peut, notamment, permettre aux bactéries de s’adapter rapidement aux antibiotiques. Les bactéries intestinales ne semblent pas faire exception à la règle, car de nombreux transferts de gènes entre bactéries intestinales ont été détectés dans le cas des évolutions expérimentales chez la souris, mais aussi chez l’être humain, notamment entre membres du même microbiote, et depuis des bactéries non humaines vers des bactéries humaines [
24
-
27
]. Les conséquences de ces transferts et de l’évolution des bactéries intestinales en général sur notre santé restent cependant encore incertaines.
Les conséquences sur la santé
Il est bien établi que l’évolution très récente des agents pathogènes au sein d’un seul individu (mais aussi dans la population en général) a parfois des conséquences négatives, comme, par exemple, l’évolution de la résistance aux antibiotiques qui complique le traitement d’une infection [
28
]. Pourtant, ce phénomène est encore méconnu pour les bactéries intestinales, et il n’est pas évident de déterminer si leur évolution est en général bénéfique, défavorable ou neutre pour la santé de leur hôte.
Quelques études montrent que l’évolution d’une bactérie intestinale au sein d’un individu peut être défavorable à l’hôte. Ainsi, des études d’évolution expérimentale chez la souris ont montré que certaines espèces présentes dans les intestins, comme
Escherichia coli
ou
Enterococcus gallinarum
, peuvent évoluer et se diversifier rapidement (de l’ordre de quelques semaines) et devenir potentiellement pathogènes. Elles peuvent notamment envahir le foie ou présenter des caractéristiques similaires à celles de souches naturelles associées aux maladies inflammatoires de l’intestin chez l’être humain (comme la maladie de Crohn) [
29
,
30
]. Ces résultats impliquent que l’évolution ne peut probablement pas être négligée quand il s’agit de mieux comprendre et de manipuler, par exemple à des fins thérapeutiques, les microbiotes.
Il apparaît donc que les bactéries intestinales évoluent de façon adaptative sur des périodes très courtes au sein même d’un individu, par exemple en réponse à des changements de régime alimentaire. Nous n’avons cependant pas évalué le devenir évolutif de ces bactéries sur des temps longs, sur plusieurs générations et même sur des millions d’années. |
Évolution du microbiote intestinal sur des temps longs
Comment l’évolution des bactéries intestinales sur des temps courts se maintient-t-elle sur des temps longs, par exemple plusieurs dizaines d’années et au fil des générations humaines (
Figure 3
) ? A-t-elle permis aux populations humaines de s’adapter à des conditions nouvelles (coévolution) ?
L’évolution bactérienne sur des temps longs va dépendre du mode de transmission
7
des bactéries entre générations humaines. Certaines bactéries intestinales peuvent être transmises verticalement, de la mère à l’enfant, mais beaucoup sont aussi transmises horizontalement, depuis d’autres individus, notamment ceux qui cohabitent, les échanges de bactéries entre populations humaines étant aussi possibles, bien que plus rares [
31
,
32
]. L’importance relative de la transmission verticale et horizontale dictera à long terme le couplage entre évolution humaine et évolution bactérienne : une transmission généralement plutôt horizontale favorisera, par exemple, un découplage entre évolution humaine et bactérienne.
Évolution sur quelques générations (
Figure 3A
)
L’évolution expérimentale est, ici encore, un outil précieux qui nous confirme que l’évolution au sein des individus peut continuer sur plusieurs générations, comme cela a été démontré chez des souris colonisées avec un microbiote simplifié (12 espèces) et maintenue en isolement pendant 6 ans et 24 générations [
16
]. Cependant, il est possible que cette évolution en isolement ne soit pas complètement représentative de la réalité où la transmission de microbes entre individus de différentes populations est possible. Des études d’évolution expérimentale plus réaliste du point de vue de ces échanges de microbes, qui comparent l’évolution du microbiote intestinal de souris en contexte social ou isolé, ont montré que les échanges entre individus peuvent fortement influencer l’évolution des populations [
33
]. Cependant, la coprophagie de ces souris augmente considérablement le taux de transmission des microbes intestinaux entre individus, et il n’est pas clair si ces conclusions s’appliquent à d’autres animaux non coprophages.
Chez l’être humain, il n’y a pas encore d’étude de suivi des microbiotes sur plusieurs dizaines d’années ; des méthodes indirectes de comparaison des microbes contemporains sont donc nécessaires pour inférer les changements dans un passé relativement lointain. Une étude a ainsi montré que l’évolution au sein des individus est probablement contrebalancée par la transmission depuis d’autres individus au bout de 6 mois, même s’il est possible que des souches de certaines espèces bactériennes puissent persister bien plus longtemps dans le même individu, probablement plusieurs années [
32
,
34
,
35
]. Plus généralement, il semble que les échanges de souches de certaines espèces bactériennes soient communs entre individus d’une même population mais aussi entre populations [
32
]. Si ces échanges inter-populationnels sont assez fréquents, il est attendu à long terme que l’évolution des bactéries intestinales soit découplée de celle des populations humaines, et que la structure génétique des populations bactériennes soit peu liée à la géographie ou à la structure génétique des populations humaines.
Évolution sur des centaines à des milliers d’années (
Figure 3B
)
Pour inférer les dynamiques évolutives du microbiote sur plusieurs centaines ou milliers d’années, il est possible 1) de directement sonder le passé en étudiant le génome de bactéries anciennes contenu dans les fossiles de selles (
Encadré 1
) ou, 2) d’inférer le passé en étudiant la structure génétique des populations bactériennes contemporaines (méthode dite « comparative »).
La rareté des données génétiques de microbiotes anciens ne permet pas encore d’étudier directement l’évolution sur plusieurs milliers d’années. En revanche, les données de séquençage d’ADN de bactéries intestinales contemporaines commencent à s’accumuler, même si la représentativité des populations humaines reste encore très biaisée vers l’Europe, l’Amérique du Nord et la Chine.
Les populations de certaines espèces bactériennes ont une structure géographique : des populations humaines habitant différents continents hébergent des souches bactériennes distinctes [
36
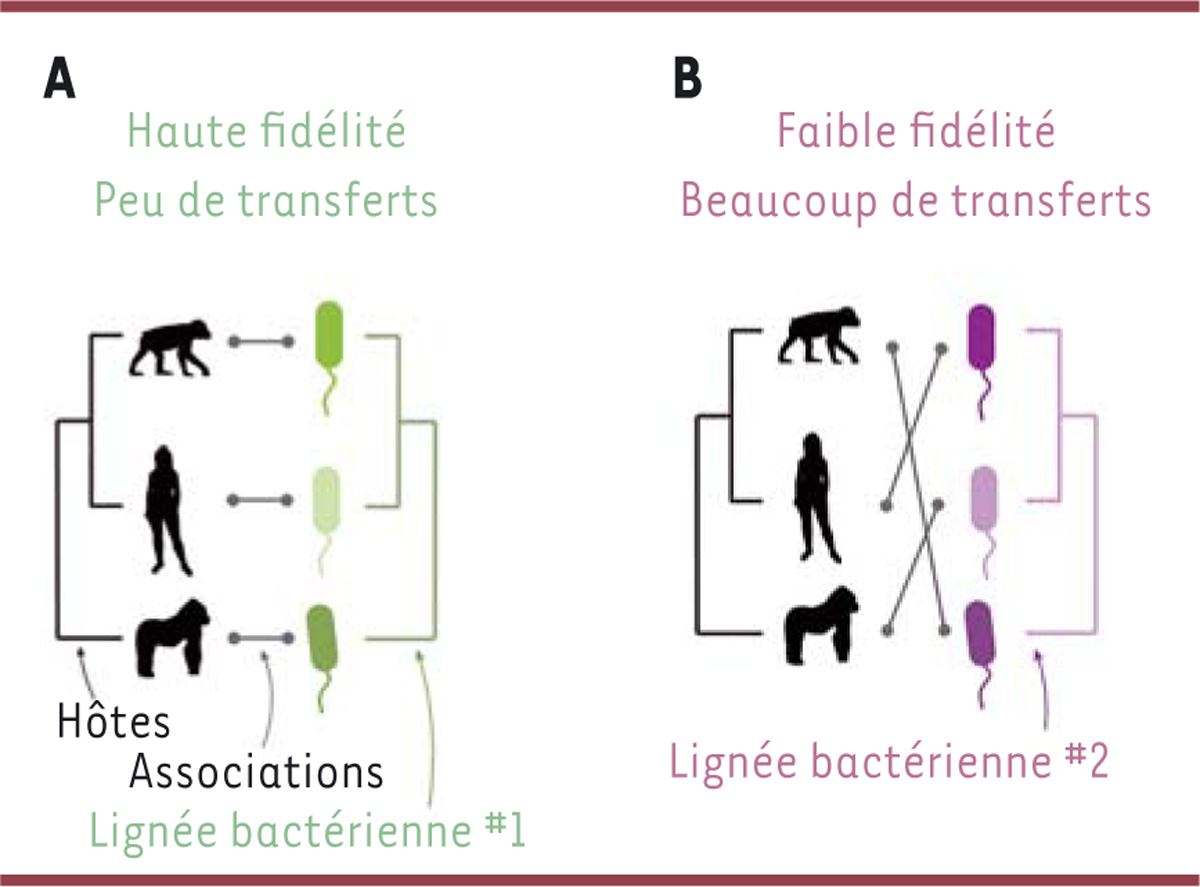
]. Cependant, pour tester si êtres humains et bactéries intestinales ont une histoire commune de « fidélité » ou si, à l’inverse, les associations entre bactéries et hôtes se font et se défont très vite, la méthode la plus pertinente est de comparer leur histoire évolutive représentée par leur phylogénies (
Figure 4
) [
37
]. En comparant les bactéries d’un nombre limité de populations humaines, une étude récente a montré que la plupart des bactéries ne sont pas strictement fidèles aux populations humaines, mais qu’elles ont, au contraire, été beaucoup échangées entre populations par le passé (
Figure 4B
). Certains membres présentent une fidélité historique non nulle avec les êtres humains (36 % des espèces, en moyennant les résultats de plusieurs méthodes) (
Figure 4A
) [
38
]. Ces résultats pourraient éclairer certaines approches thérapeutiques de modification du microbiote. Si les bactéries intestinales ne sont historiquement pas si fidèles à l’être humain, il n’est pas encore clair dans quelle mesure les thérapies microbiennes (par exemple, la confection de probiotiques) devraient utiliser des souches recueillies localement ou si des souches provenant d’autres populations auront un effet similaire.
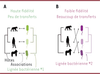 | Figure 4.
Reconstruction de l’histoire évolutive entre animaux et bactéries sur des temps longs
.
La Figure présente la méthode de comparaison des phylogénies des hôtes (phylogénies en noir sur la gauche des panneaux) et celle des bactéries intestinales (phylogénies en couleur sur la droite des panneaux). Une congruence (ou incongruence) des phylogénies (respectivement dans les panneaux A et B) suggère une histoire de fidélité (ou d’échanges).
|
Au cours de leur histoire, les populations humaines se sont adaptées à diverses contraintes environnementales locales, comme l’exposition à des agents pathogènes ou des changements climatiques ou alimentaires. Ces adaptations ont diverses origines et peuvent être génétiques ou culturelles (par exemple, l’apparition de l’agriculture, les outils). Alors que la modulation de la diversité du microbiote ou de l’abondance de certains de ces membres pourrait avoir participé à de telles adaptations [
39
] – par exemple, en facilitant certains changements de régimes alimentaires –, l’implication de l’évolution du microbiote (et donc une coévolution entre êtres humains et bactéries) n’est pas démontrée. Ainsi, l’abondance de certaines espèces de
Ruminococcus
semble être élevée dans les populations adaptées à un régime alimentaire riche en amidon, qui produisent beaucoup d’enzymes salivaires. Dans ces populations, l’amidon est mieux digéré en amont du tube digestif et les fibres non digérées atteignant le microbiote sont métabolisées plus efficacement par les
Ruminococcus
, avec peutêtre un surplus énergétique pour l’hôte. Cependant, la capacité des
Ruminococcus
à digérer les fibres complexes n’est probablement pas récente et préexiste à l’apparition du changement de régime alimentaire humain. Dans cet exemple, c’est donc la modulation de la structure préexistante du microbiote (l’abondance de certains
Ruminococcus
) (
Figure 2
, scénario 1) qui facilite l’adaptation humaine et non pas l’évolution du microbiote (
Figure 2
, scénario 2). Il semble donc trop tôt pour affirmer que l’évolution récente du microbiote humain a joué un rôle majeur dans l’adaptation humaine, mais un exemple suggère cependant que ce n’est pas impossible. Plusieurs membres du microbiote de populations asiatiques, comme au Japon, ont acquis la capacité de digérer des polysaccharides contenus dans certaines algues du régime alimentaire. Cette acquisition résulte d’un transfert horizontal de gènes, probablement assez récent, depuis une bactérie naturellement présente sur les surfaces d’algues vers le microbiote humain, peut-être facilitée par la consommation de ces mêmes algues crues (et de leur bactéries associées) dans certaines populations [
27
].
Il n’est donc pas encore clair si l’évolution récente des bactéries a pu jouer un rôle important dans l’adaptation humaine (« coévolution » au sens strict). Dans tous les cas, il semblerait que l’histoire commune entre êtres humains et bactéries intestinales soit largement marquée par de vastes échanges bactériens entre populations humaines. Cette histoire d’échanges intenses est-elle spécifique à l’être humain ou s’applique-t-elle plus généralement aux primates et à leurs microbiotes, sur des temps évolutifs de plusieurs millions d’années ?
Evolution sur des millions d’années (
Figure 3C
)
Des études de génétique comparative chez les grands singes apportent deux résultats importants : 1) une majorité de bactéries semble effectivement avoir été intensément échangée entre espèces de primates au cours des derniers millions d’années (au moins 75 % environ des bactéries étudiées), mais 2) certaines bactéries, qui montrent une histoire de relative fidélité, semblent avoir récemment disparu dans les sociétés humaines dites « industrielles » [
10
,
40
,
41
]. Ces résultats suggèrent que le principal mode d’évolution sur les temps longs repose sur les échanges de bactéries entre hôtes, et non leur fidèle transmission entre générations (modèle de la
Figure 4B
et non celui de la
Figure 4A
). Cependant, la disparition locale de bactéries « fidèles » à leurs hôtes paraît inquiétante dans l’hypothèse où ces bactéries confèrent des bénéfices à leurs hôtes.
|
La structure et la diversité du microbiote humain sont affectées par les changements de mode de vie des populations industrialisées (
Figure 2
, scénario 1). Par exemple, certaines espèces du microbiote sont moins communes dans ces populations par rapport à celles non industrialisées. Au-delà de ces variations d’abondance, il semblerait que les espèces du microbiote puissent aussi évoluer rapidement (
Figure 2
, scénario 2). Cette évolution génétique pourrait être très rapide au sein même des individus (quelques mois) et semblerait être influencée par des dynamiques adaptatives. Plus généralement, l’évolution du microbiote humain est marquée par des échanges fréquents de gènes et d’espèces entre populations humaines.
De nombreuses questions sur l’évolution du microbiote restent cependant en suspens. Par exemple, quels sont les facteurs qui déterminent ces échanges microbiens fréquents entre populations ? Quelles en sont les conséquences sur la santé humaine ? Dans quelle mesure les adaptations microbiennes et humaines sont-elles couplées à long terme (« coévolution ») ? Les progrès techniques de l’analyse « à haut débit » du microbiote humain permettront dans les prochaines années de commencer à répondre à ces questions. |
Je remercie Simon Yersin, Pascale Vonaesh, Blaise Tymen et Marion Bonnecaze pour leur relecture du manuscrit
.
|
Footnotes |
L’auteur déclare n’avoir aucun lien d’intérêt concernant les données publiées dans cet article
.
|
1.
Gutmann
L
,
Lortholary
O.
Coexister avec la résistance aux antibiotiques : une réalité internationale en 2010.
.
Med Sci (Paris)
.
2010;
;
26
:
:895.
–
6
.
2.
Janzen
DH.
When is it coevolution?
Evolution
.
1980;
;
34
:
:611.
–
2
.
3.
Mazel
F
,
Guisan
A
,
Parfrey
LW.
Transmission mode and dispersal traits correlate with host specificity in mammalian gut microbes.
.
Mol Ecol
.
2024;
;
33
:
:e16862.
.
4.
Song
SJ
,
Sanders
JG
,
Delsuc
F
,
et al.
.
Comparative Analyses of Vertebrate Gut Microbiomes Reveal Convergence between Birds and Bats.
.
mBio
.
2020;
;
11
:
:e02901.
–
19
.
5.
Amato
KR
,
Mallott
EK
,
McDonald
D
,
et al.
.
Convergence of human and Old World monkey gut microbiomes demonstrates the importance of human ecology over phylogeny.
.
Genome Biol
.
2019;
;
20
:
:201.
.
6.
Wibowo
MC
,
Yang
Z
,
Borry
M
,
et al.
.
Reconstruction of ancient microbial genomes from the human gut.
.
Nature
.
2021;
;
594
:
:234.
–
9
.
7.
Good
BH
,
Rosenfeld
LB.
Eco-evolutionary feedbacks in the human gut microbiome.
.
Nat Commun
.
2023;
;
14
:
:7146.
.
8.
Groussin
M
,
Mazel
F.
Impact of the evolution of mammalian intestinal microbiotas on human health.
.
Med Sci (Paris)
.
2017;
;
33
:
:1038.
–
42
.
9.
Moeller
AH
,
Li
Y
,
Mpoudi
Ngole E
,
et al.
.
Rapid changes in the gut microbiome during human evolution.
.
Proc Natl Acad Sci U S A
.
2014;
;
111
:
:16431.
–
5
.
10.
Rühlemann
MC
,
Bang
C
,
Gogarten
JF
,
et al.
.
Functional host-specific adaptation of the intestinal microbiome in hominids.
.
Nat Commun
.
2024;
;
15
:
:326.
.
11.
Moraïs
S
,
Winkler
S
,
Zorea
A
,
et al.
.
Cryptic diversity of cellulose-degrading gut bacteria in industrialized humans.
.
Science
.
2024;
;
383
:
:eadj9223.
.
12.
Zhao
S
,
Lieberman
TD
,
Poyet
M
,
et al.
.
Adaptive Evolution within Gut Microbiomes of Healthy People.
.
Cell Host Microbe
.
2019;
;
25
:
:656.
–
67.e8
.
13.
Vasquez
KS
,
Willis
L
,
Cira
NJ
,
et al.
.
Quantifying rapid bacterial evolution and transmission within the mouse intestine.
.
Cell Host Microbe
.
2021;
;
29
:
:1454.
–
68.e4
.
14.
Frazão
N
,
Konrad
A
,
Amicone
M
,
et al.
.
Two modes of evolution shape bacterial strain diversity in the mammalian gut for thousands of generations.
.
Nat Commun
.
2022;
;
13
:
:5604.
.
15.
Vasquez
KS
,
Wong
DPGH
,
Pedro
MF
,
et al.
.
High-resolution lineage tracking of within-host evolution and strain transmission in a human gut symbiont across ecological scales.
.
bioRxiv
.
2024
;
2024.02.17.580834.
.
16.
Yilmaz
B
,
Mooser
C
,
Keller
I
,
et al.
.
Long-term evolution and short-term adaptation of microbiota strains and sub-strains in mice.
.
Cell Host Microbe
.
2021;
;
29
:
:650.
–
63.e9
.
17.
Poyet
M
,
Groussin
M
,
Gibbons
SM
,
et al.
.
A library of human gut bacterial isolates paired with longitudinal multiomics data enables mechanistic microbiome research.
.
Nat Med
.
2019;
;
25
:
:1442.
–
52
.
18.
Chen
DW
,
Garud
NR.
Rapid evolution and strain turnover in the infant gut microbiome.
.
Genome Res
.
2022;
;
32
:
:1124.
–
36
.
19.
Dapa
T
,
Ramiro
RS
,
Pedro
MF
,
et al.
.
Diet leaves a genetic signature in a keystone member of the gut microbiota.
.
Cell Host Microbe
.
2022;
;
30
:
:183.
–
99.e10
.
20.
Fishbein
SRS
,
Mahmud
B
,
Dantas
G.
Antibiotic perturbations to the gut microbiome.
.
Nat Rev Microbiol
.
2023;
;
21
:
:772.
–
88
.
21.
Roodgar
M
,
Good
BH
,
Garud
NR
,
et al.
.
Longitudinal linked-read sequencing reveals ecological and evolutionary responses of a human gut microbiome during antibiotic treatment.
.
Genome Res
.
2021;
;
31
:
:1433.
–
46
.
22.
Nies
L de
,
Busi
SB
,
Tsenkova
M
,
et al.
.
Evolution of the murine gut resistome following broad-spectrum antibiotic treatment.
.
Nat Commun
.
2022;
;
13
:
:1.
–
11
.
23.
Barroso-Batista
J
,
Pedro
MF
,
Sales-Dias
J
,
et al.
.
Specific Eco-evolutionary Contexts in the Mouse Gut Reveal Escherichia coli Metabolic Versatility.
.
Curr Biol
.
2020;
;
30
:
:1049.
–
62.e7
.
24.
Frazão
N
,
Sousa
A
,
Lässig
M
,
et al.
.
Horizontal gene transfer overrides mutation in Escherichia coli colonizing the mammalian gut.
.
Proc Natl Acad Sci U S A
.
2019;
;
116
:
:17906.
–
15
.
25.
Smillie
CS
,
Smith
MB
,
Friedman
J
,
et al.
.
Ecology drives a global network of gene exchange connecting the human microbiome.
.
Nature
.
2011;
;
480
:
:241.
–
4
.
26.
Groussin
M
,
Poyet
M
,
Sistiaga
A
,
et al.
.
Elevated rates of horizontal gene transfer in the industrialized human microbiome.
.
Cell
.
2021;
;
184
:
:2053.
–
67.e18
.
27.
Pudlo
NA
,
Pereira
GV
,
Parnami
J
,
et al.
.
Diverse events have transferred genes for edible seaweed digestion from marine to human gut bacteria.
.
Cell Host Microbe
.
2022;
;
30
:
:314.
–
28.e11
.
28.
Didelot
X
,
Walker
AS
,
Peto
TE
,
et al.
.
Within-host evolution of bacterial pathogens.
.
Nat Rev Microbiol
.
2016;
;
14
:
:150.
–
62
.
29.
Yang
Y
,
Nguyen
M
,
Khetrapal
V
,
et al.
.
Within-host evolution of a gut pathobiont facilitates liver translocation.
.
Nature
.
2022;
;
607
:
:563.
–
70
.
30.
Elhenawy
W
,
Tsai
CN
,
Coombes
BK.
Host-Specific Adaptive Diversification of Crohn’s Disease-Associated Adherent-Invasive
Escherichia coli
.
.
Cell Host Microbe
.
2019;
;
25
:
:301.
–
12.e5
.
31.
Sarkar
A
,
Mclnroy
CJA
,
Harty
S
,
et al.
.
Microbial transmission in the social microbiome and host health and disease.
.
Cell
.
2024;
;
187
:
:17.
–
43
.
32.
Valles-Colomer
M
,
Blanco-Miguez
A
,
Manghi
P
,
et al.
.
The person-to-person transmission landscape of the gut and oral microbiomes.
.
Nature
.
2023;
;
614
:
:125.
–
35
.
33.
Frazão
N
,
Gordo
I.
Shared Evolutionary Path in Social Microbiomes.
.
Mol Biol Evol
.
2023;
;
40
:
:msad153.
.
34.
Garud
NR
,
Good
BH
,
Hallatschek
O
,
et al.
.
Evolutionary dynamics of bacteria in the gut microbiome within and across hosts.
.
PLoS Biol
.
2019;
;
17
:
:e3000102.
.
35.
Hildebrand
F
,
Gossmann
TI
,
Frioux
C
,
et al.
.
Dispersal strategies shape persistence and evolution of human gut bacteria.
.
Cell Host Microbe
.
2021;
;
29
:
:1167.
–
76.e9
.
36.
Enav
H
,
Paz
I
,
Ley
RE.
Strain tracking in complex microbiomes using synteny analysis reveals per-species modes of evolution.
.
Nat Biotechnol
.
2024
;
:1.
–
11
.
37.
Groussin
M
,
Mazel
F
,
Alm
EJ.
Co-evolution and Co-speciation of Host-Gut Bacteria Systems.
.
Cell Host Microbe
.
2020;
;
28
:
:12.
–
22
.
38.
Suzuki
TA
,
Fitzstevens
JL
,
Schmidt
VT
,
et al.
.
Codiversification of gut microbiota with humans.
.
Science
.
2022;
;
377
:
:1328.
–
32
.
39.
Suzuki
TA
,
Ley
RE.
The role of the microbiota in human genetic adaptation.
.
Science
.
2020;
;
370
:
:eaaz6827.
.
40.
Moeller
AH
,
Caro-Quintero
A
,
Mjungu
D
,
et al.
.
Cospeciation of gut microbiota with hominids.
.
Science
.
2016;
;
353
:
:380.
–
2
.
41.
Sanders
JG
,
Sprockett
DD
,
Li
Y
,
et al.
.
Widespread extinctions of co-diversified primate gut bacterial symbionts from humans.
.
Nat Microbiol
.
2023;
;
8
:
:1039.
–
50
.
|