| |
| Med Sci (Paris). 40(11): 819–821. doi: 10.1051/medsci/2024144.Un outil d’intelligence artificielle pour la découverte d’anticorps neutralisants à large spectre contre le virus VIH Mathilde Foglierini1 and Laurent Perez1* 1Département de médicine, Service d’immunologie et allergie, Centre hospitalier universitaire Vaudois (CHUV) et université de Lausanne (UNIL), Faculté de biologie et médecine
,
Lausanne
,
Suisse MeSH keywords: |
On estime à 38 millions le nombre de personnes infectées par le virus de l’immunodéficience humaine de type 1 (VIH-1) [
1
] et à environ 650 000 le nombre de décès liés au syndrome d’immunodéficience acquise (SIDA) en 2023. L’exceptionnelle diversité et rapidité d’évolution du virus constituent des obstacles majeurs à son contrôle par l’immunité adaptative ou au développement d’un vaccin. Cependant, la découverte chez certains individus infectés par le VIH-1, d’anticorps monoclonaux capables de reconnaître et neutraliser de nombreuses souches du virus suscite un grand intérêt scientifique et un important espoir médical. Les anticorps neutralisants à large spectre (
broadly neutralizing antibodies
, bNAb) contre le VIH sont capables de bloquer jusqu’à près de 90 % des isolats viraux connus. L’intérêt thérapeutique pour ces anticorps est double. D’une part, les résultats de récents essais cliniques montrent une réduction de la charge virale et la prévention de l’infection grâce à la vaccination passive par ces bNAb. D’autre part, l’étude structurale de ces anticorps permet la conception de vaccin mimant l’infection naturelle [
2
]. Ces anticorps empêchent l’entrée du virus dans la cellule en se fixant sur la glycoprotéine de l’enveloppe virale (Env). Cette protéine est présente sous forme de trimères à la surface des virions, et elle est constituée de deux sous-unités : gp120, responsable de la liaison au récepteur cellulaire CD4, et gp41, qui assure la fusion entre la membrane virale et la membrane plasmique de la cellule. Cinq sites de vulnérabilité dans les glycoprotéines d’enveloppe du virus ont été identifiés
1
: le site de liaison à CD4 (
CD4-binding site
, CD4bs), les boucles variables V1/V2 et V3 associées aux glycanes, l’interface entre les sous-unités gp120 et gp41 contenant le peptide de fusion (interface), et la région externe proche de la membrane virale de la sous-unité gp41 (
membrane-proximal external region
, MPER) [
3
]. Certaines propriétés particulières des bNAb ont été rapportées en fonction du site de vulnérabilité auquel ils se lient. Par exemple, la séquence nucléotidique codant leur région variable peut comporter un très grand nombre de mutations, et il peut exister de longues insertions de nucléotides dans la troisième région déterminant la complémentarité de la chaîne lourde à l’antigène (CDRH3).
Nous avons montré que les séquences d’acides aminés des bNAb présentent des caractéristiques spécifiques, statistiquement distinctes de celles des autres anticorps [
4
]. Grâce à l’identification et à la comparaison de ces différentes caractéristiques, nous avons développé un nouvel outil d’intelligence artificielle, appelé RAIN (
rapid automatic identification of bNAbs
), capable d’identifier facilement et rapidement les bNAb.
|
Sélection des caractéristiques distinguant les bNAb des anticorps classiques
Les approches bioinformatiques visant à identifier la spécificité des anticorps se sont révélées décevantes jusqu’à présent. En effet, l’efficacité des outils fondés sur l’alignement de séquences multiples à partir de bases de données d’anticorps (par exemple,
Observed Antibody Space
) [
5
] est limitée par la grande variabilité des régions déterminant la complémentarité à l’antigène (
complementary-determining region
, CDR). Par conséquent, nous avons voulu identifier et sélectionner des caractéristiques permettant de distinguer les bNAb des anticorps classiques (non-bNAb). Nous avons d’abord rassemblé les séquences peptidiques de bNAb répertoriées dans la base de données CATNAP (
compile, analyze and tally neutralizing antibody panels
) [
6
], puis nous avons comparé certaines de leur propriétés avec celles des anticorps provenant d’individus séronégatifs pour le VIH. En effet, outre leurs séquences, certaines propriétés des anticorps peuvent également être quantifiées, telles que la longueur des régions CDRH3 et CDRL3 (
i.e
., les troisièmes CDR de la chaîne lourde et de la chaîne légère des immunoglobulines, respectivement), leur hydrophobicité, la fréquence des mutations hypersomatiques
2,
ou le nombre de mutations dites « improbables » (c’est-à-dire présentes dans d’autres régions que les CDR). Nous avons ensuite utilisé ces caractéristiques quantifiables pour une analyse en composantes principales
3
, qui a montré qu’elles permettent de distinguer efficacement les bNAb des anticorps classiques, et peuvent donc servir de variables prédictives.
|
Formation de modèles d’apprentissage automatique pour la reconnaissance de bNAb
Dans un second temps, nous avons entraîné et testé plusieurs algorithmes d’intelligence artificielle aux spécifications variées, tels que l’algorithme de détection d’anomalies, les arbres décisionnels, et les forêts d’arbres décisionnels. L’algorithme de détection d’anomalies est couramment utilisé pour identifier des évènements qui se distinguent du reste des données standard. À l’instar des bNAb, qui sont rares au sein d’un répertoire et qui possèdent des caractéristiques différentes des anticorps classiques, de tels évènements peuvent donc être identifiés comme anomalies dans un groupe d’anticorps. Bien qu’efficace, cette méthode produisait un nombre excessif de « faux positifs » (i.e., des anticorps considérés à tort comme étant des bNAb). Nous avons donc renforcé ce procédé de détection en y intégrant d’autres algorithmes de prédiction : arbres décisionnels et forêts d’arbres décisionnels. Les arbres décisionnels sont une méthode d’apprentissage automatique capable de prise de décisions en posant une série de questions, dont les réponses sont limitées à oui ou non. Dans notre modèle, cette succession de questions conduit à une décision finale déterminant si un anticorps est un bNAb ou non. Les forêts d’arbres décisionnels, quant à elles, sont une méthode d’ensemble qui combine les résultats des différents arbres de décision qui les composent. Nous avons ainsi obtenu une classification efficace des anticorps, avec des résultats optimaux (
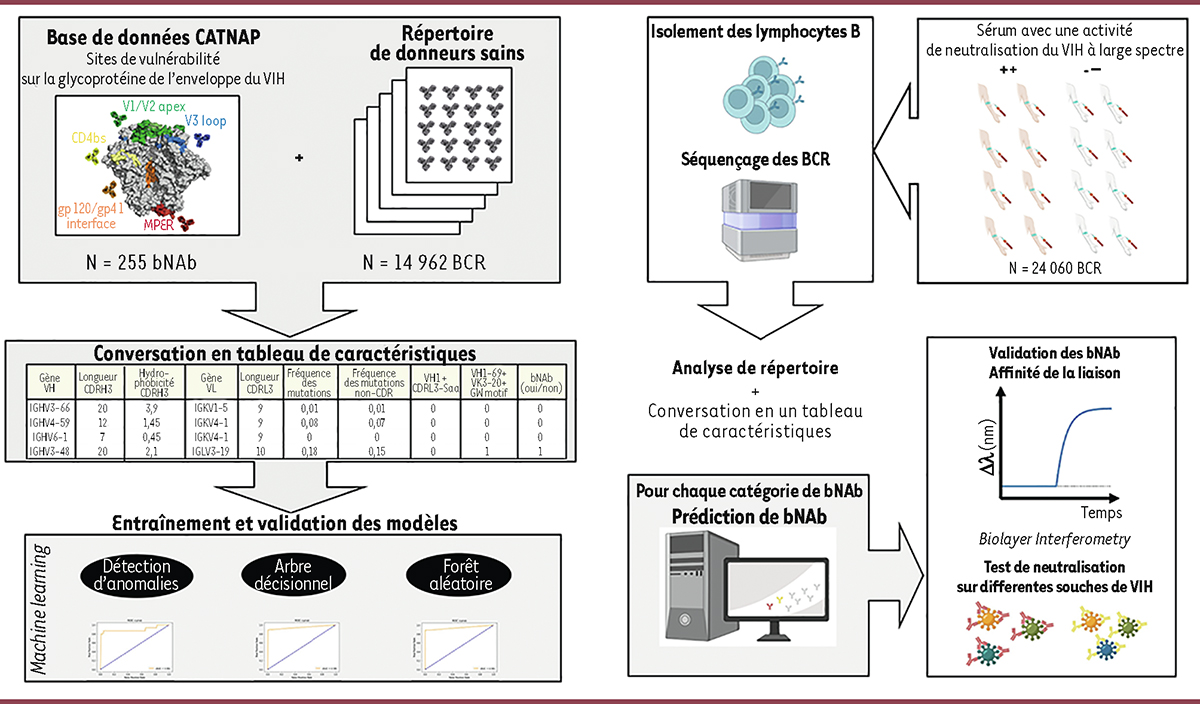
Figure 1
). Encouragés par ces résultats, nous avons souhaité montrer expérimentalement la précision de notre méthode, ce qui nous a conduits à tester RAIN sur de véritables échantillons pour découvrir de nouveaux bNAb.
 | Figure 1.
Méthode de détection RAIN pour l’identification automatique d’anticorps neutralisants à large spectre (bNAb) contre le virus VIH-1
.
Les données collectées à partir de la base de données CATNAP (
broadly neutralizing antibodies
, bNAb) et des répertoires de donneurs sains (
monoclonal antibodies
, mAb) sont converties en un tableau de caractéristiques pour entraîner et valider trois modèles d’apprentissage automatique : détection d’anomalies (AD), arbre décisionnel (DT), et forêt aléatoire (RF) d’arbres décisionnels. Les séquences des récepteurs des lymphocytes B (
B-cell receptors
, BCR) sont déterminées par séquençage nucléotidique sur cellule unique à partir de donneurs séropositifs pour le VIH dont le sérum présentait une activité de neutralisation à large spectre (+, illustré par les bras de couleur chair) ou était dépourvu d’activité de neutralisation (-, illustré par les bras de couleur blanche). Les séquences des BCR sont annotées, analysées, puis converties sous forme d’un tableau de caractéristiques. Ensuite, les bNAb prédits par les trois algorithmes sont produits et soumis à des tests de neutralisation du virus et de liaison au trimère de l’enveloppe virale stabilisé et soluble (SOSIP).
|
|
Identification et caractérisation de nouveaux bNAb
Pour valider les résultats de l’approche informatique, les anticorps sériques de douze individus séropositifs pour le VIH ont été purifiés par chromatographie d’affinité pour la protéine A
4,
, et un test de neutralisation virale, permettant de mesurer la capacité de ces anticorps à bloquer l’entrée du virus dans les cellules TZM-bl
5
, a été effectué avec plusieurs souches virales représentatives de l’épidémie [
7
]. Nous avons constaté une activité neutralisante à large spectre pour les anticorps provenant de deux des douze donneurs, tandis que les anticorps provenant des autres donneurs ne neutralisaient que quelques souches virales. Nous avons donc sélectionné l’un de ces deux donneurs pour valider notre méthode, ainsi que deux autres donneurs comme témoins négatifs. Pour connaître la séquence des anticorps exprimés par les lymphocytes B, nous avons utilisé une technique de séquençage nucléotidique sur cellule unique [
8
]. Nous avons pu alors reconstruire les séquences de 15 713 récepteurs de lymphocytes B (
B-cell receptors
, BCR)
6
pour le donneur d’intérêt, et de 8 347 pour les donneurs témoins. L’analyse par la méthode de détection RAIN a permis d’identifier trois potentiels bNAb chez le donneur d’intérêt, et comme prévu, aucun chez les donneurs témoins. Pour vérifier que ces trois anticorps étaient véritablement des bNAb, nous les avons clonés et exprimés, de même que les anticorps témoins provenant des deux autres donneurs. Les trois bNAb potentiels et les anticorps témoins ont été purifiés par chromatographie d’affinité sur la protéine A, puis nous avons testé leur spécificité par la technique d’interférométrie de bio-couche (
bio-layer interferometry
). Cette méthode a permis de mesurer l’affinité des anticorps pour le trimère de l’enveloppe virale stabilisé et soluble (
homogenous, soluble, stabilized, trimeric version of the HIV-1 envelop glycoprotein
, SOSIP) [
9
]. Les constantes de dissociation (
Kd
) mesurées pour les trois bNAb potentiels témoignaient d’une forte affinité de ces anticorps pour SOSIP, alors qu’aucune interaction n’a pu être mesurée avec les anticorps témoins. Nous avons ensuite réalisé de nouveaux tests de neutralisation du virus sur les cellules TZM-bl avec chacun des trois bNAb potentiels : deux d’entre eux ont montré une activité de neutralisation à large spectre (en particulier, pour l’un, une capacité à neutraliser environ 80 % des différentes souches du VIH à une concentration inférieure à 10 nM), tandis que la capacité de neutralisation du troisième était restreinte à une seule souche du virus. Pour déterminer le mécanisme de neutralisation des deux anticorps neutralisants à large spectre et valider leur site d’interaction avec l’antigène prédit par notre approche informatique, nous avons exploré le site de liaison au SOSIP des fragments Fab (fragment
antigen-binding
) de ces anticorps par microscopie électronique. Nous avons ainsi pu déterminer que ces deux Fab se lient au site de liaison du SOSIP au récepteur cellulaire CD4, comme prédit. Finalement, nous avons analysé, par cryo-microscopie électronique, la structure du complexe formé avec SOSIP par le Fab de l’un de ces anticorps, ce qui a révélé que c’est la région CDRH2 de cet anticorps qui bloque une cavité du trimère de l’enveloppe virale, et empêche ainsi l’interaction du virus avec le récepteur cellulaire CD4.
|
Nous avons développé une nouvelle méthode utilisant l’intelligence artificielle pour identifier des anticorps spécifiques du VIH et possédant une capacité de neutralisation à large spectre. Cette méthode facilitera la découverte de tels anticorps contre le VIH. Notre stratégie offre aussi une alternative aux approches par homologie de séquence, relativement peu efficaces pour identifier la spécificité des anticorps. |
Footnotes |
Les auteurs déclarent n’avoir aucun lien d’intérêt concernant les données publiées dans cet article
.
|
1.
Barré-Sinoussi
F
,
Chermann
JC
,
Rey
F
,
et al.
Isolation of a T-lymphotropic retrovirus from a patient at risk for acquired immune deficiency syndrome (AIDS).
.
Science
.
1983;
;
220
:
:868.
–
71
.
2.
Kwong
PD
,
Mascola
JR.
HIV-1 vaccines based on antibody identification, B cell ontogeny, and epitope structure.
.
Immunity
.
2018;
;
48
:
:855.
–
71
.
3.
Sok
D
,
Burton
DR.
Recent progress in broadly neutralizing antibodies to HIV.
.
Nat Immunol
.
2018;
;
19
:
:1179.
–
88
.
4.
Foglierini
M
,
Nortier
P
,
Schelling
R
,
et al.
RAIN: machine learning-based identification for HIV-1 bNAbs.
.
Nat Commun
.
2024;
;
15
:
:5339.
.
5.
Olsen
TH
,
Boyles
F
,
Deane
CM.
Observed Antibody Space: A diverse database of cleaned, annotated, and translated unpaired and paired antibody sequences.
.
Protein Sci
.
2022;
;
31
:
:141.
–
6
.
6.
Yoon
H
,
Macke
J
,
West
AP
Jr.
,
et al.
CATNAP: a tool to compile, analyze and tally neutralizing antibody panels.
.
Nucleic Acids Res
.
2015;
;
43
:
:W213.
–
9
.
7.
deCamp
A
,
Hraber
P
,
Bailer
RT
,
et al.
Global panel of HIV-1 Env reference strains for standardized assessments of vaccine-elicited neutralizing antibodies.
.
J Virol
.
2014;
;
88
:
:2489.
–
507
.
8.
Hoehn
KB
,
Kleinstein
SH.
B cell phylogenetics in the single cell era.
.
Trends Immunol
.
2024;
;
45
:
:62.
–
74
.
9.
Gulla
K
,
Cibelli
N
,
Cooper
JW
,
et al.
A non-affinity purification process for GMP production of prefusion-closed HIV-1 envelope trimers from clades A and C for clinical evaluation.
.
Vaccine
.
2021;
;
39
:
:3379.
–
87
.
|