Nous avons décrit une plateforme de recherche de nouveaux candidats médicaments (
drug discovery
) biomimétiques de la façon dont les virus détournent les fonctions cellulaires [
3
]. Cette plateforme s’inspire de millions d’années de coévolution des virus avec leurs hôtes ainsi que de dizaines d’années d’études des interactions virus-hôtes par la communauté scientifique internationale.
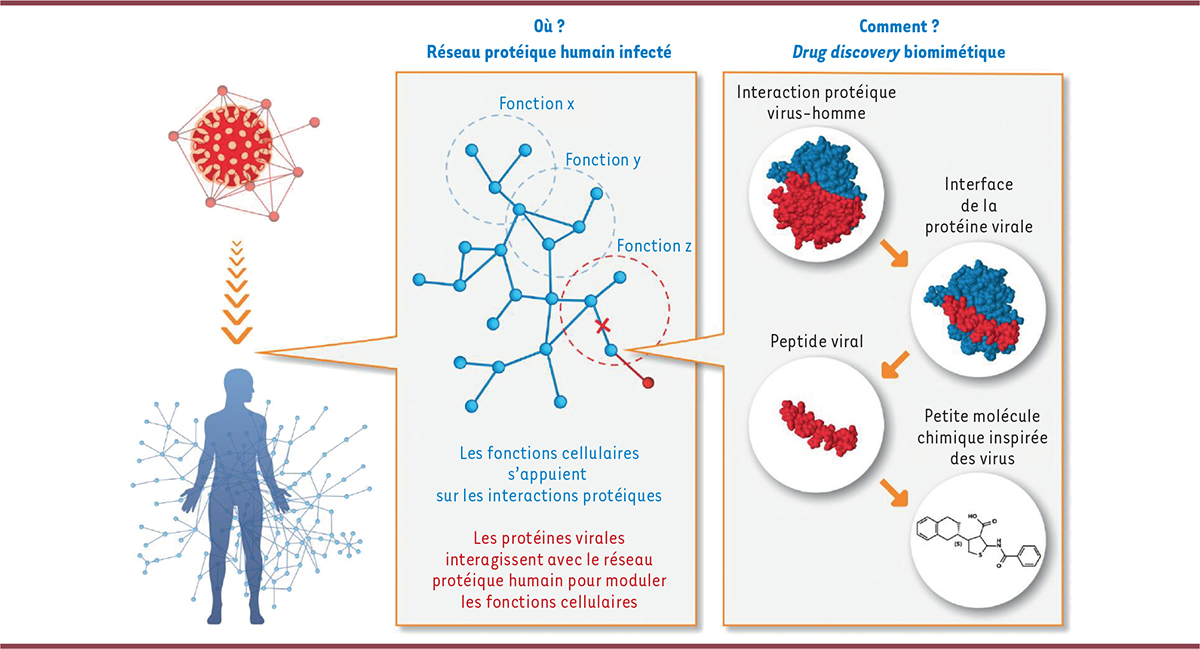
Nous avons tout d’abord établi la plus grande carte d’interactions entre toutes les protéines de tous les virus qui infectent l’homme et toutes les protéines humaines. Cette carte procède d’une fouille méticuleuse de la littérature scientifique et d’une annotation précise des interactions qui y sont décrites. Un « paysage de l’infection virale », formé à ce jour de 100 000 interactions, est ainsi dévoilé dans la base de données Vinland (
viral infection landscape
,
https://vinland.network
).
Ainsi, l’identification systématique de couples peptide viral-protéine humaine dans Vinland permet d’établir une banque de peptides viraux susceptibles de moduler une grande variété de fonctions cellulaires. Cette banque peut être criblée sur la base de n’importe quel modèle de processus biologique, ou de maladie, infectieuse ou non (
Figure 2
). Nous avons réalisé plusieurs criblages et prouvé que des peptides viraux de toutes origines sont capables, en ciblant des fonctions cellulaires, d’empêcher ou au contraire d’activer la réplication de différents virus ainsi que d’une bactérie substitut d’un agent bioterroriste [
3
].
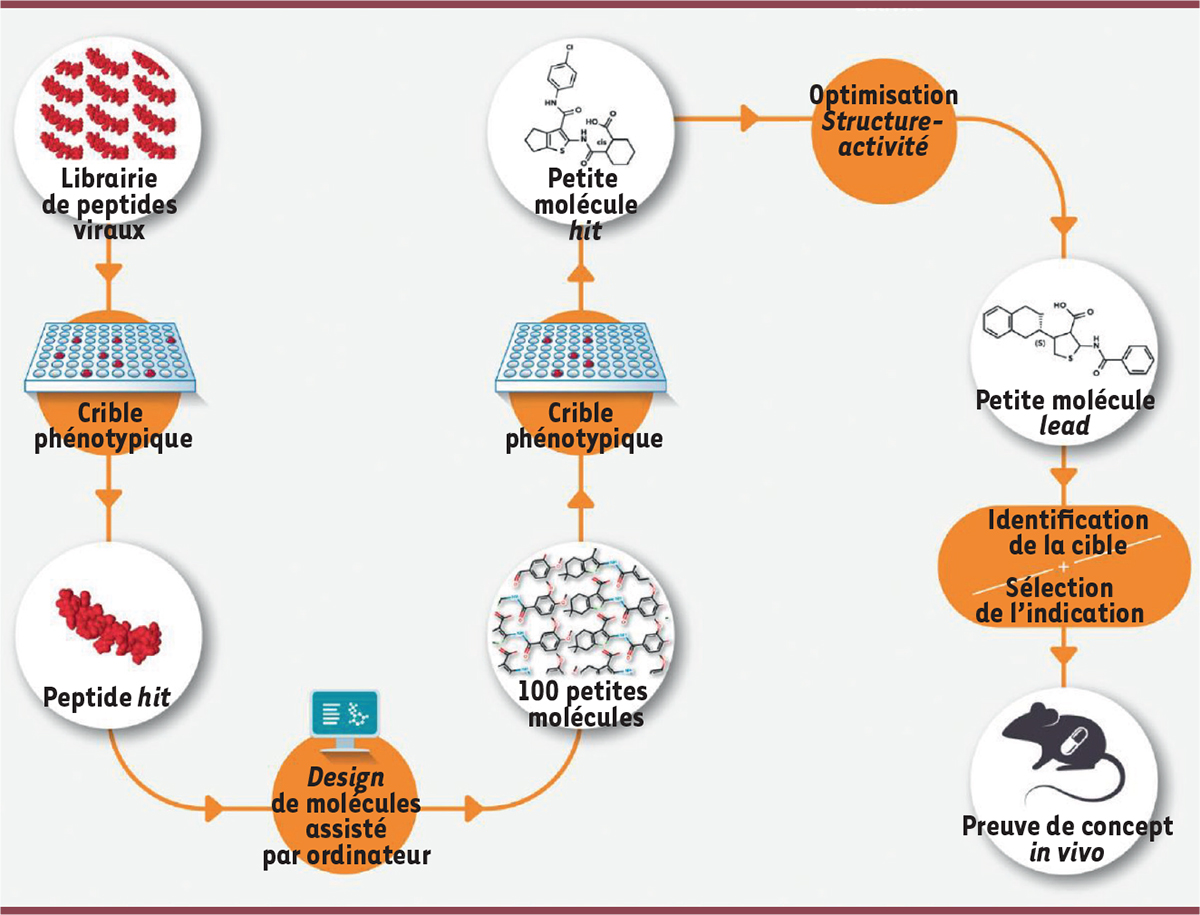
 | Figure 2.
Étapes séquentielles du processus de découverte de médicaments biomimétiques.
La banque de peptides viraux est criblée dans un test cellulaire. Les pharmacophores (molécule ou région d’une molécule dont l’activité biologique possède un effet thérapeutique) d’un peptide sont traduits par un chimioinformaticien en une banque de petites molécules. À la suite du crible, les molécules actives servent de matrice structurale pour une optimisation selon une approche fondée sur la relation structure-activité. L’identification de la cible de la molécule la plus active est alors un préalable au choix de l’indication thérapeutique.
|
Nous avons poursuivi la démarche de preuve de concept en nous focalisant sur un peptide issu d’une protéine d’un herpèsvirus [
4
]. Ce peptide est très actif pour empêcher la réplication du virus de la grippe, et n’est pas toxique pour la cellule. Nous avons montré qu’il interagit avec une famille de trois protéines humaines appelées protéines NEET
1,
[
3
]. Si les peptides sont de très bons outils pour explorer des fonctions cellulaires dans les laboratoires de recherche, ils présentent de nombreux obstacles à un développement préclinique et clinique. Un moyen de contourner ces obstacles est de mimer les déterminants structuraux du peptide qui interagissent avec la protéine humaine par une petite molécule chimique dite peptidomimétique (
Figure 2
). Les premières molécules peptidomimétiques ainsi identifiées présentaient le même niveau d’activité que le peptide contre la réplication du virus de la grippe [
3
]. La molécule chimique a ensuite été optimisée (
Figure 2
) de telle sorte que la dernière molécule présentée (dEF3122) est aussi active dans la cellule infectée que la molécule de référence utilisée pour le traitement de la grippe (oseltamivir) [
3
]. Tout au long de ce processus d’optimisation, l’interaction des différentes molécules synthétiques avec les protéines NEET était conservée [
4
].
Les protéines NEET étant impliquées dans diverses maladies, dont la stéatose hépatique non alcoolique [
5
], la molécule dEF3122 a été utilisée pour traiter des souris présentant cette maladie (
Figure 2
). Après huit semaines de traitement, nous avons constaté une amélioration de paramètres hépatiques et rénaux en rapport avec la maladie [
3
].
Cette démarche innovante profite d’abord à la recherche académique, qui bénéficie désormais de la base de données d’interaction virus-hôte la plus complète à ce jour, et aussi d’outils originaux pour explorer un large éventail de fonctions cellulaires dans tous les domaines de la biologie. Elle profite également à l’industrie pharmaceutique, car elle fournit de nombreux points de départ pour une recherche de nouvelles molécules thérapeutiques, y compris contre des protéines humaines considérées jusqu’alors impossibles à cibler.