| |
| Med Sci (Paris). 40(2): 209–211. doi: 10.1051/medsci/2023213.Décryptage de l’histoire naturelle des néoplasmes myéloprolifératifs grâce à une approche d’arbres phylogénétiques Jackson Josil,1a Eva Thuillier,1b Laetitia Chambrun,1c and Isabelle Plo2,3,4d 1M1 Biologie-Santé Université Paris-Saclay
,
Orsay
,
France 2Inserm UMR 1287, Gustave Roussy
,
Villejuif
,
France 3Université Paris-Saclay, Inserm UMR 1287, Gustave Roussy
,
Villejuif
,
France 4Gustave Roussy, Inserm UMR 1287
,
Villejuif
,
France MeSH keywords: Humains, Phylogenèse, Tumeurs, Syndromes myéloprolifératifs, génétique |
Déterminer les étapes initiales d’un cancer et l’histoire naturelle de son développement est un enjeu important pour la prédiction des patients à risque, la prévention de ce cancer et son traitement précoce. Les cancers sont des maladies multi-étapes qui résultent généralement de l’accumulation de différentes mutations dans une même cellule et qui aboutissent à une prolifération cellulaire excessive accompagnée ou pas d’une différenciation anormale. Williams
et al.
ont publié récemment dans la revue
Nature
une étude importante sur les néoplasmes myéloprolifératifs classiques (NMP) ayant pour objectif de dater l’apparition des mutations motrices et leur dynamique d’expansion grâce à une étude phylogénétique [
1
].
|
Les NMP classiques sont des hémopathies malignes chroniques dont la prévalence est d’environ 1/1 000 et dont l’incidence augmente avec l’âge. Les NMP regroupent 3 maladies : la thrombocytémie essentielle, la polyglobulie de Vaquez et la myélofibrose primaire, qui sont toutes caractérisées par une production excessive de cellules myéloïdes (plaquettes, granulocytes, globules rouges). Leurs principales complications sont les hémorragies, les thromboses et une transformation vers des leucémies de très mauvais pronostics. Ces maladies sont d’excellents modèles de leucémogenèse car elles évoluent de façon lente et permettent de suivre les différentes étapes de la transformation [
2
]. Les NMP sont dus au départ à l’acquisition d’une mutation dite « motrice » dans une cellule souche hématopoïétique (CSH). Ces mutations, dont la plus récurrente est
JAK2
V617F
1
, sont « gain-de-fonction » et affectent des gènes impliqués dans les voies de signalisation récepteur de cytokine/JAK2/STAT [
3
]. Les CSH mutées sont aussi susceptibles d’acquérir d’autres anomalies génétiques ou des mutations additionnelles de façon linéaire ou branchée (accumulation de mutations successives dans le clone
JAK2
V617F
initial ou dans des sous-clones de façon simultanée). Ces dernières mutations sont moins spécifiques des NMP puisqu’elles sont retrouvées dans d’autres hémopathies malignes mais expliquent en partie leur hétérogénéité. Elles affectent des facteurs impliqués dans l’épigénétique (DNMT3A [
DNA methyl-3-transferase
] ou TET2 [
ten-eleven translocation 2
]), l’épissage, et ou la transcription [
3
]. Dans certains cas, des mutations comme celles affectant
TET2
ou
DNMT3A
peuvent précéder l’acquisition de la mutation
JAK2
V617F
, ce qui peut conduire à l’amplification en parallèle de CSH mutées seulement pour
TET2
ou pour
DNMT3A
, ou co-mutés avec
JAK2
V617F
.
|
Comprendre l’histoire naturellede la maladie : quelle méthodologie ?
Une question majeure pour comprendre le développement de ces maladies est d’identifier la date d’apparition de ces mutations « motrices » au niveau des CSH et leur dynamique d’expansion en fonction des anomalies génétiques associées. Dans cette étude, Williams
et al.
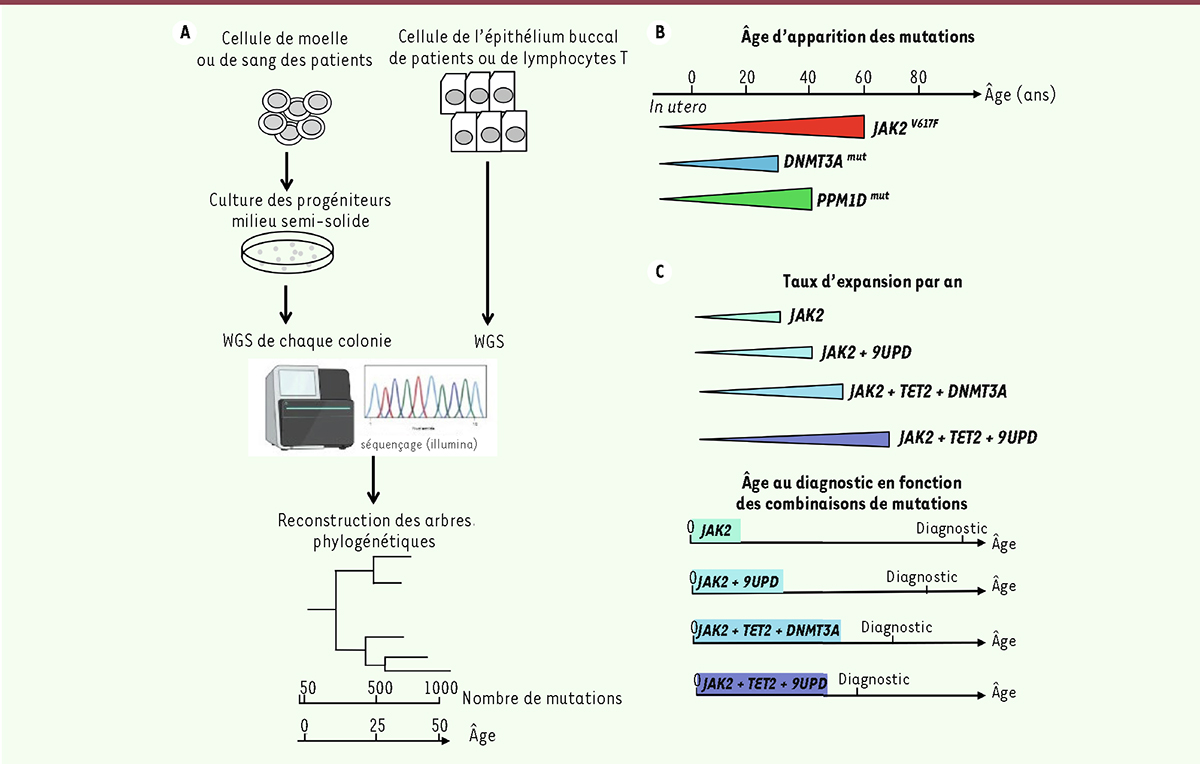
ont reconstruit l’histoire naturelle du développement clonal en étudiant de façon détaillée des patients atteints de NMP. Pour cela, ils ont purifié et mis en culture à l’échelle unicellulaire des cellules souches et progénitrices hématopoïétiques provenant de la moelle osseuse de 12 patients. Chaque progéniteur a donné naissance à une colonie de cellules dont l’ADN a été extrait et soumis à un séquençage entier du génome (
whole genome sequencing
, WGS) en parallèle de l’ADN germinal de cellules de l’épithélium buccal ou de lymphocytes T de chaque patient. Une analyse différentielle des variants du génome a été conduite sur un total de 1 000 colonies et a permis d’identifier toutes les mutations somatiques passagères apparues lors de l’hématopoïèse de chaque patient. En identifiant les mutations somatiques communes ou différentes des colonies et en déterminant les ancêtres communs, les auteurs ont construit des arbres généalogiques des mutations, appelés arbres phylogénétiques. Ceux-ci ont été couplés à une échelle de temps puisque les auteurs ont découvert que les mutations passagères sont acquises de façon linéaire au cours de la vie avec un taux d’environ 18 mutations/cellule/an. Ainsi, le nombre de mutations dans les progéniteurs à un temps donné peut servir d’horloge moléculaire (
Figure 1A
). Grâce à cette méthode, les auteurs ont pu dater l’origine des mutations « motrices », comme celles de
JAK2
V617F
,
TET2
ou
DNMT3A
, leur ordre d’apparition et leur impact sur la prolifération des CSH.
 | Figure 1.
Méthodologie et conclusions sur l’origine des mutations et le déclenchement de la maladie. A.
Description schématique de la méthodologie employée. Les cellules de la moelle ou du sang des patients atteints de NMP ont été ensemencées en milieu semi-solide. Cette technique permet d’obtenir des colonies de progéniteurs qui ont ensuite été analysées par séquençage entier du génome (WGS). Les mutations acquises durant l’hématopoïèse ont ensuite été identifiées pour chaque patient en comparant le génome de chaque progéniteur hématopoïétique avec le génome des cellules de l’épithélium buccal ou des lymphocytes T. Après cette étape, les auteurs ont reconstruit les arbres phylogénétiques des mutations acquises en les couplant à leur âge d’apparition en fonction du nombre de mutations par colonie.
B, C..
Le déclenchement de la maladie dépend de plusieurs paramètres : 1) de l’âge d’apparition des mutations qui surviennent le plus souvent
in utero
ou dans l’enfance et 2) du nombre de mutations associées qui influence le taux d’expansion/an des cellules souches mutées. Ainsi, la latence de la maladie jusqu’au diagnostic varie selon le type et le nombre de mutations dans un même clone. Par exemple, JAK2
V617F
seul a une expansion très hétérogène avec un maximum de 73 % par an, cette expansion est très augmentée en présence de mutations multiples (
TET2
,
DNMT3A
,
9UPD
) et peut aller jusqu’à 303 % par an.
|
|
Apparition précoce de la mutation
JAK2
V617F
, des décennies avant le diagnostic
Cette étude a permis de caractériser une certaine hétérogénéité et complexité génétique des NMP dans les CSH. D’abord, elle révèle l’apparition fréquente d’anomalies génétiques similaires, principalement sur les gènes
JAK2
,
DNMT3A
,
TET2,
dans différentes CSH d’un même patient à des temps différents, ce qui souligne « une évolution clonale en parallèle ». Elle décrit aussi de multiples acquisitions de perte d’hétérozygotie du chromosome (chr) 9p, des acquisitions d’autres anomalies cytogénétiques indépendantes, ou pas, de l’acquisition de
JAK2
V617F
, des acquisitions de plusieurs autres mutations, comme celles affectant
TET2
,
DNMT3A
et
PPM1D
(
protein phosphatase, Mg
2+
/Mn
2+
dependent 1D
), qui évoluent dans le même clone que
JAK2
V617F
ou en parallèle dans des clones séparés. Ensuite, de façon très nouvelle et inattendue, contrairement au fait que les NMP soient des maladies du sujet âgé, les auteurs ont observé que les mutations
JAK2
V617F
et celles de
DNMT3A
et
PPM1D
surviennent très tôt au cours de la vie des individus, quel que soit l’âge du diagnostic (
Figure 1B
). Alors que chez 10 patients, les mutations apparaissent au cours de la première décennie de l’enfance, pour 2 autres patients, les mutations sont acquises
in utero
(à 33 semaines post-conception pour
JAK2
V617F
et à 8 semaines post-conception pour
DNMT3A
). De manière intéressante, les auteurs montrent aussi que l’acquisition de mutations additionnelles peut survenir longtemps après celle de
JAK2
V617F
et que des clones génétiquement plus complexes peuvent prendre le pas sur les clones initiaux. Une modélisation mathématique a ensuite été conduite en se fondant sur les arbres phylogénétiques pour déduire le taux d’expansion par an des différents clones mutés (ou
fitness
) ; celle-ci a aussi été corrélée à la latence de la maladie. Ainsi, il est apparu que le taux de croissance du clone
JAK2
V617F
seul est très hétérogène selon les patients avec un maximum de 73 % par an, ce taux semble très augmenté en présence de mutations multiples (
TET2
,
DNMT3A
,
9UPD
) et peut aller jusqu’à 303 % par an, ce qui conduit au diagnostic plus précoce de la maladie par rapport à un patient qui a un clone muté uniquement
JAK2
V617F
. Ainsi, deux patients peuvent être diagnostiqués au même âge et avoir une dynamique d’expansion très différente à cause du nombre/type de mutations. (
Figure 1C
) L’expansion peut néanmoins être ralentie par les traitements, comme cela a été mis en évidence chez un patient traité par l’IFN (interféron)-a. Enfin de façon assez surprenante, la mutation
DNMT3A,
la plus récurrente dans l’hématopoïèse clonale de signification indéterminée (définie par l’expansion d’un clone chez des individus sans maladie), présente une expansion très lente quand elle est isolée.
|
Vers une stratégie de dépistage et d’intervention précoce
Dans l’ensemble, l’aspect le plus intéressant de ce travail est de montrer que les mutations comme
JAK2
V617F
,
DNMT3A
ou
PPM1D
arrivent tôt au cours de la vie des individus (
in utero
ou dans l’enfance), soit des dizaines d’années avant le diagnostic de la maladie. Il souligne la latence longue entre l’acquisition de la mutation et le déclenchement de la maladie et vient conforter avec la même méthode mais avec plus de patients un travail précédent de l’équipe de van Egeren
et al.
(
Figure 1C
) [
4
]. Cette étude vient aussi étayer le travail de Hirsch
et al.
[
6
], montrant la présence de la mutation
JAK2
V617F
dans le sang de cordon, et celui de Watson
et al.
[
7
] montrant l’expansion lente du clone
JAK2
V617F
dans la population générale. Un autre aspect très intéressant est de souligner que d’autres facteurs modifient cette latence du NMP en particulier les anomalies génétiques associées, comme celles affectant l’épigénétique. En outre, les variabilités de latence observées avec une mutation
JAK2
V617F
seule suggèrent fortement que tous ces clones n’évoluent pas forcément vers un NMP mais augmentent seulement la probabilité de déclencher une maladie. Ainsi, comprendre quels sont les autres facteurs, génétiques (somatiques ou germinaux) ou extrinsèques, qui, une fois combinés, entraînent la maladie est une perspective intéressante de cet article. Les données demandent cependant à être confirmées avec d’autres approches et avec de plus larges cohortes de patients plus hétérogènes en âges. À la suite de ce travail, l’équipe de Hermange
et al.
a validé les mêmes conclusions en utilisant des données d’architecture clonale de progéniteurs de patients et un modèle mathématique, ce qui pourrait permettre la mise en place de méthodes de dépistage précoce [
5
].
Cette étude étend largement nos connaissances sur la progression des NMP et pourrait être pertinente pour d’autres tumeurs malignes myéloïdes et pour les tumeurs solides. Elle ouvre aussi des perspectives médicales intéressantes notamment sur la détection des patients à risque de développer ces maladies dans la population générale. Identifier ces individus pourrait permettre de prédire une éventuelle apparition de NMP et permettrait de traiter les patients précocement avec des anticoagulants avant qu’ils ne développent des hémorragies/thromboses souvent inaugurales. Alternativement, des thérapies comme l’IFN-a limitant le développement de la maladie ou mettant en rémission moléculaire certains patients pourraient être utilisées pour éviter le développement de la maladie [
8
,
9
]. Une intervention thérapeutique précoce pourrait être une approche prometteuse dans la grande majorité des cancers avant l’acquisition par le clone malin d’une architecture clonale complexe associée à une résistance à toute thérapie.
|
Les auteurs déclarent n’avoir aucun lien d’intérêt concernant les données publiées dans cet article.
|
Footnotes |
1.
Williams
N
,
Lee
J
,
Mitchell
E
,
et al.
Life histories of myeloproliferative neoplasms inferred from phylogenies.
.
Nature
.
2022;
;
602
:
:162.
–
8
2.
Arber
DA
,
Orazi
A
,
Hasserjian
R
,
et al.
The 2016 revision to the World Health Organization classification of myeloid neoplasms and acute leukemia.
.
Blood
.
2016;
;
127
:
(2)
:391.
–
405
.
3.
Vainchenker
W
,
Kralovics
R
.
Genetic basis and molecular pathophysiology of classical myeloproliferative neoplasms.
.
Blood
.
2017;
;
129
:
:667.
–
679
.
4.
Van Egeren
D
,
Escabi
J
,
Nguyen
M
,
et al.
Reconstructing the Lineage Histories and Differentiation Trajectories of Individual Cancer Cells in Myeloproliferative Neoplasms.
.
Cell Stem Cell
.
2021;
;
28
:
:514.
–
523.e9
.
5.
Hermange
G
,
Rakotonirainy
A
,
Bentriou
M
,
et al.
Inferring the initiation and development of myeloproliferative neoplasms.
.
Proc Natl Acad Sci U S A
.
2022;
;
119
:
:e2120374119.
.
6.
Hirsch
P
,
Mamez
AC
,
Belhocine
R
,
et al.
Clonal history of a cord blood donor cell leukemia with prenatal somatic JAK2 V617F mutation.
.
Leukemia
.
2016;
;
30
:
(1)
:756.
–
759
.
7.
Watson
CJ
,
Papula
AL
,
Poon
GYP
,
et al.
The evolutionary dynamics and fitness landscape of clonal hematopoiesis.
.
Science
.
2020;
;
367
:
:1449.
–
54
.
8.
Mosca
M
,
Hermange
G
,
Tisserand
A
,
et al.
Inferring the dynamics of mutated hematopoietic stem and progenitor cells induced by IFNα in myeloproliferative neoplasms.
.
Blood
.
2021;
;
138
:
(2)
:231.
–
43
.
9.
Gisslinger
H
,
Klade
C
,
Georgiev
P
,
et al.
Ropeginterferon alfa-2b versus standard therapy for polycythaemia vera (PROUD-PV and CONTINUATION-PV) : a randomised, non-inferiority, phase 3 trial and its extension study.
.
Lancet Haematol
.
2020;
;
7
:
:e196.
–
208
.
|