Lors du recours aux expérimentations animales, le protocole expérimental est d’une importance cruciale, tant d’un point de vue scientifique pour garantir fiabilité et reproductibilité des analyses, que du point de vue éthique. En effet, améliorer la reproductibilité des expériences animales est une condition fondamentale pour respecter les recommandations relatives au remplacement, à la réduction et au raffinement (règle des 3R), cela afin de réduire le nombre d’animaux utilisés et d’affiner les procédures mises en œuvre dans le but de minimiser la douleur et la détresse des animaux [ 1 ]. En particulier, concernant la réduction du nombre d’animaux, une conception adéquate des groupes expérimentaux est essentielle pour garantir des résultats robustes et reproductibles, tout en maximisant les informations collectées sur chaque animal afin d’éviter l’utilisation d’animaux supplémentaires, et en minimisant les animaux écartés car associés à des valeurs aberrantes [ 1 ].
Nombreuses peuvent être les raisons expliquant la faible reproductibilité des résultats des expérimentations animales, telles qu’un design d’expérience inadapté ou l’utilisation de méthodes statistiques inadéquates. De multiples facteurs génétiques ou environnementaux, dont certains sont incontrôlables, sont susceptibles d’influer sur les observations recueillies lors des expérimentations [ 2 ]. Parmi ces facteurs de confusion, les différences de microbiote intestinal entre les groupes expérimentaux ont longtemps été ignorées, bien qu’elles contribuent à cette faible reproductibilité [ 3 ]. L’influence du microbiote intestinal sur la physiologie, le développement du système immunitaire et des muqueuses de son hôte, humain ou animal, est aujourd’hui admise [ 3 ]. En outre, l’utilisation de modèles animaux, tels que le modèle murin classique Mus musculus , joue un rôle primordial dans le défi consistant à dépasser les études de corrélation pour établir les liens de causalité dans les relations hôte-microbiote [ 4 ].
Compte tenu de ces éléments, il semble rationnel que le microbiote intestinal soit pris en considération lors de la conception et de l’interprétation des expériences utilisant des modèles animaux, que le sujet d’étude porte ou pas sur le microbiote.
Dans les faits, malgré les efforts de standardisation des procédures d’élevage et d’expérimentation, malgré les efforts afin d’offrir un cadre contrôlé pour étudier l’association entre patrimoine génétique de l’hôte, facteurs environnementaux et composition du microbiote, les modèles murins ne sont pas exempts de variabilité du microbiote intestinal. La constitution des groupes d’animaux à comparer lors d’une expérimentation est actuellement une étape retenant peu l’attention de l’expérimentateur, qui s’en remet généralement au hasard. Il s’agit cependant d’une source de biais, parfois majeurs, établis avant même que l’expérimentation ne débute réellement, et pouvant conduire à écarter par la suite des valeurs jugées aberrantes. La probabilité d’être confronté à des groupes dissimilaires en termes de microbiote intestinal lors d’une répartition aléatoire des animaux est assez forte (plus de 10 % des designs expérimentaux, selon notre expérience, sont soumis à des biais initiaux) et les solutions ne visant qu’à atténuer ce risque peuvent être insuffisamment efficaces. Aussi proposons nous une nouvelle stratégie qui prend en considération la réalité des différences d’empreintes de microbiote entre les animaux, à partir des données issues d’analyses métagénomiques 16S, et qui utilise ces différences pour guider la répartition de ces animaux dans les différents groupes d’une étude expérimentale. Cette stratégie permet de constituer des groupes similaires quant à leur profil de microbiote fécal avant le début de l’expérience [ 5 ].
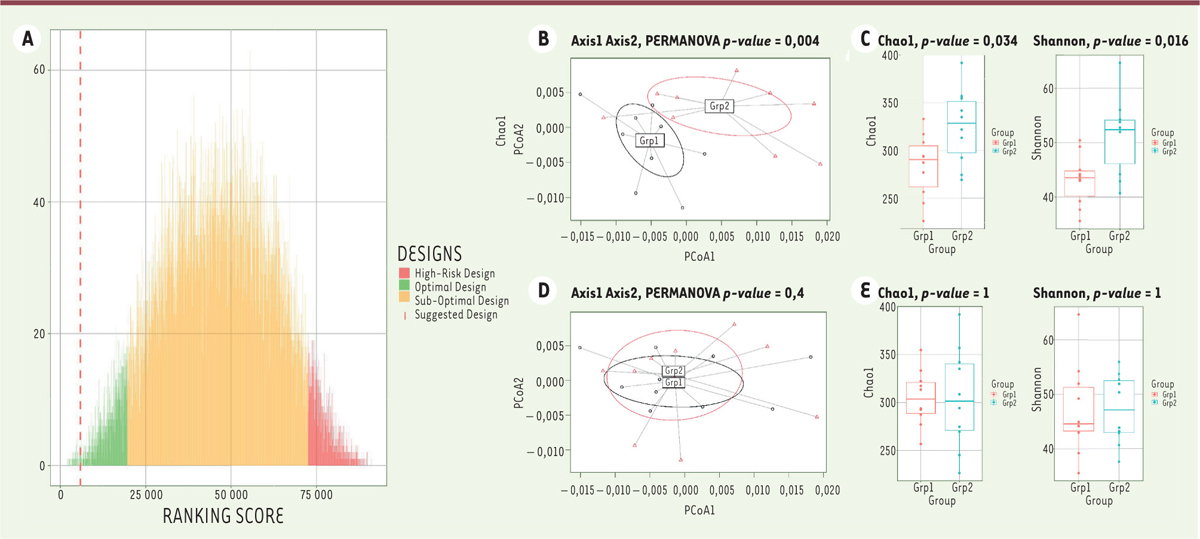
Pour cela, nous avons élaboré un nouvel algorithme, que nous avons nommé « Bact-to-Batch » [ 5 ]. L’algorithme se fonde sur les mesures d’indices de diversité alpha et bêta des microbiotes intestinaux de tous les animaux destinés à l’étude expérimentale, pour proposer un ensemble de designs d’assignation des animaux considérés comme optimaux, grâce à une méthode permettant de maximiser les distances intra-groupe tout en minimisant les distances inter-groupes. Cette méthode a été mise au point sur un total de 20 souris, que nous souhaitions répartir en deux lots similaires en termes de microbiote. Après analyse métagénomique classique du microbiote bactérien de ces souris et obtention des indices de diversité alpha et bêta pour chaque animal, nous avons utilisé l’algorithme Bact-to-Batch pour obtenir un design optimal d’assignation des animaux en deux groupes. Afin de situer la qualité de ce design par rapport à l’exhaustivité des designs possibles, nous avons évalué les scores de similarité sur les plus de 90 000 possibilités de combinaisons de répartition des 20 souris en deux lots de 10 souris ( Figure 1 ). Les cinq premiers centiles des scores de classement étaient considérés comme des « modèles optimaux », et les scores de classement supérieurs au 95 e centile comme « modèles à haut risque ». Les modèles se situant entre le 5 e et le 95 e centile étaient considérés comme des « modèles sous-optimaux ». L’algorithme Bact-to-Batch a systématiquement proposé des designs dans les cinq premiers centiles ( Figure 1 ).
| Figure 1.
Scores de similarité, en termes de microbiote intestinal, de toutes les combinaisons de répartition possibles de 20 souris en deux lots de 10 animaux, et différences de diversité du microbiote intestinal entre les deux groupes du modèle de répartition le plus optimal et de celui le moins favorable, grâce à l’algorithme Bact-to-Batch. (A)
Diagramme des scores de similarité des microbiotes intestinaux des deux groupes d’animaux pour les 92 378 combinaisons de répartition possibles. Les scores de couleur verte, orange et rouge désignent respectivement les modèles optimaux, sous-optimaux et à risque de répartition des animaux. La ligne en trait pointillé correspond au résultat optimal fourni par l’algorithme. Pour le modèle le moins favorable :
(B)
analyse en coordonnées principales mettant en évidence deux groupes distincts en termes de composition du microbiote intestinal ;
(C)
« boîtes à moustaches » mettant en évidence les différences de deux indices d’alpha-diversité du microbiote entre les deux groupes d’animaux. Pour le modèle le plus favorable :
(D)
analyse en coordonnées principales montrant deux groupes similaires en termes de composition du microbiote intestinal ;
(E)
« boîtes à moustaches » montrant des indices d’alpha-diversité semblables (figure d’après [
5
]).
|
Dans cette étude, le temps de calcul de l’exhaustivité des combinaisons était de 55 minutes, alors que l’algorithme Bact-to-Batch proposait une solution optimale en 13 secondes. Si le calcul exhaustif et la sélection de la solution « exacte » étaient possibles dans ce cas, le nombre de modèles possibles dans d’autres études expérimentales (x animaux répartis en y groupes) et le temps nécessaire pour calculer les scores de similarité et leur classement peuvent rapidement devenir un verrou. Par exemple, si l’on souhaite évaluer exhaustivement la répartition de 20 animaux en quatre groupes, il faudrait plus de 30 ans avec un ordinateur de configuration classique. L’approche heuristique proposée avec l’algorithme Bact-to-Batch permet au contraire d’obtenir une solution optimale (toujours proche de la solution exacte) en quelques secondes, et ce, quel que soit le nombre de modèles possibles.
L’algorithme Bact-to-Batch a également été appliqué à des résultats publiés précédemment avec un modèle murin de lupus [ 6 ], en utilisant les données métagénomiques disponibles pour les 28 souris au début de cette étude. Le classement de l’allocation des souris en trois groupes homogènes proposé par notre algorithme était le 6 e /90 000 combinaisons 1 ( top 0,007 %). Selon Bact-to-Batch, la répartition des souris en trois lots qui a été sélectionnée par les auteurs, vraisemblablement fondée sur la randomisation, a obtenu un score de similarité au-dessus du 95 e centile, considéré comme la limite de la catégorie de conception à haut risque de biais. Ainsi, sans remettre en cause les résultats de cette étude, nous pouvons souligner le fait que, selon notre algorithme, la répartition des souris en trois groupes expérimentaux n’était pas optimale pour ce qui concerne la similarité des microbiotes intestinaux.
En conclusion, le microbiote intestinal pouvant avoir une influence globale sur les résultats de certaines expérimentations animales (qu’elles soient fondées ou non sur une étude métagénomique), nous proposons d’utiliser ce nouvel algorithme « Bact-to-Batch » pour constituer les groupes d’animaux à comparer. Cette stratégie s’appuie sur une image métagénomique du microbiote fécal, qui peut être obtenue selon diverses modalités : en utilisant un séquenceur d’ADN haut-débit de deuxième génération (comme dans cette étude pilote) ou en utilisant un séquenceur de haut-débit portatif de troisième génération, ce qui facilite la mise en œuvre de cette stratégie tout en minimisant les délais de restitution des analyses.
La sécurisation de l’étape d’assignation des animaux aux différents groupes expérimentaux, qui permet de disposer de groupes similaires en termes de microbiote intestinal, devrait permettre, en limitant les biais expérimentaux, de favoriser la reproductibilité expérimentale, tout en minimisant le nombre de valeurs aberrantes, donc en réduisant le nombre d’animaux non contributifs à la démarche expérimentale.
Dans un contexte où l’emploi d’animaux dans les expérimentations doit être mûrement réfléchi, il convient de mettre en œuvre des procédures garantissant l’utilité de tous les animaux contributeurs. Les quelques contraintes de cette nouvelle stratégie de répartition des animaux dans les groupes expérimentaux (expertise nécessaire, coût financier, etc. ) s’effacent derrière les gains de robustesse et de reproductibilité des résultats expérimentaux qu’elle permet. Cela ne fait aucun doute dès lors que le design expérimental inclut des analyses du microbiote dans les différents groupes constitués, mais cela semble valoir aussi pour les autres cas, tant le microbiote intestinal influe sur l’immunité et bien d’autres fonctions physiologiques de l’animal.