| |
| Med Sci (Paris). 39(10): 732–737. doi: 10.1051/medsci/2023122.Épigénétique et vieillissement Comment l’épigénétique est associée au vieillissement Jean-Pierre Henry1* 1Ancien directeur de l’institut de biologie physico-chimique, Fondation Edmond de Rothschild
,
13 rue Pierre et Marie Curie
,
75005Paris
,
France |
Vignette (© Jean-Pierre Henry).
Comprendre comment les cellules eucaryotes modulent l’expression de leur patrimoine génétique est l’objectif de l’épigénétique. Toutes les cellules possèdent le même ADN codant : chez
Homo sapiens
, une vingtaine de milliers de gènes. Mais chacune n’utilise qu’une petite fraction de ces gènes : ceux adaptés à sa structure et importants à ses fonctions. Au cours de la différenciation, l’épigénétique va assurer la sélection de ces gènes en favorisant ou en inhibant leur expression, aboutissant à environ 200 types cellulaires différents. Chez l’organisme adulte, l’épigénétique permettra à la cellule de s’adapter aux changements de l’environnement, en modifiant l’expression de certains gènes, les réprimant ou les favorisant. Le vieillissement est reconnu comme un processus cellulaire aboutissant à la diminution des performances physiques et psychologiques, et ouvrant la voie à de nombreuses maladies, cardiaques, articulaires, respiratoires, des cancers et des maladies neurodégénératives. Mais dans quelle mesure l’épigénétique est-elle associée au vieillissement ? Les dysfonctionnements cellulaires liés au vieillissement trouvent leur origine en particulier dans l’accumulation de mutations somatiques, mais ils pourraient aussi provenir de l’épigénome, aboutissant à la modification des gènes qui sont exprimés ou non.
L’ADN cellulaire humain comporte plus de trois milliards de paires de bases nucléotidiques. Sa longueur totale est estimée à 1,80 m. Il est enfermé dans le noyau de la cellule, une sphère d’une dizaine de microns de rayon. Cette organisation requiert une structuration complexe de l’ADN, en sous-ensembles, les chromosomes, eux-mêmes organisés en chromatine, avec l’aide de protéines, en particulier des histones. Cette structuration doit laisser possible la lecture des gènes par les complexes de transcription. L’épigénétique impose à l’ADN une structure dynamique dans laquelle, selon les besoins de la cellule, ses parties lisibles (formant l’euchromatine) apparaissent séparées de ses structures de stockage qui restent illisibles (l’hétérochromatine). Cette organisation de la chromatine fait appel à des protéines, dont les histones, mais elle repose également sur des modifications biochimiques de l’ADN. En particulier, dans l’hétérochromatine, on observe, au niveau des paires CpG
1,
, une méthylation
2
des cytosines (C). L’équilibre entre méthylation et déméthylation de ces cytosines apparaît ainsi comme un mécanisme permettant de réguler la lisibilité des gènes. Les progrès du séquençage de l’ADN autorisent désormais un suivi de cet équilibre sur l’ensemble de l’ADN, ce que l’on nomme méthylome.
|
Vieillissement et méthylation de l’ADN : l’horloge épigénétique
Si le vieillissement est d’origine épigénétique, l’analyse de la méthylation de l’ADN doit pouvoir fournir des indications sur son implication. Une première indication de l’implication de l’épigénome [
1
] a été donnée par l’analyse de l’ADN salivaire de jumeaux homozygotes (donc possédant exactement le même ADN) âgés de 21 à 55 ans. En effet, la méthylation des génomes des jumeaux s’est révélée être différente entre jumeaux et les différences observées étaient accentuées avec l’âge des individus. La méthylation de 88 sites CpG varie ainsi avec l’âge, augmentant pour certains et diminuant pour d’autres. Steve Horvath, l’un des auteurs de cette observation, a approfondi l’analyse. Dans une publication parue en 2013 [
2
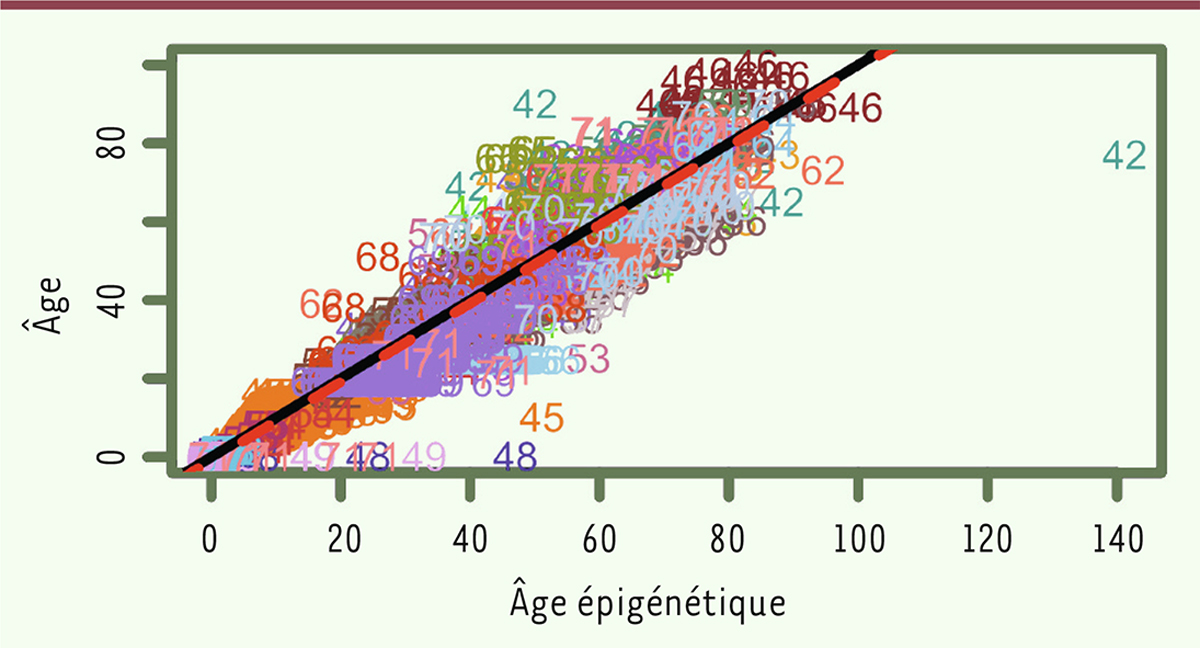
], dont il est le seul auteur, il analyse 8 000 échantillons humains, provenant de 51 tissus et types cellulaires. Fondé sur l’état de 21 369 paires CpG, il développe alors une approche statistique qui sélectionne 353 de ces paires de bases dont la méthylation varie avec l’âge. Un algorithme, s’appuyant sur les coefficients de corrélation entre l’âge chronologique et la méthylation de chaque site, définit un âge épigénétique (en abrégé, ADNm). La correspondance entre âge épigénétique et âge chronologique s’est révélée être bonne sur l’ensemble des données, avec un taux d’erreur entre les deux âges de 3,6 ans (ce qui signifie que pour 50 % des sujets, la différence entre les deux âges est inférieure à 3,6 ans) (
Figure 1
). Étonnamment, cette corrélation reste bonne pour des tissus à divisions rapides, comme le foie, mais aussi pour des tissus à temps de division longs, comme le cortex occipital. Les cellules sanguines, non fractionnées, tout comme les monocytes CD14, les lymphocytes T CD4 ou des cellules mononucléées périphériques isolées, donnent des âges épigénétiques pertinents. Pour un même individu, les valeurs enregistrées sur différents organes sont voisines, et l’analyse de cellules souches pluripotentes ou embryonnaires indique des âges voisins de zéro.
 | Figure 1.
Corrélation existant entre l’âge chronologique (en ordonnée) et l’âge épigénétique ADNm (en abscisse), déterminée d’après la méthylation des paires CpG.
Coefficient de régression : 0,96. Les différentes couleurs correspondent à des échantillons de tissus ou de cellules différents (figure adaptée de [
2
]).
|
Ainsi, qu’elle soit effectuée sur des tissus ou des cellules, l’analyse de la méthylation des paires CpG révèle non pas l’âge des cellules, qui peut être très variable, mais l’âge du propriétaire de ces cellules !
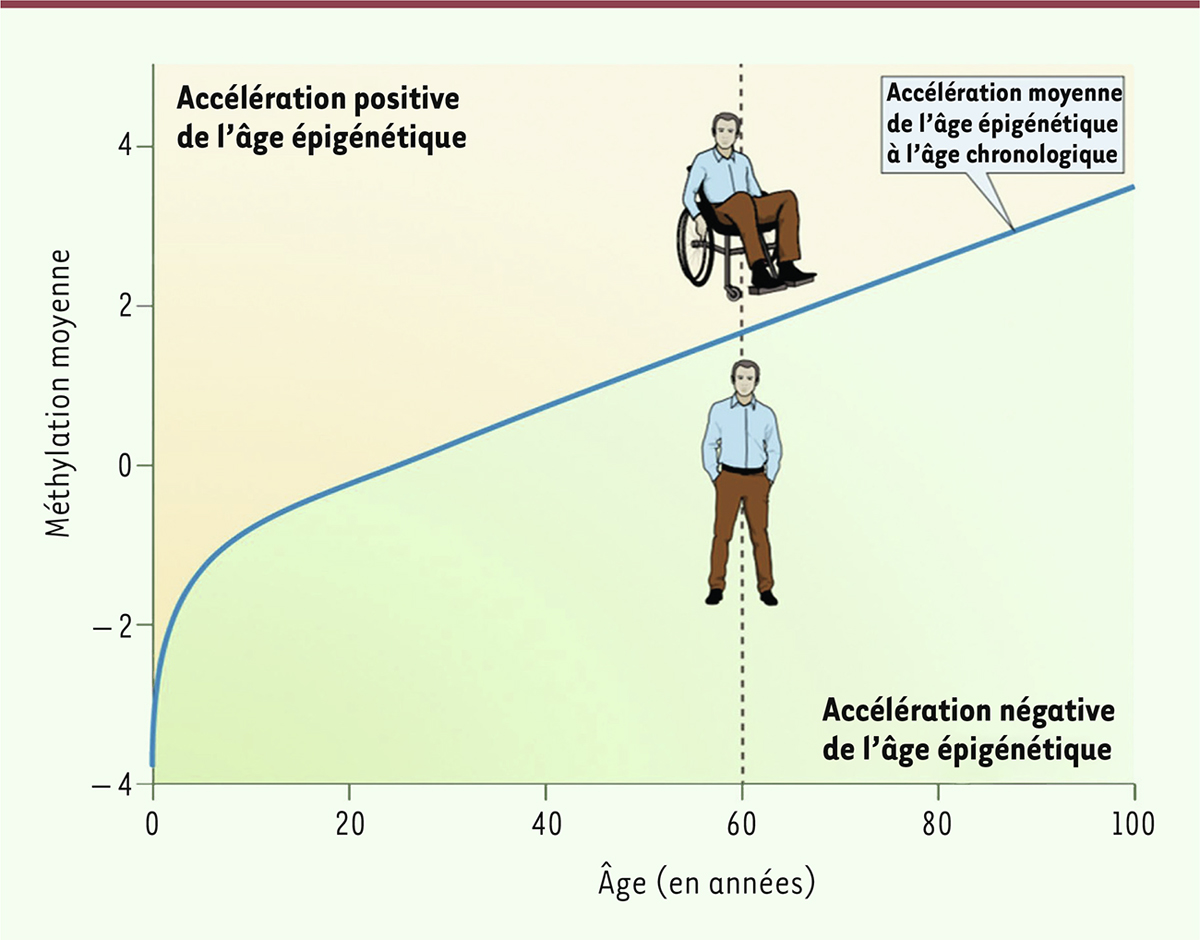
Lorsque la méthylation moyenne des 353 paires CpG est représentée en fonction de l’âge, on observe une courbe nettement biphasique, avec une augmentation de la méthylation rapide jusqu’à l’âge de 20 ans, suivie d’une croissance lente, linéaire (
Figure 2
). Pour Steve Horvath, cette courbe est une image du fonctionnement d’une « horloge épigénétique ADNm » [
3
]. Elle partage le plan en deux parties, la partie supérieure dans laquelle l’âge ADNm est supérieur à l’âge chronologique, correspondant à un vieillissement prématuré, et la partie inférieure indiquant un vieillissement ralenti.
 | Figure 2.
Méthylation moyenne des paires CpG avec l’âge.
La courbe montre un phénomène continu, rapide au cours des 20 premières années, suivi d’une augmentation linéaire. Cette courbe est obtenue à partir de l’étude de la méthylation des 353 paires CpG retenues pour leur variation avec l’âge. Elle représente l’âge épigénétique moyen : des individus avec un âge épigénétique supérieur subissent un vieillissement accéléré et sont susceptibles aux maladies associées (figure adaptée de [
3
]).
|
Cette courbe est importante sur le plan médical, car elle permet d’anticiper, chez des sujets jeunes, des problèmes médicaux, en particulier, des maladies associées au vieillissement. Un travail fondé sur l’étude des constantes biologiques de cohortes de sujets, suivis sur un long terme, avait déjà montré la possibilité de suivre le vieillissement chez des sujets jeunes [
4
].
Le laboratoire d’Horvath s’est alors efforcé de montrer la validité de son approche dans différentes situations. En utilisant la méthylation de l’ADN de cellules mononucléées de sang périphérique pour déterminer l’âge épigénétique, il a montré que les individus issus d’une cohorte de centenaires italiens avaient un âge épigénétique inférieur de 8,6 ans à leur âge chronologique, et que, chez leurs descendants, âgés en moyenne de 71,8 ans, l’âge épigénétiques était également inférieur de cinq ans à l’âge chronologique [
5
]. Une autre étude [
6
] utilisant une variante de l’âge épigénétique appliquée à la cohorte de Framingham, une localité de la Nouvelle-Angleterre où tous les échantillons et dossiers médicaux sont conservés depuis 1948, indique que l’ADNm prédit la mortalité, toutes causes confondues : une année de différence entre âge chronologique et âge épigénétique indique un risque de mortalité augmenté de 4,5 %.
Dans les tissus cancéreux, Steve Horvath a noté une accélération marquée de l’âge épigénétique, une accélération qui s’est révélée pronostique dans le cas de cancers du poumon chez la femme. La morbidité de cette maladie est très variable dans la population et l’accélération de l’âge épigénétique est une indication de la sensibilité des individus à ce cancer. L’étude d’une cohorte de 2 000 femmes sur une période de 20 ans a, en effet, montré que l’accélération de l’âge épigénétique était significativement associée à l’incidence de cancer du poumon [
7
].
|
Conditions du vieillissement et épigénétique
L’étape suivante était de savoir si une modification des conditions du vieillissement était perçue au niveau épigénétique. Chez l’homme, le vieillissement est marqué par la diminution des défenses immunitaires (ce qui est appelé sénescence immunitaire). Celle-ci est attribuée à la disparition, aux environs de 63 ans, du thymus, organe lymphoïde primaire participant à la maturation des lymphocytes T. Cette disparition peut être contrôlée par un traitement par l’hormone de croissance. Après 12 mois de ce traitement, on observe en effet une diminution de l’âge épigénétique des sujets traités d’environ 2,5 ans [
8
]. Mais l’éthique restreint ce type d’expériences, et elles n’ont été développées que dans des modèles animaux. La durée de vie de souris peut également être prolongée par différents traitements, comme un traitement par la rapamycine
3
ou par restriction calorique (une diminution de l’apport alimentaire) ; elle est plus longue chez les souris mutantes naines Dwarf. Chez toutes ces souris, en copiant l’approche définie chez l’homme, un âge épigénétique a été défini à partir de la méthylation de l’ADN des cellules de leur foie. Remarquablement, que ce soit le traitement par l’hormone de croissance, par la rapamycine ou par restriction calorique, tous induisent une diminution de l’âge épigénétique par rapport à l’âge chronologique [
9
].
Actuellement, plus de dix variantes de l’approche de l’âge épigénétique ont été publiées [
10
], en fonction de leur utilisation pour répondre à différentes questions et de la façon dont elles sont développées de manières différentes. Des sites commerciaux proposent désormais des solutions pour prédire son âge épigénétique, même si la valeur médicale de ces solutions n’est pas établie
4
. Au-delà de ces succès qui ouvrent de nouvelles voies de recherche, de nombreux problèmes théoriques sur son mécanisme biologique restent posés.
|
Âge épigénétique et mécanisme du vieillissement Le mécanisme du vieillissement peut être expliqué selon deux hypothèses, pas nécessairement exclusives. Selon la première, l’accumulation de défauts à différents niveaux, par exemple des mutations somatiques, le raccourcissement des télomères, la sénescence, explique les phénomènes cellulaires caractérisant le vieillissement. La seconde hypothèse propose un phénomène progressif associé au développement comme nous le détaillons ci-dessous.
L’âge épigénétique fourni par l’algorithme élaboré par Horvath est déterminé à partir des méthylations des paires CpG de l’ADN retenues pour leur variation avec l’âge. L’ADN initial provient généralement de mélanges de cellules et, pour chaque position analysée, la méthylation est donc une moyenne [
11
]. Avec l’âge, cette moyenne peut augmenter ou diminuer, et la dispersion des points (la variance) peut changer. L’augmentation de la variance est considérée comme une perte d’information. Elle serait due à des variations intrinsèques, mais aussi à l’environnement. Quant à la variation de l’intensité de la méthylation, elle produirait des changements dans l’expression des gènes. Cette variation est faible : la différence entre des individus de moins de 35 ans et de plus de 55 ans est de l’ordre de 3,2 %, et l’effet ne sera détectable que dans des cellules soumises à une agression, comme une infection, et qui voient leur environnement modifié. L’augmentation de la méthylation produit une diminution de l’accessibilité de la chromatine aux régulateurs géniques. Elle est observée à proximité de gènes codant des facteurs de transcription qui se lient aux domaines
Homeobox
, des séquences d’ADN que l’on retrouve dans certains gènes essentiels au développement embryonnaire, à la morphogenèse et la différenciation cellulaire. La diminution de la méthylation de CpG est quant à elle localisée dans des tissus particuliers et a des rôles plus spécifiques.
La confirmation du rôle général (ou global) de ces méthylations est donnée par l’existence de réseaux, montrant une méthylation concomitante de différentes régions de l’ADN (co-méthylation). Les paires connectées (de plusieurs centaines à plusieurs milliers) forment des modules qui sont impliqués dans des fonctions différentes. On distingue en effet des modules participant au fonctionnement mitochondrial, à la modification de la chromatine, à des processus immunologiques ou à l’autophagie.
La notion d’âge épigénétique suggère donc que le vieillissement pourrait être un processus physiologique continu et non une suite d’accidents. Un article récent [
12
] confirme indirectement cette hypothèse. S’appuyant sur les précédents travaux, les auteurs, dont Steve Horvath, développent l’idée d’une horloge épigénétique qui serait valable pour tout tissu et applicable à tous les mammifères. Ils ont utilisé 11 754 échantillons provenant de 59 tissus différents, obtenus à partir de 185 espèces de mammifères appartenant à 19 ordres taxonomiques, allant de l’homme à la baleine, en passant par les marsupiaux et l’ornithorynque.
L’approche est celle suivie précédemment mais, pour effectuer des comparaisons entre espèces ayant des durées de vie très différentes, une autre horloge est proposée. Dans cette horloge, la durée de vie n’est pas mesurée en années, mais est donnée par une valeur comprise entre zéro et un, le « un » étant l’âge maximal observé chez l’espèce. Une valeur est également prise en considération : l’âge de la maturité sexuelle. Ces horloges ont été évaluées dans de multiples tissus et elles donnent une bonne estimation de l’âge chronologique, avec une erreur médiane inférieure à un an, et une erreur relative inférieure à 3,3 %, pour des tissus aussi différents que le cerveau, la rate, le foie, le rein, la peau et les cellules sanguines. La recherche des gènes où se trouvent des paires CpG variant avec l’âge dans différentes espèces identifie les mêmes gènes. Ainsi, dans 28 espèces, les paires CpG 12841266 et 11084334 sont associées avec l’âge (coefficient de corrélation supérieur ou égal à 0,8). Les gènes correspondants sont donc probablement impliqués dans le vieillissement chez ces espèces. Parmi les paires CpG modulées positivement ou négativement, 20 sont proches de gènes participant à des processus de développement, incluant le développement embryonnaire. Les sites CpG, dont la méthylation augmente avec l’âge, sont situés sur des régions de l’ADN impliquées dans la régulation de l’expression de gènes du développement, par des mécanismes très conservés parmi les mammifères. Quant aux sites dont la méthylation diminue avec l’âge, ils paraissent associés à des mécanismes différents : la régulation des rythmes circadiens, le fonctionnement mitochondrial ou le fonctionnement de mTOR (complexe contrôlant la croissance cellulaire). Ces mécanismes participent au vieillissement et l’ensemble des changements de méthylation apparaissent comme liés aux différentes facettes du vieillissement, favorisant l’hypothèse d’un vieillissement progressif lié au développement. |
Vieillissement et développement
Si l’âge épigénétique est associé au développement, il doit être voisin de zéro dans les cellules embryonnaires pluripotentes. Les premiers travaux de Steve Horvath avaient confirmé ce point. Des cellules souches pluripotentes induites par l’expression des gènes de Yamanaka
5
s’avèrent aussi d’un âge épigénétique très faible par rapport à des cellules témoins non induites. Cette notion est conforme à la vision classique d’August Weismann (1834-1914) opposant les cellules reproductrices, « le germen », qui ne vieillissent pas, au « soma », soumis au vieillissement. Toutefois, les cellules germinatives restent dans l’organisme pendant des temps très longs, plusieurs dizaines d’années, et elles subissent des accidents qui doivent probablement avoir pour conséquences un certain vieillissement. Des travaux récents [
13
] ont mis en évidence un rajeunissement de l’âge épigénétique d’embryons de souris, entre quatre et six jours de développement. Le développement d’une approche permettant de mesurer l’âge épigénétique de cellules isolées [
14
] a montré que ce rajeunissement, définissant ce que les auteurs ont appelé «
ground zero
», s’est effectué au stade gastrulation du développement, là où les trois feuillets de l’embryon apparaissent. C’est à ce moment que commence le vieillissement, au cœur du développement.
|
Quelques conclusions : fonctionnement de l’horloge épigénétique Si l’ensemble des travaux récents semble révéler un rapport de causalité entre épigénétique et vieillissement, une question reste sans réponse : quel est le mécanisme qui meut l’horloge épigénétique ?
Le vieillissement est, d’abord, un processus cellulaire marqué par un certain nombre d’anomalies, dont la plus apparente est la sénescence. Ce processus est observable
in vitro.
En effet, lorsque des cellules mitotiques sont mises en culture, on observe un nombre limité de divisions (le plafond de Helfrick) au-delà duquel les cellules arrêtent de se diviser, changent de forme, de physiologie et sécrètent des molécules inflammatoires, caractérisant l’état de sénescence. La sénescence participe-t-elle aux processus épigénétiques à l’origine de l’âge épigénétique ? Lorsqu’elles atteignent la sénescence, des cultures de cellules endothéliales montrent une nette augmentation de leurs âges épigénétiques [
15
]. Dans ce cas, la sénescence est induite par le raccourcissement des télomères (c’est la sénescence réplicative). Cet état peut également être induit par des cassures de l’ADN, produites par des rayonnements ionisants. Dans ce cas, l’âge épigénétique n’augmente pas, infirmant donc la causalité entre épigénétique et sénescence.
La seule hypothèse proposant un mécanisme pour l’horloge épigénétique a été avancée par Horvath et Raj [
3
]. Elle suggère que la progression de l’horloge (qu’ils appellent
ticking
, « tic-tac » en français) soit associée à des cellules souches et à leurs divisions asymétriques. De multiples cellules souches assurent la continuité de l’identité des 200 différents types cellulaires qui composent notre corps. Situés dans des niches, elles subissent des divisions mitotiques particulières, asymétriques, une cellule fille gardant les propriétés de la cellule mère, tandis que l’autre cellule devient un progéniteur, capable d’engendrer un ou plusieurs types cellulaires, après migration hors de sa niche. La dérégulation des gènes de développement contrôlés par l’épigénome pourrait engendrer la perte de l’identité cellulaire qui accompagne le vieillissement.
Horvath et Raj reconnaissent toutefois que leur hypothèse est soutenue par peu de preuves expérimentales… Ils citent des travaux effectués sur l’épigénome de cellules uniques [
14
] qui montrent que les cellules de foie de souris, dont les propriétés régénératives sont bien connues, subissent clairement un vieillissement épigénétique, alors que des cellules souches musculaires, peu actives, n’obéissent pas à cette règle.
Les résultats obtenus dans le cadre de l’épigénétique permettent donc de proposer une théorie cohérente du vieillissement : associé au développement, le vieillissement commencerait dès le stade embryonnaire. Les courbes de méthylation moyenne suivent le développement, avec une pente initiale forte, qui devient constante à l’état adulte, ce qui correspond au renouvellement cellulaire. L’existence de ce renouvellement dans tous les organes rendrait compte de cette observation extraordinaire, attribuant à des cellules différentes, à des organes différents, un même âge épigénétique. Le travail effectué chez les mammifères va dans ce sens, montrant qu’une horloge multi-tissu peut s’appliquer à des animaux dont la durée de vie est très différente.
Cette vision du vieillissement a été initialement proposée par Steve Horvath, un bio-mathématicien. Ses résultats sont d’abord apparus incompréhensibles, car sans véritable explication biologique. La cohérence de ces travaux est maintenant patente. Elle ouvre des voies de recherche fascinantes. Mesurer le vieillissement chez des adultes jeunes permettrait en effet de développer des approches préventives. Intervenir sur l’âge épigénétique modifierait le vieillissement, et cela semble possible en ciblant les gènes de Yamanaka [
16
]. Toutefois, il est clair qu’à la lumière de ces travaux, des recherches restent nécessaires pour comprendre les résultats obtenus par ces méthodes statistiques. Par exemple, quels liens existent-ils entre mitose asymétrique et méthylation de l’ADN ?
|
L’auteur déclare n’avoir aucun lien d’intérêt concernant les données publiées dans cet article.
|
Footnotes |
1.
Bocklandt
S
,
Lin
W
,
Sehl
ME
,
et al.
Epigenetic Predictor of Age.
.
PLoS One.
2011;
;
6
:
:e14821.
.
2.
Horvath
S.
DNA methylation age of human tissues and cell types.
.
Genome Biol.
2013;
;
14
:
:R115.
.
3.
Horvath
S
,
Raj
K
.
DNA methylation-based biomarkers and the epigenetic clock theory of ageing.
.
Nat Rev Genet.
2018;
;
19
:
:371.
–
384
.
4.
Belsky
DW
,
Caspi
A
,
Houts
R
,
et al.
Quantification of biological aging in young adults.
.
Proc Natl Acad Sci U S A.
2015;
;
112
:
:E4104.
–
E4E10
.
5.
Horvath
S
,
Pirazzini
C
,
Bacalini
MG
,
et al.
Decreased epigenetic age of PBMCs from Italian semi-supercentenarians and their offspring.
.
Aging.
2015;
;
7
:
:1159.
–
1170
.
6.
Marioni
RE
,
Shah
S
,
McRae
AF
,
et al.
DNA methylation age of blood predicts all-cause mortality in later life.
.
Genome Biol.
2015;
;
16
:
:25.
.
7.
Levine
ME
,
Hosgood
HD
,
Chen
B
,
et al.
DNA methylation age of blood predicts future onset of lung cancer in the women’s health initiative.
.
Aging.
2015;
;
7
:
:690.
–
700
.
8.
Fahy
GM
,
Brooke
RT
,
Watson
JP
,
et al.
Reversal of epigenetic aging and immunosenescent trends in humans.
.
Aging Cell.
2019;
;
18
:
:e13028.
.
9.
Wang
T
,
Tsui
B
,
Kreisberg
JF
,
et al.
Epigenetic aging signatures in mice livers are slowed by dwarfism, calorie restriction and rapamycin treatment.
.
Genome Biol.
2017;
;
18
:
:57.
.
10.
Liu
Z
,
Leung
D
,
Thrush
K
,
et al.
Underlying features of epigenetic aging clocks in vivo and in vitro.
.
Aging Cell.
2020;
;
19
:
:e13229.
.
11.
Seale
K
,
Horvath
S
,
Teschendorff
A
,
et al.
Making sense of the ageing methylome.
.
Nat Rev Genet.
2022;
;
23
:
:585.
–
605
.
12.
Lu
AT
,
Fei
Z
,
Raj
K
,
et al.
Universal DNA methylation age across mammalian tissues.
.
Innov Aging.
2021;
;
5
:
:410.
.
13.
Kerepesi
C
,
Zhang
B
,
Lee
SG
,
et al.
E
pigenetic clocks reveal a rejuvenation event during embryogenesis followed by aging.
.
Sci Adv.
2021
;
:7.
.
14.
Trapp
A
,
Kerepesi
C
,
Gladyshev
VN
.
Profiling epigenetic age in single cells.
.
Nat Aging.
2021;
;
1
:
:1189.
–
201
.
15.
Lowe
D
,
Horvath
S
,
Raj
K
.
Epigenetic clock analyses of cellular senescence and ageing.
.
Oncotarget.
2016;
;
7
:
:8524.
–
8531
.
16.
Takahashi
K
,
Yamanaka
S
.
Induction of Pluripotent Stem Cells from Mouse Embryonic and Adult Fibroblast Cultures by Defined Factors.
.
Cell.
2006;
;
126
:
:663.
–
676
.
|