| |
| Med Sci (Paris). 39(8-9): 665–667. doi: 10.1051/medsci/2023090.Tout savoir sur les mutations germinales chez les vertébrés Chroniques génomiques Bertrand Jordan1* 1Biologiste, généticien et immunologiste, Président d’Aprogène (Association pour la promotion de la Génomique)
,
13007Marseille
,
France MeSH keywords: Humains, Animaux, Enfant, Vertébrés, Mutation, Analyse de séquence d'ADN, Cartographie chromosomique, génétique |
Dans une chronique récente [
1
] (
→
) nous avions analysé les résultats d’études mesurant le taux de mutations somatiques [
2
], ces mutations qui apparaissent dans les cellules de l’organisme et sont impliquées notamment dans le vieillissement cellulaire et la cancérogenèse, mais ne sont pas transmises à la descendance. On constatait notamment que des animaux de longévités très différentes ont, vers la fin de leur vie, accumulé un nombre comparable de mutations somatiques (quelques milliers). La présente chronique concerne, elle, les mutations
germinales
, celles qui apparaissent dans les oocytes et les spermatozoïdes et sont retrouvées dans l’ADN de la descendance. Ces mutations sont très importantes car elles sont responsables de l’évolution des espèces et, parfois, provoquent l’apparition de maladies génétiques. Leur mesure est relativement aisée puisqu’il « suffit » de comparer les séquences intégrales d’ADN de trios (père/mère/enfant) : si ces séquences sont de bonne qualité et que leur analyse informatique est bien conduite, on pourra repérer les mutations apparues
de novo
, absentes chez les parents et présentes dans leur descendance [
3
]. Du point de vue expérimental, cette analyse est nettement moins ardue que la détection de mutations somatiques qui est compliquée par l’hétérogénéité des cellules analysées [
1
,
2
]. Pour fixer les idées, disons que chez l’homme, le nombre de mutations germinales est de quelques dizaines par génération. La plupart de ces mutations n’ont aucun effet, mais certaines peuvent, rarement, inactiver un gène ou changer certaines propriétés de la protéine que celui-là code.
(→) Voir la Chronique génomique de B. Jordan,
m/s
n° 8-9, août-septembre 2022, page 723
|
Une étude de grande ampleur
L’article qui fait l’objet de cette chronique [
4
] émane d’un ensemble de laboratoires européens et chinois, notamment BGI (
Beijing Genomics Institute
) [
5
] qui a réalisé l’ensemble des séquences à l’aide des machines BGI/MGI
1,
[
5
] (
→
).
→ Voir la Chronique génomique de B. Jordan,
m/s
n° 5, mai 2023, page 469
Les auteurs ont choisi 68 espèces de vertébrés (de l’alpaca
Vicugna pacos
à l’orque
Orcinus orca
en passant par l’homme et la souris) pour lesquels une séquence de référence est disponible, ce qui permet d’assembler les courtes séquences produites par les machines
2
. Ils ont identifié 151 trios (souvent deux ou trois trios par espèce) pour lesquels les informations (notamment l’âge à la reproduction) et l’ADN étaient disponibles, ont vérifié les parentés par analyse d’ADN, et procédé à un séquençage intégral et soigné de ces 453 (151 × 3) génomes : la redondance moyenne de lecture était de 70 fois, alors qu’on se contente généralement de 30 fois. Les séquences obtenues pour chacun des trios ont ensuite été comparées selon un schéma très sophistiqué [
6
] pour éliminer autant que possible les artefacts, avec un ensemble de contrôles qui donne confiance dans les résultats.
|
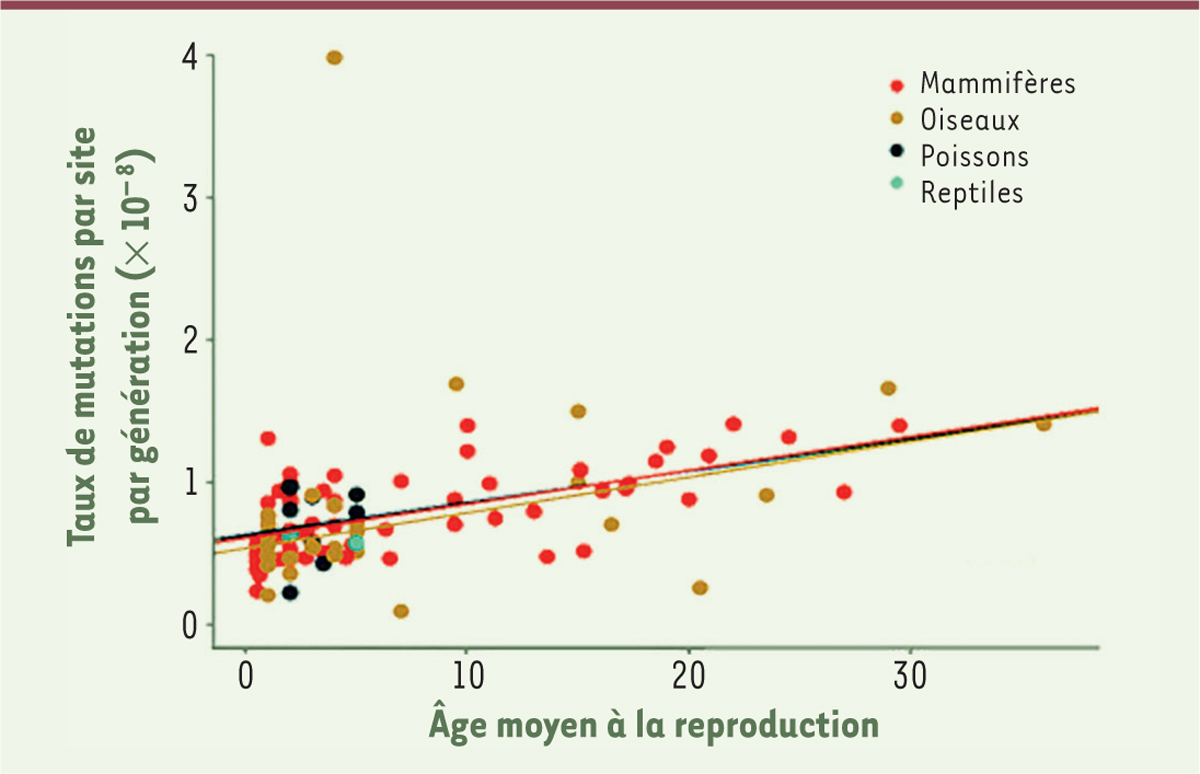
Le résultat le plus frappant de cette comparaison d’un ensemble d’animaux très divers est que le nombre de mutations germinales par génération est du même ordre de grandeur dans tous les cas, alors que les masses des animaux varient d’un facteur de plus d’un million (entre l’orque et le poisson combattant chinois [
Betta splendens
]) et que leur temps de génération va de six mois (pour le chat et le porc) à une trentaine d’années (pour notre espèce et pour certains oiseaux). La
Figure 1
montre l’ensemble des données : le taux de mutation par site et par génération augmente un peu avec l’âge à la reproduction mais reste assez proche de 10
– 8
par site par génération, soit environ 60 mutations germinales pour un génome humain diploïde de 6 000 mégabases (6 × 10
9
bases).
 | Figure 1.
Taux moyen de mutations germinales par génération en fonction de l’âge à la reproduction
(valeur similaire à celle du temps de geìneìration). On voit que ce taux augmente (leìgèrement) avec l’âge à la reproduction, mais qu’il reste du même ordre pour toutes les 68 espèces. Le groupe auquel appartient l’espèce est donneì par la couleur du point ; le point « oiseau » le plus à droite correspond à un couple de pingouins empereur (
Aptenodytes forsteri
) dont les deux partenaires eìtaient âgeìs de 36 ans lors de la reproduction (extrait partiel et modifieì de la
figure 1
de [
4
]).
|
On retrouve aussi, dans cette étude très large, un phénomène déjà connu, la prédominance des mutations germinales provenant des mâles [
7
]. Cela correspond au fait que les spermatozoïdes sont produits tout au long de l’existence, alors que le « stock » d’oocytes est constitué dès la naissance. Les résultats montrent que cet excès est assez variable selon les groupes d’espèces : faible chez les reptiles et les poissons, il est de l’ordre d’un facteur trois chez les primates, et supérieur à six chez certains oiseaux (notamment les passereaux). Reptiles et poissons ont généralement des périodes de reproduction courtes et espacées, avec une production de spermatozoïdes par intermittence et donc un nombre de divisions cellulaires réduit par rapport à une production continue, ce qui a pour conséquence une moindre accumulation de mutations germinales chez les mâles.
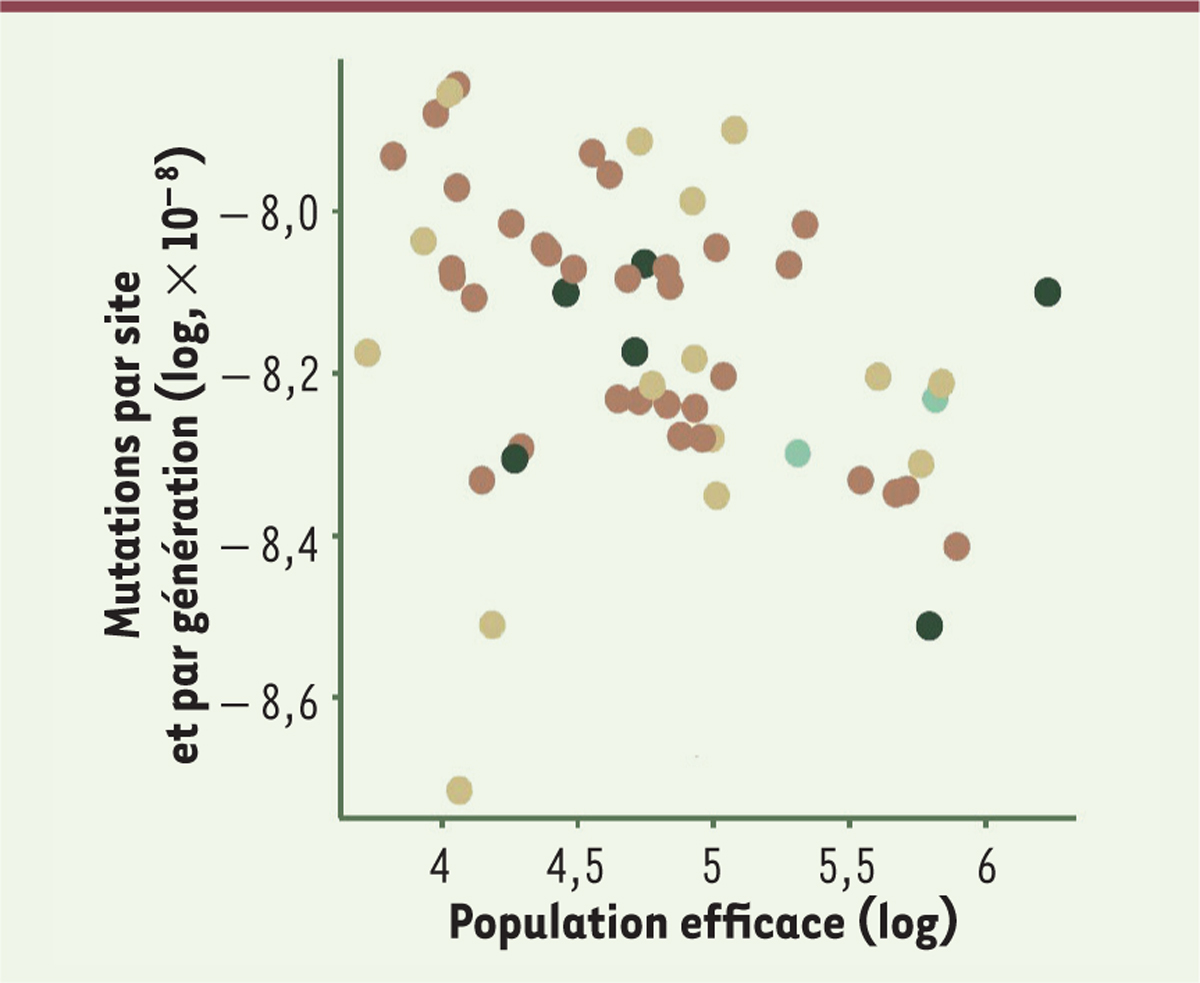
Il a été proposé que le taux de mutations germinales dépend de la taille effective de la population
3
[
8
], la sélection étant plus efficace sur de grandes populations et tendant ainsi à réduire ce taux. Cette taille effective peut être estimée à partir du taux de substitutions [
9
], et la
Figure 2
montre effectivement une (faible) corrélation négative entre ce paramètre et le taux de mutations par génération, comme attendu.
 | Figure 2.
Rapport entre le taux de mutation par génération et la taille effective de la population.
Les codes de couleur sont identiques à ceux de la
Figure 1
. La correìlation est relativement marginale (extrait partiel et modifieì de la figure 3 de [
4
]).
|
Si l’on s’intéresse enfin au taux de mutations par année (et non plus par génération), ce paramètre est très différent d’une espèce à l’autre. Il est notamment bien plus élevé chez les espèces domestiquées que chez les espèces sauvages ; les auteurs montrent que cette différence ne provient vraisemblablement pas d’un changement du processus de mutation dû aux conditions de l’élevage, mais de la diminution de l’âge de reproduction et donc du temps de génération : un nombre équivalent de mutations est ainsi concentré sur une durée plus courte. |
Les secrets des mutations
Si l’ADN est au centre de la vie, ses mutations sont le matériau de l’évolution, sans laquelle on ne peut rien comprendre à la biologie («
Nothing in biology makes sense except in the light of evolution
»
,
comme l’écrivait Theodosius Dobzhansky
4
il y a cinquante ans [
10
]). L’étude globale de ces mutations est longtemps restée hors de portée, et pendant des dizaines d’années, on a dû se limiter à l’analyse de celles qui sont à l’origine de maladies génétiques, ou à l’examen de petites zones de l’ADN présentant un intérêt particulier. Mais aujourd’hui, la rapidité, la précision et le coût abordable du séquençage des génomes entiers permettent d’avoir une vision globale de ces processus de variation. L’étude des génomes anciens permet même de remonter dans le temps et de tester directement des hypothèses en génétique des populations. Le catalogue très large établi par Bergeron
et al.
[
4
] n’a pas fini d’être utile, et, au-delà de ses premières conclusions, devrait servir de base à de nombreuses recherches.
|
L’auteur déclare n’avoir aucun lien d’intérêt concernant les données publiées dans cet article.
|
Footnotes |
1.
Jordan
B
.
Mutations somatiques et longévité.
.
Med Sci (Paris)
.
2022;
;
38
:
:723.
–
6
.
2.
Cagan
A
,
Baez-Ortega
A
,
Brzozowska
N
,
et al
.
Somatic mutation rates scale with lifespan across mammals.
.
Nature
.
2022;
;
604
:
:517.
–
24
.
3.
Tatsumoto
S
,
Go
Y
,
Fukuta
K
,
et al.
Direct estimation of de novo mutation rates in a chimpanzee parent offspring trio by ultra-deep whole genome sequencing.
.
Sci Rep
.
2017;
;
7
:
:13561.
.
4.
Bergeron
LA
,
Besenbacher
S
,
Zheng
J
,
et al
.
Evolution of the germline mutation rate across vertebrates.
.
Nature
.
2023;
;
615
;
:285.
–
91
.
5.
Jordan
B
.
Séquençage d’ADN, la fin d’un quasi-monople ?
Med Sci (Paris)
.
2023;
;
39
:
:469.
–
73
.
6.
Bergeron
LA
,
Besenbacher
S
,
Bakker
J
,
et al
.
The germline mutational process in rhesus macaque and its implications for phylogenetic dating.
.
Gigascience
.
2021;
;
10
;
giab029.
.
7.
de Manuel
M
,
Wu
FL
,
Przeworski
M
.
A paternal bias in germline mutation is widespread in amniotes and can arise independently of cell division numbers.
.
eLife
.
2022;
;
11
:
:e80008.
.
8.
Lynch
M.
Evolution of the mutation rate.
.
Trends Genet
.
2010;
;
26
:
:345.
–
352
.
9.
Wang
J
,
Santiago
E
,
Caballero
A
.
Prediction and estimation of effective population size.
.
Heredity
.
2016;
;
117
:
:193.
–
206
.
10.
Dobzhansky
T.
Nothing in Biology Makes Sense Except in the Light of Evolution.
.
American Biology Teacher
.
1973;
;
35
:
:125.
–
129
.
|