Les virus sont des « parasites » intracellulaires obligatoires, ce qui signifie qu’ils nécessitent des cellules pour se multiplier. Les cellules de notre organisme se sont adaptées afin de détecter la présence de ces virus et déclencher de puissantes défenses intracellulaires pour limiter leur multiplication [ 1 , 2 ]. Ces premières lignes de défense agissent en amont de l’immunité adaptative. Elles permettent une réponse antivirale rapide et avec un spectre large de reconnaissance [ 3 ]. Elles sont composées de l’immunité intrinsèque et de l’immunité innée. L’immunité intrinsèque repose sur la capacité de certaines protéines, exprimées de façon constitutive, à inhiber l’infection virale avant même l’activation de l’immunité innée. En parallèle, la présence dans une cellule d’éléments viraux, comme des acides nucléiques, est détectée par des récepteurs spécialisés, qui déclenchent une cascade de signalisation conduisant à l’activation de l’immunité innée, avec la sécrétion d’interférons [ 4 , 5 ]. Les interférons agissent comme des signaux d’alarme. Ils induisent une reprogrammation transcriptionnelle qui permet d’instaurer, dans les cellules infectées et leurs voisines, un état « antiviral » constituant un environnement hostile pour les virus, grâce à l’expression d’effecteurs antiviraux, des protéines capables d’inhiber certaines étapes du cycle de réplication virale [ 6 ].
Notre étude visait à d’identifier de nouveaux gènes impliqués dans la réponse intrinsèque ou la réponse immunitaire innée contre le virus de l’immunodéficience humaine de type 1 (VIH-1) [ 7 ]. Nous avons recherché, dans l’ensemble des gènes humains, ceux dont le produit avaient une activité antivirale. Ce criblage, à l’échelle du génome, a comporté trois étapes : 1) la production d’une population de cellules mutantes, chacune étant porteuse de mutations dans un seul gène, par la technique CRISPR/Cas9 ; 2) la sélection de cellules mutantes incapables d’inhiber l’infection par le VIH-1, malgré l’état antiviral induit par l’interféron ; et 3) l’identification des gènes mutés, par séquençage à haut débit de l’ADN.
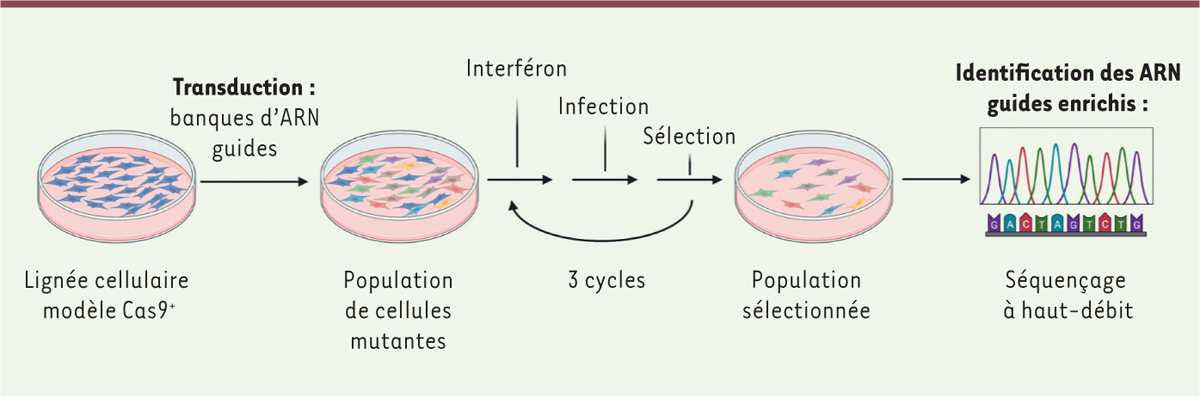
Des cellules humaines d’une lignée de glioblastome (T98G) ont été transduites avec des vecteurs lentiviraux codant l’endonucléase Cas9 et une banque d’environ 120 000 ARN guides différents, ciblant plus de 19 000 gènes humains (les ARN guides permettent de cibler l’activité de Cas9 sur des gènes donnés et de les rendre ainsi non fonctionnels) [ 8 ]. Les cellules ont été transduites de telle manière que chaque cellule exprime un seul ARN guide et soit donc invalidée pour un seul gène. La population de cellules mutantes a ensuite été traitée avec de l’interféron a pour induire un état antiviral, puis infectée avec un virus VIH-1 modifié génétiquement afin qu’il exprime une cassette de résistance à un antibiotique ; le traitement des cellules par l’antibiotique correspondant permettant de sélectionner les cellules ayant été infectées par le virus, malgré le traitement par l’interféron ( Figure 1 ) . Ce processus de sélection a été répété trois fois, en utilisant à chaque fois des antibiotiques différents afin de limiter le nombre de faux résultats positifs. Les ARN guides (et par conséquent leurs gènes cibles) présents dans les cellules sélectionnées ont été identifiés par la technique de PCR ( polymerase chain reaction ) et par séquençage nucléotidique à haut débit, ce qui a permis d’établir une liste de gènes contribuant potentiellement à l’immunité innée ou intrinsèque anti-VIH-1.
| Figure 1.
Stratégie du criblage génomique par la technique CRISPR/Cas9.
Les cellules d’une lignée cellulaire humaine exprimant l’endonucléase Cas9 (Cas9
+
) ont été transduites avec des vecteurs lentiviraux codant une banque d’ARN guides ciblant plus de 19 000 gènes, soit quasiment la totalité du génome humain (qui comporte environ 21 000 gènes). Ces cellules ont été transduites de façon à ce que chaque cellule exprime un seul ARN guide et soit donc invalidée seulement pour un gène unique. Dans cette population de cellules mutantes, celles incapables d’inhiber le VIH-1 malgré un pré-traitement avec de l’interféron α ont ensuite été sélectionnées. L’interféron permet normalement l’établissement d’un état cellulaire antiviral
via
l’induction de gènes codant des protéines effectrices antivirales et inhibant fortement la réplication du VIH-1. Pour ce criblage, les cellules pré-traitées à l’interféron ont été infectées avec un VIH-1 génétiquement modifié afin d’exprimer une cassette de résistance à un antibiotique. L’ajout de l’antibiotique a permis de sélectionner uniquement les cellules infectées par le virus, qui sont les seules capables de survivre en présence de l’antibiotique. Ce procédé de sélection a été réalisé sucessivement trois fois au total, en utilisant des cassettes de résistance à des antibiotiques différents. Les ARN guides enrichis dans les cellules sélectionnées ont été identifiés par séquençage nucléotidique à haut débit. Ainsi, les ARN guides les plus enrichis ciblent des gènes candidats potentiellement impliqués dans la défense antivirale (figure réalisée sur BioRender.com).
|
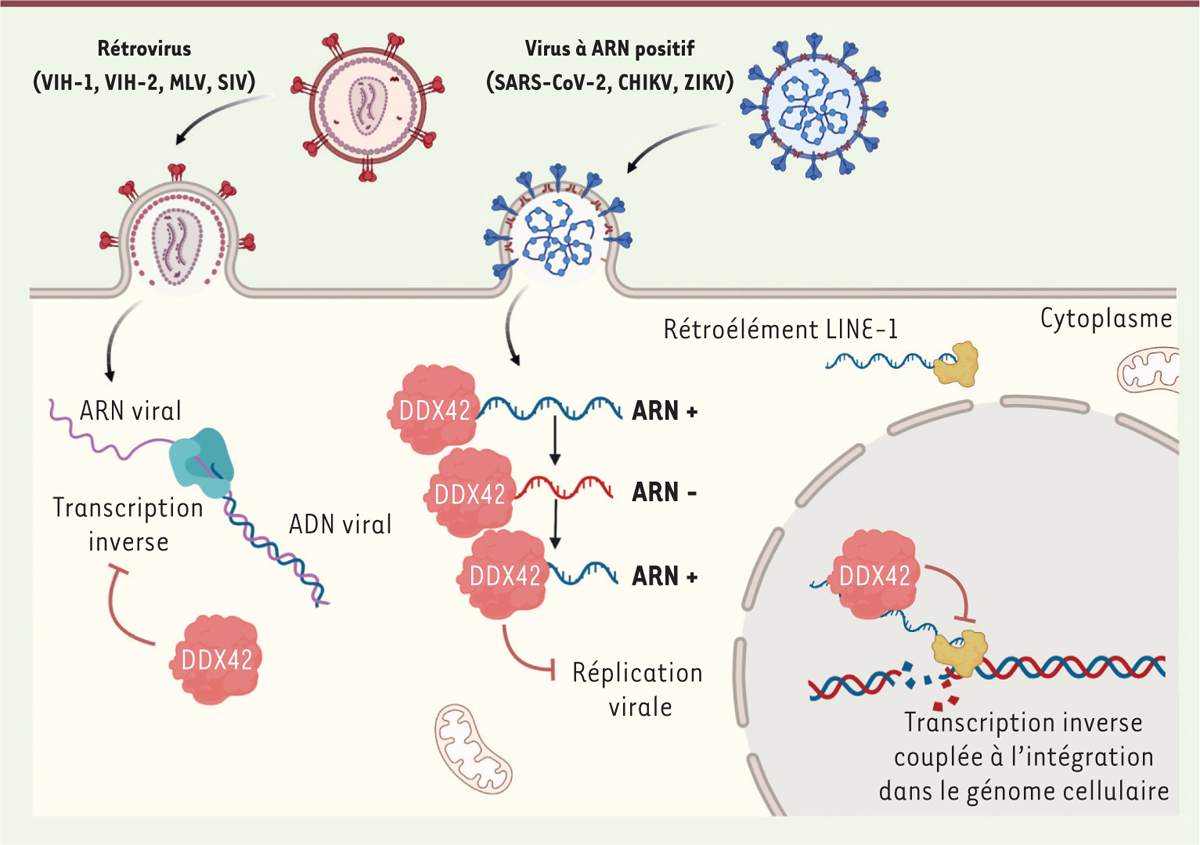
| Figure 2.
L’hélicase à ARN DDX42 est un inhibiteur antiviral intrinsèque.
DDX42 inhibe l’infection par des rétrovirus, notamment les virus de l’immunodéficience humaine de type 1 (VIH-1) et 2 (VIH-2), ainsi que par plusieurs virus à ARN positif, tels que le coronavirus 2 responsable du syndrome respiratoire aigu sévère (SARS-CoV-2), le virus Chikungunya (CHIKV) ou le virus Zika (ZIKV). DDX42, constitutivement exprimé dans les cellules, inhibe l’infection par le VIH-1 en bloquant l’étape de transcription inverse de son génome. Identifiée à proximité des sites de réplication du SARS-CoV-2, la protéine DDX42 se lie aux ARN viraux du SARS-CoV-2 et du CHIKV, et inhibe leur réplication. De plus, DDX42 se lie aux ARN des rétroéléments du type LINE-1, et inhibe leur rétrotransposition, qui nécessite une étape de transcription inverse couplée à l’intégration dans le génome de la cellule hôte (figure réalisée sur BioRender.com).
|
La validation (par une approche CRISPR/Cas9) de l’implication de ces gènes candidats dans la réponse antivirale a permis d’identifier l’hélicase à ARN DDX42 comme un inhibiteur intrinsèque de l’infection par le VIH-1 [ 7 ]. Les hélicases à ARN, présentes dans l’ensemble du monde vivant, constituent une grande famille de protéines impliquées dans le métabolisme des acides nucléiques [ 9 ]. Elles modifient la structure des acides nucléiques ainsi que leurs interactions avec des protéines. Les hélicases à ARN sont donc essentielles, notamment pour l’expression des gènes [ 10 ]. La participation de l’hélicase DDX42 à des complexes ribonucléoprotéiques impliqués dans l’épissage des ARN messagers avait précédemment été proposée, mais son rôle dans l’épissage n’avait pas été caractérisé [ 11 ]. L’étude de ses propriétés biochimiques avait établi son activité hélicase, ainsi que sa capacité à remodeler des structures d’ARN et à déplacer des protéines se liant aux ARN [ 12 ]. Nous avons pu prouver l’activité antivirale de DDX42, en diminuant son expression (grâce à la technique CRISPR/Cas9 ou à la technique d’interférence ARN) ou en la surexprimant. Nous avons constaté que l’expression endogène de DDX42 inhibait l’infection par le VIH-1, non seulement dans des lignées cellulaires modèles, mais également dans les cellules cibles physiologiques du virus : les lymphocytes T CD4 + et les macrophages (différenciés à partir de monocytes sanguins).
Nous sommes parvenus à identifier l’étape du cycle de réplication du VIH-1 sur laquelle agit DDX42. Nous avons en effet observé, par PCR quantitative, une augmentation significative de la quantité d’ADN viral (synthétisé par transcription inverse du génome viral) lorsque l’expression de DDX42 était inhibée.
DDX42 inhibe donc l’infection par le VIH-1 en ciblant l’étape de transcription inverse de son génome, ce qui se répercute sur les étapes suivantes du cycle de réplication du virus : import de l’ADN viral dans le noyau, intégration dans le génome humain, et production de nouvelles particules virales. En réalisant des expériences de transcription inverse in vitro , nous avons pu montrer que la présence de la protéine DDX42 purifiée (produite par des bactéries) dans un système minimal, inhibe la transcription inverse du génome du VIH-1, mimant ainsi l’effet observé dans les cellules infectées par le virus. Nous avons constaté que DDX42 inhibe également d’autres rétrovirus, tels que le virus de l’immunodéficience simienne (SIV) ou le virus de la leucémie murine (MLV). Des travaux de recherche antérieurs avaient montré que les effecteurs antiviraux ciblant la transcription inverse tendent à inhiber également la multiplication des rétroéléments du génome, des éléments mobiles présents dans l’ADN de nos cellules, transcrits par la machinerie cellulaire de l’hôte et capables de s’intégrer à nouveau dans le génome par transcription inverse [ 13 , 14 ]. Comme nous l’avons observé pour le VIH-1, la surexpression de DDX42 inhibe la rétrotransposition des rétroéléments du type LINE-1. Ainsi, en plus d’être un inhibiteur de rétrovirus, DDX42 participe également au maintien de l’intégrité du génome en réprimant son invasion par les rétroéléments.
Nous avons également cherché à préciser le spectre d’activité de DDX42 sur d’autres familles de virus. L’activité antivirale de DDX42 ne semble pas inhiber l’infection par trois virus à ARN de polarité négative, le virus de la grippe influenza A (orthomyxovirus), le virus de la stomatite vésiculaire (rhabdovirus) et le virus de la rougeole (paramyxovirus), ce qui exclut la possibilité que la modulation de l’expression de cette hélicase affecte tous les virus de manière aspécifique. En revanche, la déplétion de DDX42 favorise l’infection par des virus à ARN de polarité positive, dont l’alphavirus Chikungunya (CHIKV), les coronavirus SARS-CoV-2 (responsable de la pandémie actuelle de Covid-19) et HCoV-229E, et, dans une moindre mesure, certains flavivirus tels que le virus Zika, le virus de la fièvre jaune et le virus de l’encéphalite japonaise. L’effet antiviral de DDX42 est d’ailleurs plus marqué sur les virus Chikungunya, SARS-CoV-2 et HCoV-229E que sur le VIH-1. Des essais de ligation de proximité ont montré que DDX42 était situé à proximité de la capside du VIH-1 ainsi que des sites de réplication du SARS-CoV-2. Étant donné que DDX42 est une protéine qui se lie aux ARN, nous avons supposé qu’elle pouvait interagir avec les ARN viraux. En immunoprécipitant les ARN associés à DDX42, nous avons constaté que cette protéine interagissait spécifiquement avec les ARN du SARS-CoV-2, du CHIKV et des rétroéléments du type LINE-1, mais pas avec les ARN du virus influenza A. Cette corrélation entre la capacité de DDX42 à se lier à certains ARN viraux et son activité antivirale est un argument en faveur d’une action directe de DDX42 contre ces virus.
Nous avons donc identifié la protéine cellulaire DDX42, une hélicase à ARN peu étudiée jusqu’à présent, comme étant un inhibiteur antiviral intrinsèque à large spectre. Il reste à comprendre ce qui détermine la liaison de cette protéine aux ARN viraux. Comme pour d’autres hélicases, la liaison de DDX42 aux ARN pourrait dépendre de la présence de certains motifs spécifiques ou bien de structures secondaires de l’ARN [ 15 , 16 ]. On sait que DDX42 se lie à certaines structures secondaires, comme les quartets de guanines (G4) [ 17 ], qui sont notamment présents dans les génomes viraux et sont impliqués dans la régulation post-transcriptionnelle [ 18 , 19 ]. Cependant, on ignore encore si l’activité antivirale de DDX42 passe réellement par la liaison aux G4 des génomes viraux dans les cellules infectées. L’identification des déterminants de liaison de DDX42 permettrait d’élucider le mécanisme d’action de cette hélicase à ARN, et de mieux comprendre quelles faiblesses l’immunité intrinsèque exploite pour inhiber les infections virales.