Le pergélisol, un sol gelé en permanence, peut préserver des organismes vivants durant des centaines de milliers d’années. On y trouve des bactéries et des eucaryotes, y compris des eucaryotes multicellulaires, cultivables à partir d’échantillons anciens. Outre les organismes cellulaires, il a été montré dès 2014 que certains virus dits « géants » (par la taille des virions et celle de leur génome) pouvaient également être réactivés à partir de prélèvements issus de ce type de milieu. Deux virus, Pithovirus sibericum [ 1 ] et Mollivirus sibericum [ 2 ], premiers membres de familles virales distinctes, ont en effet été isolés à partir d’une couche de pergélisol datant de plus de 30 000 ans. Pithovirus sibericum possède à ce jour la plus grande particule virale connue, dont la structure ovoïde mesure 1,5 µm en longueur et 0,5 µm en largeur [ 1 ]. Son génome à ADN double brin circulaire est complexe, avec plus de 600 000 paires de bases et de nombreux gènes de fonction inconnue. Ces virus sont pathogènes pour les amibes du genre Acanthamoeba , des organismes unicellulaires se nourrissant par phagocytose. Pas de danger direct pour l’homme donc, mais par analogie, cette découverte à fait couler beaucoup d’encre sur le risque sanitaire lié à la réactivation de virus anciens résultant de la fonte du pergélisol. De nombreuses amibes ont également pu être cultivées à partir d’échantillons anciens [ 3 ].
Cependant, on ne sait quasiment rien de l’abondance et de la diversité des virus géants dans le pergélisol. En effet, la culture cellulaire et virale ne permet d’étudier qu’une petite fraction d’un microbiome donné. La métagénomique 1 est un excellent moyen complémentaire d’explorer les microbiomes mal connus. En analysant les séquences nucléotidiques de l’ADN environnemental « total », on accède à une grande partie de la diversité génétique d’un milieu donné, y compris les virus [ 4 ] ( → ).
(→) Voir la Synthèse de E. Olo Ndela et al ., m/s n° 12, décembre 2022, page 999
Toutefois, peu d’études de métagénomique ont concerné les virus anciens du pergélisol, à l’exception notable d’une analyse de la couche active (les premiers centimètres de sol qui dégèlent l’été) et du pergélisol sous-jacent en Suède [ 5 ], qui a mis en évidence près de 2 000 populations virales différentes. Mais ces virus sont tous des virus de procaryotes (des bactériophages), et cette étude n’apporte donc pas d’information sur les virus qui infectent les eucaryotes. En revanche, la métagénomique des milieux marins a révélé l’existence de nombreux virus géants. L’analyse de milliers de séquences nucléotidiques associées aux Mimiviridae et Phycodnaviridae , des virus d’algues et de protozoaires, a montré en effet que ces virus sont abondants et ubiquitaires dans les océans, où ils pourraient jouer un rôle important dans les cycles biogéochimiques par l’interaction avec leurs hôtes [ 6 ]. Peu de traces en revanche des Pithoviridae , famille à laquelle appartient Pithovirus sibericum , sauf dans des sédiments marins prélevés dans l’océan Atlantique à 3 000 m de profondeur, où l’on a trouvé une grande diversité de séquences virales associées à cette famille [ 7 ].
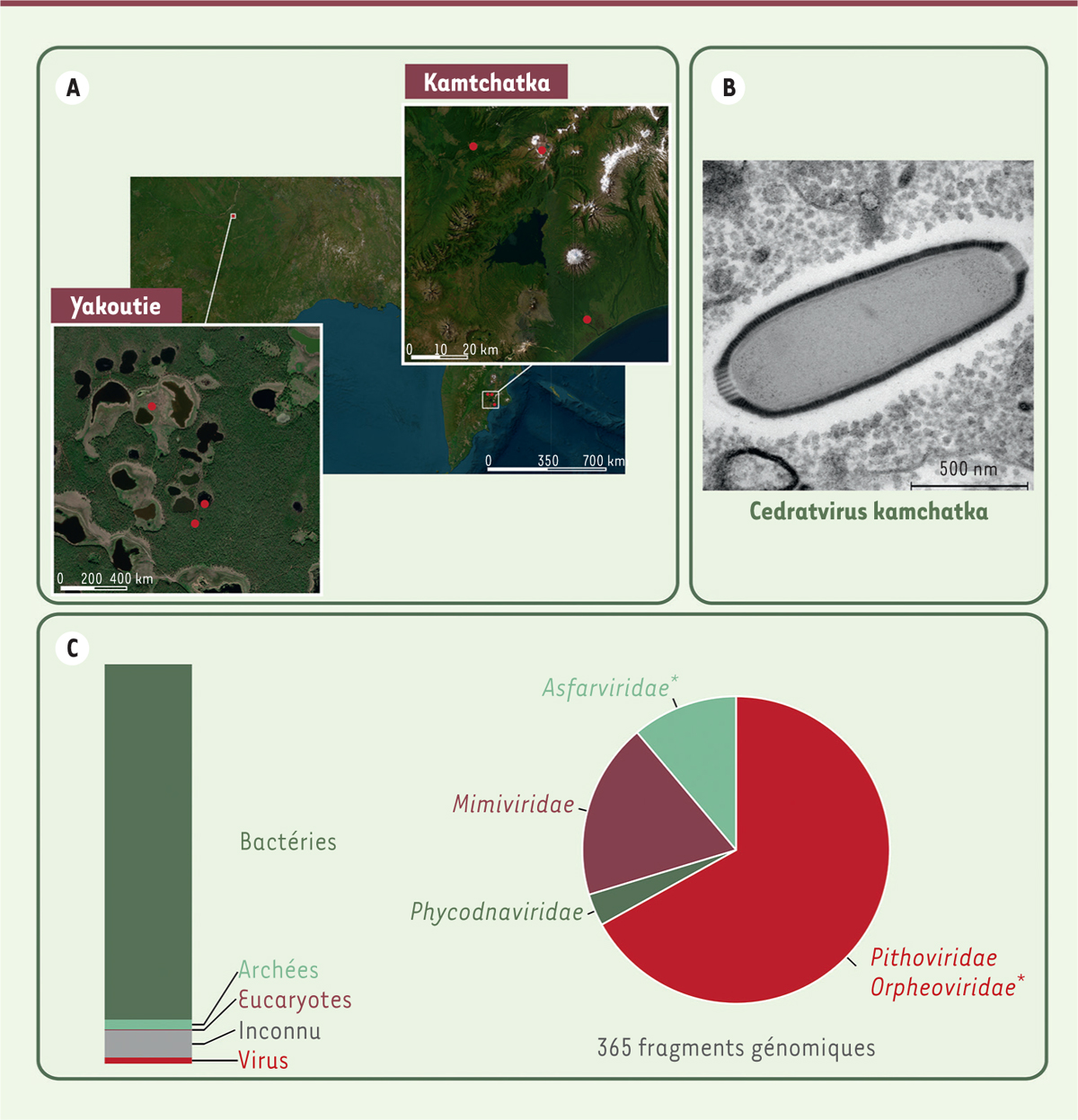
Notre équipe de recherche a décidé d’explorer le pergélisol par une analyse métagénomique, afin d’y découvrir d’éventuelles populations de virus géants [ 8 ]. Cette étude inclut huit échantillons provenant de Yakoutie, en Sibérie, issus de trois forages et prélevés à des profondeurs différentes ( Figure 1A ) . L’échantillon le moins profond, prélevé à 6 mètres de profondeur, n’était pas tout à fait gelé, contrairement aux autres échantillons. Quant au plus profond, prélevé à 19 mètres de profondeur, il est daté de 42 000 ans. Nous avons également étudié l’ADN environnemental d’échantillons de surface provenant du Kamtchatka, dans lesquels nous avions précédemment isolé un autre virus de la famille des Pithoviridae : Cedratvirus kamchatka ( Figure 1B ) [ 9 ].
| Figure 1.
A.
Localisation géographique des échantillons de pergélisol prélevés en Russie, dans lesquels les virus géants ont été identifiés par analyse métagénomique. Les lieux de prélèvement (en rouge) et les barres d’échelle (en blanc) sont indiqués sur les photographies satellites.
B.
Photographie en microscopie électronique d’une particule virale de
Cedratvirus kamchatka
, membre de la famille des
Pithoviridae
(image fournie par Lionel Bertaux, UMR7256).
C.
À gauche : abondances des différents organismes cellulaires et virus estimées par métagénomique. À droite : les 365 fragments génomiques viraux qui ont pu être classés sont présentés selon la famille virale à laquelle ils sont associés. Les astérisques indiquent de nouvelles familles virales, proches du groupe défini.
|
L’analyse métagénomique permet en théorie d’accéder à tous les organismes présents dans ces échantillons, mais rend complexe l’identification d’un groupe d’intérêt donné, les virus dans ce cas. Pour étudier les viromes, une première étape consiste donc généralement à filtrer les échantillons, à 0,2 µm 2 , par exemple, pour éliminer les cellules et ne conserver que les virus. Mais cette procédure ne convient pas à l’étude des virus géants, dont la taille est trop proche de celle des cellules ! Après avoir déterminé les séquences de l’ADN total des échantillons, nous avons donc d’abord assemblé les lectures obtenues en plus grands fragments continus, appelés contigs , dont la plupart appartiennent à des bactéries telles que les Protéobactéries, les Actinobactéries, ou encore les Firmicutes [ 10 ]. Pour identifier les contigs de virus géants, il a fallu ensuite reconnaître ceux contenant des gènes spécifiques des virus géants connus. Cependant, les virus ont la particularité d’avoir des génomes qui varient beaucoup d’une espèce à l’autre. Nous avons donc utilisé des méthodes de recherche de gènes homologues « distants », fondées sur des profils HMM ( hidden Markov models ). Nous avons également recherché les gènes potentiellement d’origine cellulaire par la méthode BLASTp ( basic local alignment search tool for proteins ), fondée sur la similarité de séquence polypeptidique des protéines codées par ces gènes. En combinant les résultats de ces deux analyses (présence de gènes de virus géants connus et de gènes d’origine cellulaire), nous avons ainsi révélé la présence, dans ces échantillons, de fragments génomiques de virus géants (avec un taux de contamination par des séquences d’ADN cellulaire particulièrement faible).
Près de 2 000 séquences d’ADN ont ainsi été identifiées comme provenant de virus géants, dont 365 ont pu être classées dans des familles ( Figure 1C ) . Ces séquences sont réparties très inégalement entre les échantillons. Un échantillon de pergélisol de 42 000 ans sort du lot, avec 721 séquences provenant de virus géants, ce qui représente, d’après nos estimations, 12 % de l’abondance des différents organismes dont l’ADN a pu être séquencé. Cet échantillon est également le plus riche en cellules eucaryotes, les hôtes des virus géants. Parmi ces virus géants, on trouve des Mimiviridae , un groupe de virus abondants dans tout type d’environnement, en particulier dans les océans. Étonnamment, on y trouve également de nouveaux et nombreux groupes de virus associés aux Pithoviridae ( Figure 1C ) , dont certains ont divergé très tôt dans l’histoire évolutive de ces virus. Pour nous convaincre de la plus grande diversité des Pithoviridae dans le pergélisol, nous avons récupéré les données de métagénomique des bases de données mondiales. La comparaison, pour chaque environnement, du nombre de séquences associées aux Pithoviridae a montré que nos échantillons de pergélisol contiennent effectivement un nombre anormalement élevé de Pithoviridae . C’est aussi le cas d’échantillons issus de sols de forêts et d’eaux usées, milieu dans lequel différents Pithoviridae avaient précédemment été isolés [ 11 , 12 ].
Lorsqu’un organisme est suffisamment abondant dans un milieu, l’analyse métagénomique peut révéler de grands fragments de son génome, voire son génome complet. L’abondance des Pithoviridae dans le pergélisol nous a permis d’analyser de grands fragments génomiques, supérieurs à 500 kilobases, de différents virus associés à cette famille. L’un d’eux correspond avec certitude à un génome complet, car ce génome de 1,6 mégabases est circulaire. Nous avons appelé ce virus ancien, daté de 42 000 ans, Hydrivirus . Paradoxalement, il n’y a pas nécessairement de concordance entre les résultats de l’analyse métagénomique d’un échantillon et les virus identifiés par la mise en culture cellulaire de cet échantillon. Notamment, un virus, même très abondant dans un échantillon, ne sera jamais cultivé en l’absence de la cellule hôte adéquate, souvent inconnue. À l’inverse, il arrive que des virus isolés grâce à la mise en culture d’échantillons ne soient pas retrouvés par l’analyse métagénomique. Cela peut s’expliquer par le fait que des virus, même peu abondants dans l’échantillon, peuvent se multiplier efficacement avec leur cellule hôte en culture. Notre objectif est désormais de tenter d’isoler Hydrivirus , d’identifier son hôte, et d’étudier son cycle de réplication virale.