| |
| Med Sci (Paris). 39(1): 20–22. doi: 10.1051/medsci/2022189.Autophagie et co-infection
«
C’est double plaisir que de tromper le trompeur
»
*Aude Lavedrine,1 Guénaëlle Lamiral,1 Aurore Rozières,1 Christophe Viret,1 and Mathias Faure1** 1Centre international de recherche en infectiologie (CIRI), Université de Lyon, Inserm U1111, Université Claude Bernard Lyon 1, CNRS UMR5308, École normale supérieure de Lyon
,
Lyon
,
France MeSH keywords: Humains, Plaisir, Co-infection, Autophagie |
Lors d’une infection par un agent pathogène, l’hôte met en place une réponse immunitaire afin de limiter la prolifération du micro-organisme jusqu’à l’éradiquer. À l’échelle de la cellule infectée, l’autophagie, connue pour son rôle dans l’homéostasie cellulaire [
1
] (
→
), est l’un des premiers évènements cellulaires de la réponse anti-microbienne. En effet, ce processus, actif et fonctionnel dans toutes les cellules nucléées, permet à chacune d’elles de lutter de manière intrinsèque et autonome contre les micro-organismes intracellulaires, quelle que soit leur nature. Cependant, nombreux sont les agents pathogènes qui se sont adaptés pour échapper à l’autophagie, ou même pour l’utiliser à leur avantage [
2
,
3
] (
→
).
(→) Voir l’Éditorial de P. Codogno,
m/s
n° 4, avril 2009, page 323
(→) Voir les Synthèses de P. Lapaquette
et al
.,
m/s
n° 3, mars 2017, page 305, et P.E. Joubert
et al.
,
m/s
n° 1, janvier 2011, page 41
À cet égard, la conséquence de l’interaction d’un agent pathogène avec l’autophagie sur le devenir d’un second agent pathogène a fait l’objet d’une étude récente révélant l’influence que celle-ci exerce dans le cadre de co-infections [
4
].
L’autophagie, terme inventé en 1963 par Christian de Duve (prix Nobel en 1974) pour définir la dégradation de composants internes à la cellule par la voie du lysosome, fut très tôt supposée être un processus sélectif, sans que l’on ait compris cependant comment certains éléments sont spécifiquement reconnus puis dégradés par cette machinerie autophagique [
5
]. C’est l’entrée dans l’ère moléculaire de l’autophagie, grâce à la découverte par Yoshinori Ohsumi (prix Nobel en 2016) des gènes autophagiques
ATG,
qui permit la compréhension des rouages moléculaires de la formation des autophagosomes et des évènements permettant le ciblage sélectif concomitant de cargos cytosoliques destinés à la dégradation [
5
]. Cette sélectivité dépend de protéines appelées récepteurs autophagiques, capables de reconnaître spécifiquement un cargo (qui, dans le cadre d’une infection, peut être un micro-organisme pathogène entier, ou certains de ses composants) et de le cibler vers une vésicule en formation, qui s’allonge et se clôt pour séquestrer le cargo au sein d’une vacuole à double membrane néoformée : l’autophagosome. Ce dernier fusionne ensuite avec un lysosome pour former un autolysosome, au sein duquel le cargo est dégradé. La dégradation de micro-organismes intracellulaires par l’autophagie est appelée xénophagie.
|
Autophagie et simple infection
De nombreuses bactéries intracellulaires, comme
Salmonella enterica
Typhimurium, responsable de douleurs abdominales, de fièvre ou encore de diarrhée, peuvent être efficacement dégradées par autophagie [
6
]. À cette fin, ces bactéries, après avoir été détectées par la cellule, peuvent être marquées par des ubiquitines. Cette « étiquette » présente à la surface du micro-organisme pathogène dans le cytosol est alors reconnue par des récepteurs autophagiques, parmi lesquels SQSTM1 (séquestosome-1, ou protéine de liaison à l’ubiquitine p62), CALCOCO2 (
calcium binding and coiled-coil domain 2
), TAX1BP1 (
Tax1-binding protein 1
) ou encore optineurine, qui ciblent le micro-organisme vers la machinerie autophagique pour sa dégradation [
7
] (
→
).
(→) Voir la Nouvelle de P. Verlhac
et al
.,
m/s
n° 6-7, juin-juillet 2015, page 594
La xénophagie étant très efficace, nombreux sont les micro-organismes pathogènes qui ont développé des stratégies leur permettant d’échapper à l’autophagie, ou même de l’utiliser à leur avantage. Parmi eux, le virus de la rougeole (une maladie infectieuse transmissible par voie respiratoire) induit l’autophagie dans les cellules qu’il infecte, ce qui lui permet d’optimiser sa réplication. Ce virus induit l’autophagie
via
au moins trois voies successives et indépendantes [
8
]. Après sa liaison à son récepteur cellulaire, il induit une première vague d’autophagie précoce, mais transitoire [
9
]. Une seconde vague d’autophagie est cependant rapidement déclenchée à la suite de l’accumulation de protéines virales synthétisées lors de la réplication du virus [
10
]. Une autophagie active est alors maintenue dans la cellule infectée grâce à la formation par le virus de la rougeole de syncytiums, des cellules multinucléées résultant de la fusion de cellules infectées avec des cellules pas encore infectées [
11
]. Le maintien dans le temps d’une activité autophagique intense permet au virus de la rougeole de prolonger la durée de vie de la cellule syncytiale, ce qui contribue à optimiser la réplication virale [
11
].
|
Autophagie et co-infection
Alors que l’infection par un agent pathogène est souvent associée à l’établissement d’infections secondaires, on sait peu de choses concernant l’impact d’un agent pathogène sur le devenir d’un second micro-organisme à l’échelle de la cellule, et encore moins concernant la place de l’autophagie en cas de co-infection. Aussi nous sommes nous posé la question de l’impact de l’induction de l’autophagie par le virus de la rougeole sur une infection secondaire par
Salmonella enterica
Typhimurium, deux agents pathogènes aux destins individuels opposés au regard de l’autophagie.
Étonnamment, bien que
Salmonella
Typhimurium soit une bactérie efficacement dégradée par autophagie [
6
], lorsque cette bactérie infecte des cellules préalablement infectées par le virus de la rougeole, au sein desquelles l’autophagie est intense et dégradative, elle se réplique très activement, ce qui indique qu’elle échappe à l’autophagie [
4
]. Cette hyper-réplication de la bactérie prend place spécifiquement dans le cytosol des syncytiums produits par le virus de la rougeole. En effet, malgré l’étiquetage de
Salmonella
Typhimurium par des ubiquitines dans le cytosol du syncytium, les bactéries ne sont pas ciblées vers les autophagososomes, ce qui suggère l’existence d’un défaut de l’étape intermédiaire impliquant les récepteurs autophagiques. L’analyse de ces derniers a permis de montrer que deux des récepteurs autophagiques impliqués dans la xénophagie dirigée contre
Salmonella
, SQSTM1 et TAX1BP1, sont fortement « consommés » par l’autophagie induite par le virus de la rougeole [
4
]. Ainsi, la bactérie
Salmonella
Typhimurium, en infectant une cellule préalablement infectée par le virus de la rougeole, est-elle confrontée à un environnement intracellulaire moins apte à assurer son ciblage vers les autophagosomes en formation, ce qui facilite son échappement à l’autophagie et favorise sa réplication intracellulaire. En effet, la simple surexpression de l’un des récepteurs autophagiques dont la concentration est diminuée en condition de co-infection, SQSTM1 ou TAX1BP1, suffit à relocaliser
Salmonella
Typhimurium dans les autophagosomes et à contrôler sa réplication.
Cependant, de manière tout aussi étonnante, cet environnement ne bénéficie pas à toutes les bactéries sensibles à l’autophagie. Ainsi, à l’inverse de ce qu’on observe pour
Salmonella
Typhimurium, des cellules infectées par le virus de la rougeole contrôlent bien plus efficacement une autre bactérie à Gram négatif,
Shigella flexneri.
Il a été montré que pour optimiser sa réplication intracellulaire,
Shigella flexneri
utilise la protéine SQSTM1 [
12
]. Ainsi, alors que la diminution de la quantité de SQSTM1 associée à l’autophagie soutenue des cellules co-infectées par le virus de la rougeole favorise la réplication intracellulaire de
Salmonella
Typhimurium, elle compromet celle de
Shigella flexneri
(
Figure 1
).
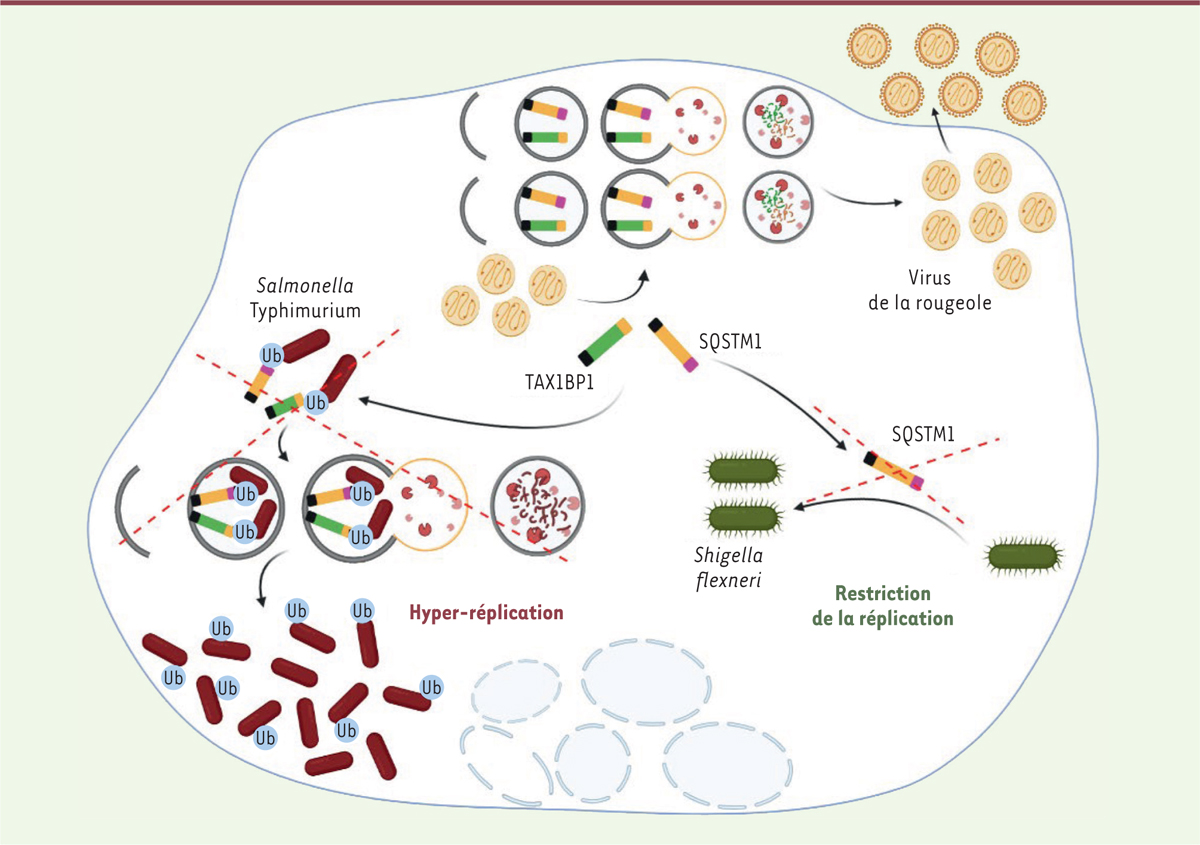
 | Figure 1.
Place de l’autophagie induite par le virus de la rougeole lors d’une co-infection par
Salmonella enterica
Typhimurium et
Shigella flexneri
. L’infection par le virus de la rougeole induit l’autophagie, que le virus exploite afin d’optimiser sa réplication intracellulaire. Cette autophagie consomme les récepteurs autophagiques SQSTM1 et TAX1BP1, dont la baisse de concentration ne permet plus d’assurer le ciblage de la bactérie
Salmonella
Typhimurium vers la machinerie autophagique. La bactérie peut alors se répliquer massivement dans le syncytium.
Shigella flexneri
, quant à elle, utilise SQSTM1 pour optimiser sa réplication en condition de simple infection. Aussi, une co-infection avec le virus de la rougeole, qui dégrade SQSTM1, restreint la capacité de prolifération intracellulaire de la bactérie. Pour simplifier la figure, les bactéries
Salmonella
Typhimurium et
Shigella flexneri
sont représentées au sein de la même cellule co-infectée avec le virus de la rougeole, mais ces deux bactéries doivent être considérées indépendamment (voir texte). Ub : ubiquitine. Illustration créée à partir du logiciel Biorender.com.
|
|
Malgré des avancées majeures dans la compréhension de la relation entre autophagie et agents infectieux durant ces dernières années, notamment grâce à une meilleure définition du rôle des récepteurs autophagiques, de nombreuses questions demeurent, en particulier en contexte de co-infection. Dans le cadre de la rougeole, la manipulation de l’autophagie par le virus à l’échelle cellulaire contribue-t-elle à l’établissement d’infections bactériennes responsables des complications observées chez certaines personnes ? D’autres (ou tous les ?) contextes de co-infection influent-ils significativement sur le comportement infectieux d’agents pathogènes, à l’instar de l’infection par le virus de la rougeole ? La dégradation autophagique de composants autres que les récepteurs autophagiques contribue-t-elle à décider du devenir des agents co-infectants ? Au-delà d’une primo-infection, la modulation de l’autophagie induite par des stress ou des facteurs génétiques témoigne-t-elle d’autant de facteurs de susceptibilité à l’établissement d’infections responsables de complications ? Ainsi, des micro-organismes pathogènes infectant une cellule déjà corrompue par un « tiers » trouvent un environnement qui leur ouvre des possibilités dont ils peuvent tirer avantage. Comme dans la fable (II, 15) de Jean de La Fontaine, où un vieux coq feint de céder à Maître renard, n’est-ce pas
« double plaisir que de tromper le trompeur »
?
|
Les auteurs déclarent n’avoir aucun lien d’intérêt concernant les données publiées dans cet article.
|
Nos travaux de recherche sur l’autophagie sont subventionnés par la FRM, l’ANR et l’Association François Aupetit (AFA).
|
1.
Codogno
P.
L’autophagie, dégrader plus pour vivre plus… mais attention !
.
Med Sci (Paris).
2009;
;
25
:
:323.
–
324
.
2.
Lapaquette
P
,
Nguyen
HTT
,
Faure
M
.
L’autophagie garante de l’immunité et de l’inflammation « Tout est bien, tout va bien, tout va pour le mieux qu’il soit possible».
.
Med Sci (Paris).
2017;
;
33
:
:305.
–
311
.
3.
Joubert
PE
,
Pombo Grégoire
I
,
Meiffren
G
,
et al.
Autophagie et pathogènes : « Bon appétit Messieurs ! ».
.
Med Sci (Paris).
2011;
;
27
:
:41.
–
47
.
4.
Claviere
M
,
Lavedrine
A
,
Lamiral
G
,
et al
.
Measles virus-imposed remodeling of the autophagy machinery determines the outcome of bacterial coinfection.
.
Autophagy.
2022
;
:1.
–
15
.
5.
Kirkin
V
,
McEwan
DG
,
Novak
I
,
et al.
A role for ubiquitin in selective autophagy.
.
Mol Cell.
2009;
;
34
:
:259.
–
269
.
6.
Birmingham
CL
,
Smith
AC
,
Bakowski
MA
,
et al.
Autophagy controls Salmonella infection in response to damage to the Salmonella-containing vacuole.
.
J Biol Chem.
2006;
;
281
:
:11374.
–
11383
.
7.
Verlhac
P
,
Viret
C
,
Faure
M
.
NDP52, autophagie et pathogènes : « Et le combat cessa faute de combattants ».
.
Med Sci (Paris).
2015;
;
31
:
:594.
–
597
.
8.
Rozières
A
,
Viret
C
,
Faure
M
.
Autophagy in measles virus infection.
.
Viruses.
2017
;
:9.
.
9.
Joubert
P-E
,
Meiffren
G
,
Grégoire
IP
,
et al.
Autophagy induction by the pathogen receptor CD46.
.
Cell Host Microbe.
2009;
;
6
:
:354.
–
366
.
10.
Grégoire
IP
,
Richetta
C
,
Meyniel-Schicklin
L
,
et al.
IRGM is a common target of RNA viruses that subvert the autophagy network.
.
PLoS Pathogens.
2011;
;
7
:
:e1002422.
.
11.
Richetta
C
,
Grégoire
IP
,
Verlhac
P
,
et al.
Sustained autophagy contributes to measles virus infectivity.
.
PLoS Pathogens.
2013;
;
9
:
:e1003599.
.
12.
Lobato-Márquez
D
,
Krokowski
S
,
Sirianni
A
,
et al.
A requirement for septins and the autophagy receptor p62 in the proliferation of intracellular Shigella.
.
Cytoskeleton (Hoboken).
2019;
;
76
:
:163.
–
172
.
|