| |
| Med Sci (Paris). 38(12): 1016–1027. doi: 10.1051/medsci/2022171.Des virus bénéfiques pour les plantes et les animaux Camille Heisserer,1* Marc-André Selosse,2** and Jean-Michel Drezen1*** 1Institut de recherche sur la biologie de l’insecte, UMR 7261 CNRS-Université de Tours, Parc de Grandmont37200Tours
,
France 2Institut de systématique, évolution, biodiversité, UMR 7205 MNHN-CNRS-SU-EPHE-UA
,
12 rue Buffon75005Paris
,
France |
Vignette (© Philippe Roingeard).
Durant la pandémie de Covid-19 (
coronavirus disease 2019
) survenue en 2020, les connaissances sur les virus et leur évolution se sont popularisées. Compte-tenu du coût humain de la pandémie, il peut paraître maladroit d’évoquer les bénéfices que les virus apportent parfois sur le long terme aux organismes qu’ils infectent. Pourtant, beaucoup de contributions positives des virus aux défenses, à la physiologie, ou à l’adaptation des organismes, ont été décrites et constituent une facette non négligeable du monde viral.
|
Des virus bénéfiques pour les plantes La connaissance des virus bénéfiques des plantes reste très limitée. Les virus pathogènes ont permis l’émergence du concept de virus : le biologiste Dmitri Ivanovski montra en 1898 que l’agent transmettant la mosaïque du tabac passait à travers des filtres de très petite taille et avait donc une taille inférieure à celle des bactéries. Le microbiologiste Martinus Beijerinck le nomma « virus ». Si de nombreuses autres maladies virales sont connues, les exemples de virus favorables sont disparates.
Grâce notamment aux travaux de Marilyn Roossinck (Université de Pennsylvanie), il a été établi que les tissus végétaux en apparence sains comportent des virus cytoplasmiques variés, à ARN double-brin ou simple-brin, ou encore à ADN simple-brin. Certains de ces virus, qualifiés de persistants, ne se propagent jamais d’une cellule à l’autre mais se répartissent entre cellules filles lors de la division cellulaire et sont en faible nombre dans chaque cellule [
1
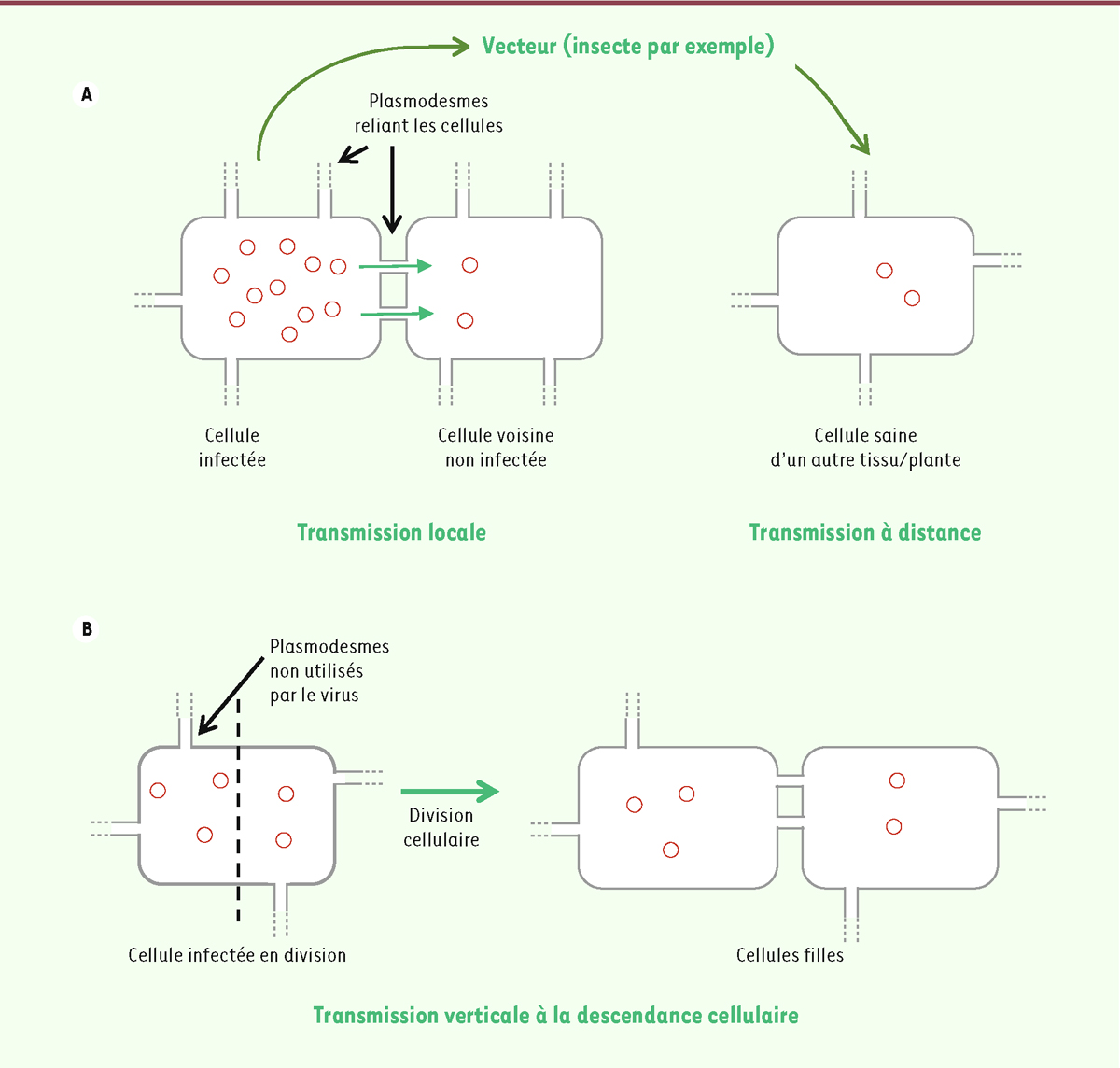
]. Les virus persistants représentent plus de la moitié de l’ensemble des virus présents dans la cellule, et leur transmission strictement verticale diffère radicalement de celle des virus pathogènes qui se propagent au sein de la plante grâce aux plasmodesmes, ces communications intercellulaires propres aux plantes (
Figure 1
). La transmission intergénérationnelle des virus persistants, par le biais des gamètes, avoisine 100 % et sélectionne sans doute les formes virales bénéfiques. En effet, la propagation strictement verticale aligne les intérêts évolutifs des partenaires, c’est-à-dire que sans propagation vers d’autres hôtes, le nombre de descendants de ces virus ne peut être augmenté que par des mécanismes augmentant le nombre de descendants de l’hôte. La transmission verticale sélectionne donc chez le virus des traits bénéfiques à l’hôte comme, par exemple, la résistance à d’autres pathogènes ou aux stress.
 | Figure 1.
Transmission des virus pathogènes et persistants des plantes.
Les virus sont indiqués en rouge et les cellules végétales en noir, avec leurs communications caractéristiques (ou plasmodesmes). Les virus pathogènes
(A)
se transmettent de cellule en cellule horizontalement, localement par les plasmodesmes, et à distance, soit intra-hôte par la circulation dans les vaisseaux conducteurs d’une même plante, soit dans d’autres plantes par le biais de vecteurs. Les virus persistants
(B)
, qui ont une abondance cellulaire inférieure et n’engendrent pas de symptômes, se transmettent verticalement lors de la division cellulaire aux cellules filles. Ils sont donc strictement intracellulaires (certains intègrent le génome de l’hôte).
|
Certains virus persistants à ARN double-brin sont de fait bénéfiques. L’un d’eux assure une protection antivirale chez le poivron : la plante infectée par le virus pathogène de la mosaïque du concombre émet normalement des signaux volatils attirant les pucerons qui transmettent ce pathogène ; mais la présence d’un partitivirus
1
persistant chez le poivron limite cette attractivité et donc la propagation du virus pathogène [
2
]. L’interaction est parfois plus complexe : une graminée des sols volcaniques,
Dichanthelium lanuginosum
, survit à des températures supérieures à 45 °C uniquement si elle est colonisée par un champignon (
Curvularia protuberata
) porteur d’un virus à ARN double-brin ; sans l’union de ces trois partenaires, ni le champignon, ni la plante ne sont thermotolérants [
3
]. Récemment, des travaux expérimentaux ont comparé l’évolution de la résistance à l’absence d’eau sur la plante modèle
Arabidopsis thaliana
, infectée ou non par le Turnip mosaic potyvirus [
4
], un virus à génome ARN simple-brin de polarité positive non persistant. Les virus ayant évolué au sein de plantes stressées confèrent une résistance au stress hydrique, liée à une réorganisation de l’expression génétique de la plante, bien supérieure à celle apportée par les virus des plantes contrôles non stressées. Cela confirme que l’évolution de la plante peut s’appuyer sur celle de ses virus et recruter leurs propriétés pour conférer une adaptation, notamment au stress.
La présence de virus persistants a conduit parfois à leur intégration dans le génome végétal, selon un mécanisme sans doute accidentel mais qui s’est fréquemment produit dans l’évolution [
5
]. Cette situation poursuit la transmission verticale et donc l’émergence de traits bénéfiques. Ainsi, chez le trèfle blanc, un gène nucléaire impliqué dans la régulation de l’interaction de la plante avec les bactéries fixant l’azote,
TrEnodDR1
, dérive d’un gène de protéine de capsule du White clover cryptic virus 1, un partitivirus (virus à ARN double-brin segmenté) persistant de cet hôte [
6
].
Si les cas documentés et les processus de symbioses virales dans les plantes restent peu nombreux [
1
], ils montrent néanmoins l’existence de symbioses entre plantes et virus, et informent sur leurs rôles possibles dans l’adaptation aux contraintes biotiques et abiotiques. Ils suscitent également le développement de recherches qui permettront de déterminer s’ils sont le sommet d’un iceberg regroupant de très nombreuses interactions bénéfiques pour la physiologie végétale.
|
Des virus bénéfiques pour les animaux
Pour les virus infectant les animaux, les associations bénéfiques décrites entre virus et organismes sont principalement issues d’évènements d’intégration de séquences virales dans les génomes. Il existe une très grande diversité de virus ordonnés, selon la classification de Baltimore, suivant la nature et l’organisation du support de leur information génétique (ADN ou ARN, simple-brin ou double-brin, à polarité positive ou négative). Les virus intégrés correspondent le plus souvent à des rétrovirus (virus à ARN simple-brin à polarité positive) dont le cycle comprend une étape d’intégration dans l’ADN de l’hôte, ce qui est de nature à augmenter la probabilité de leur intégration dans les génomes. On retrouve cependant dans les génomes, des restes de tous les types de virus, même de virus à ARN qui ne produisent pourtant aucun intermédiaire ADN au cours de leur cycle de réplication [
7
]. Le plus souvent, ces séquences virales n’apportent pas de fonction à leur hôte et se dégradent progressivement jusqu’à disparaître. Dans de nombreux cas, ils produisent néanmoins des protéines virales modifiées, par exemple des protéines tronquées, qui continuent de se fixer à leur cible mais ont perdu leur fonction catalytique. Ces protéines interférent donc avec le cycle des virus apparentés et protègent ainsi l’hôte contre une nouvelle infection [
8
]. Les séquences virales intégrées peuvent également être la source de petits ARN régulateurs, qui activent la réponse de défense immunitaire [
9
]. Enfin, certaines intégrations virales ont apporté aux animaux des fonctions que les virus utilisent lors de leur cycle infectieux, comme des propriétés de fusion membranaire, ou la production de particules capables d’entrer efficacement dans des cellules et de transmettre ainsi l’information qu’elles contiennent [
10
].
|
Des gènes humains d’origine rétrovirale
Les rétrovirus endogènes, qui sont les témoins d’infections anciennes, constituent environ 8 % du génome humain. On les classe également comme « rétrotransposons à longue répétition terminale » ou à LTR (l’acronyme anglais de
long terminal repeat
), en raison de la similarité de leur structure avec ces éléments mobiles. La structure des rétrovirus suggère en effet fortement qu’ils dérivent des rétrotransposons à LTR [
11
]. La principale différence entre ces deux entités est l’acquisition par les rétrovirus d’un troisième cadre de lecture (
env
), qui code une protéine transmembranaire s’associant à l’enveloppe virale et qui est indispensable à la première étape de l’infection : l’entrée du virus dans les cellules de l’hôte. Par souci de simplification, nous parlerons dans les lignes qui suivent sans les distinguer de « rétrovirus/rétrotransposons ».
Les gènes Arc de la mémoire ont une origine rétrovirale
Biologiquement, la mémorisation repose sur la synthèse rapide et
de novo
d’ARN et de protéines dans les neurones, ainsi que sur les circuits formés par ces neurones en interactions. En effet, les souvenirs se forment grâce à la reconfiguration constante et le renforcement des réseaux neuronaux dans le cerveau, particulièrement au niveau des synapses. On désigne cette propriété des neurones sous le terme de « plasticité synaptique » [
12
] (
Figure 2A
).
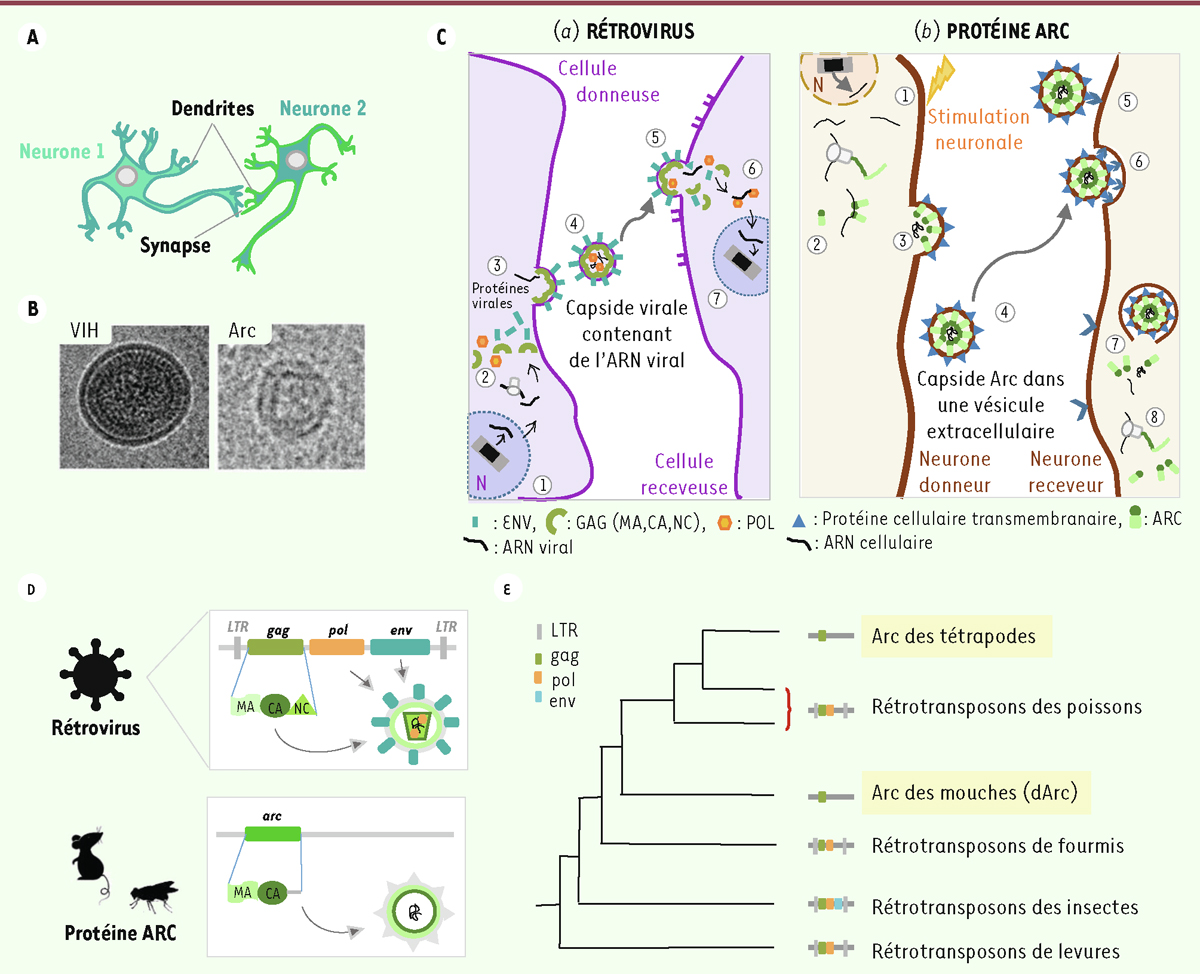
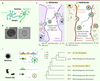 | Figure 2.
ARC, une protéine rétrovirale domestiquée. A.
Représentation schématique de deux neurones en contact.
B.
Images de microscopie électronique. VIH : particule rétrovirale du virus de l’immunodéficience humaine [
14
] ; ARC : capside ARC de rat [
46
] (avec l’autorisation d’Elsevier et de l’
American Society for Microbiology
).
C.
Comparaison du transfert d’ARN messager chez les rétrovirus (a) et
via
la pseudo-particule ARC (b). (a)
1.
Transcription de l’ADN viral.
2.
Traduction
3.
Assemblage de la capside et bourgeonnement. L’enveloppe de la particule provient de la cellule mais porte les protéines ENV.
4.
Transport de l’ARN viral dans la particule virale.
5.
Fusion de la particule avec la membrane cellulaire et désassemblage.
6.
Rétrotranscription.
7.
Intégration de l’ADN viral au génome cellulaire de la cellule receveuse
(b)
1.
Transcription cellulaire induite par l’activité neuronale.
2.
Traduction.
3.
Oligomérisation des protéines ARC pour former la capside contenant l’ARNm de
Arc
et des ARNm cellulaires.
4.
Relargage de la capside dans une vésicule extracellulaire au niveau de la membrane cellulaire.
5.
Reconnaissance des vésicules extracellulaires
via
des récepteurs membranaires.
6.
Endocytose.
7.
Désassemblage de la capside ARC.
8.
Traduction de l’ARNm de Arc en protéine.
D.
Comparaison de l’architecture génétique d’un rétrovirus et de la région génomique codant ARC.
E.
Phylogénie des gènes
Arc
des tétrapodes
, dArc
de la drosophile et
gag
des rétrovirus. N : noyau ; LTR :
long-terminal repeat
(séquences spécifiques partagées par les rétrovirus et les rétrotransposons apparentés qui sont nécessaires à leur mobilité).
|
De façon très simplifiée, un souvenir est le résultat du trajet entre neurones qu’emprunte un signal électrique. Le maintien de ces connexions synaptiques permet la mobilisation rapide de ce souvenir. La communication directe entre neurones est donc fondamentale dans les fonctions d’apprentissage et la mémoire. La fonction de mémoire, clef de notre identité et de notre interaction avec notre environnement, se trouve altérée dans de nombreuses maladies.
En 2018, une découverte surprenante a révélé que notre capacité à enregistrer, conserver et restituer des informations ou des événements impliquerait le détournement d’une fonction propre à ces rétrovirus/rétrotransposons. Les équipes de Vivan Budnik (université du Massachusetts) et de Jason Shepherd (université de l’Utah) ont étudié un gène neuronal impliqué dans les processus de mémorisation et d’apprentissage : le gène
Arc
(
activity-regulated cytoskeleton-associated protein
), qui s’exprime rapidement à la suite de l’activation des neurones. Les souris dont le gène
Arc
a été délété, sont dépourvues de mémoire à long terme. Chez l’homme, ce gène est associé à des troubles neurologiques comme la maladie d’Alzheimer [
13
].
In vitro,
les protéines ARC s’autoassemblent pour former une structure similaire à une capside [
14
,
15
], la structure protéique qui entoure habituellement le génome d’un virion (
Figure 2B
) et qui le protège. Les capsides transportent le matériel génétique du virus (ADN ou ARN) nécessaire à sa réplication, d’une cellule infectée vers une cellule non infectée (
Figure 2C
). Les « capsides » formées par les protéines ARC incorporent également leur propre ARN messager (ARNm) et circulent d’un neurone donneur vers un neurone receveur en utilisant des vésicules extracellulaires (
Figure 2C
). Après fusion de cette capside avec la membrane du neurone receveur, l’ARNm de
Arc
s’y déverse, entraînant la production de protéines ARC et leur accumulation dans le neurone receveur. L’ARNm de
Arc
circule ainsi de neurone en neurone [
16
,
17
].
Les études phylogénétiques ont montré que la séquence de la protéine ARC présente de fortes similitudes avec la protéine GAG, le produit d’un gène des rétrovirus/rétrotransposons bien connu qui permet la formation des capsides virales. GAG est une poly-protéine structurale (
Figure 2D
) qui est clivée en différentes protéines : la protéine de capside (CA), qui s’oligomérise en capside ; la protéine de matrice (MA), qui participe à l’encapsulation de l’ARN et cible les membranes ; et la protéine de nucléocapside (NC), qui interagit également avec l’ARN. ARC présente de fortes similarités de séquences avec GAG, mais aussi des identités de structure avec les domaines CA et MA de GAG, ainsi qu’avec le domaine MA de la protéine GAG du VIH (virus d’immunodéficience humaine). Seul le domaine NC n’est pas retrouvé dans la protéine ARC. Ces similarités suggèrent ainsi une appropriation du gène
gag
des rétrovirus par les organismes, pour le transport de matériel génétique entre neurones.
Les analyses phylogénétiques ont révélé une origine du gène
Arc
qui remonte à l’ancêtre des tétrapodes (amphibiens, reptiles, oiseaux et mammifères) qui vivait il y a environ 350 millions d’années [
14
]. Les études d’homologie ont quant à elles montré l’existence de caractéristiques communes entre le gène
Arc
et un rétrotransposon présent chez les poissons, appartenant au groupe «
gypsy
», à l’origine de la protéine ARC [
14
].
Gypsy
a d’abord été classé comme un rétrotransposon, puis comme un rétrovirus d’insecte, lorsque sa capacité infectieuse conférée par un gène
env
a été démontrée [
18
].
Plus surprenant encore, au sein des protéines homologues d’ARC, le domaine CA s’avère conservé entre les drosophiles (on parle alors de la protéine dARC) et les mammifères. Mais, à la différence des gènes des tétrapodes, les gènes
dArc
trouvés chez les insectes auraient une origine plus proche de celle d’un «
gypsy
» présent chez les insectes [
14
]. Cette observation indique que le recrutement de la protéine ARC a eu lieu plusieurs fois au cours de l’évolution animale, à partir de deux lignées différentes de
gypsy
, par une convergence évolutive entre vertébrés et insectes.
Cette histoire évolutive du gène
Arc
est d’autant plus originale qu’il s’agit de la première description d’une communication entre neurones reposant sur des vésicules extracellulaires, et non sur des signaux électriques ou chimiques. Il existe bien d’autres exemples d’appropriation d’un gène viral qui a été détourné par les organismes afin d’en tirer un avantage. Nous en décrirons deux, d’abord celui du troisième gène rétroviral
env,
qui est à l’origine de la fusion de cellules nécessaire au développement du placenta des mammifères, et, chez les insectes, l’exemple des pucerons qui utilisent une séquence virale présente dans leur génome pour produire des individus ailés.
Les syncytines impliquées dans la formation du placenta ont une origine rétrovirale
Sans les virus, nous ne serions pas nés…
Dans une série d’articles parus dans les années 2010, l’équipe de Thierry Heidmann (Institut Gustave Roussy, Villejuif) a montré que des copies d’un gène rétroviral, le gène
env
, intégrées dans le génome des mammifères placentaires, participaient à des fonctions physiologiques essentielles du placenta [
19
,
20
]. Au stade initial de l’infection par ces virus, la protéine ENV interagit avec un récepteur spécifique qui dépend du virus et induit la fusion de l’enveloppe virale avec la membrane plasmique de la cellule cible, permettant ainsi l’entrée du virus. Synthétisée par les cellules infectées et exposée à leur surface, la protéine ENV peut également être à l’origine de leur fusion avec les cellules voisines, sous réserve qu’elles expriment le récepteur approprié, ce qui induit la formation de syncytiums, des cellules géantes comportant de nombreux noyaux. Cette fusion entre cellules peut être obtenue
in vitro
, en culture cellulaire [
21
]. De tels syncytiums sont également produits naturellement chez l’homme, au cours du développement du syncytiotrophoblaste, la partie du placenta qui permet les échanges entre le sang du fœtus et celui de la mère, et qui forme une barrière continue évitant tout mélange entre les sangs. Selon les lignées de mammifères, l’organisation des placentas diffère, mais tous comportent des syncytiums. Plusieurs gènes, codant des protéines ENV et exprimés spécifiquement dans le placenta, ont été identifiés dans différentes espèces. Leurs produits ont été nommés syncytines en fonction de leur propriété d’induire des fusions de cellules en syncytium.
In vivo
, l’élimination des deux gènes codant la syncytine, dans des souris génétiquement modifiées (par «
knock out »
), a révélé l’implication de ces gènes dans le développement du placenta et dans la survie des embryons [
22
]. Étonnamment, les gènes codant les syncytines proviennent de familles de rétrovirus endogènes qui sont différentes selon les lignées de mammifères placentaires. Ils correspondent donc à des évènements de captures de gènes de virus qui sont indépendants [
23
]. L’hypothèse évolutive proposée pour expliquer cette multiplicité des évènements de capture est complexe. À l’origine de la lignée des mammifères placentaires, une syncytine, aujourd’hui disparue, aurait permis initialement les échanges fœtaux-maternels. Puis, de nouvelles acquisitions de gènes
env
, selon les lignées (à partir d’autres rétrovirus), auraient ensuite fourni des syncytines différentes, conférant des propriétés de fusion spécifiques pour chaque lignée. Ces syncytines auraient remplacé la syncytine ancestrale et auraient permis l’apparition des différents types de placentas actuels. Des captures du gène
env
se sont aussi produites en dehors des mammifères : des lézards vivipares pourvus d’une sorte de placenta disposent également d’une syncytine d’origine rétrovirale [
24
] !
|
Pour le puceron, avoir ou non des ailes dépend des gènes d’un densovirus
Les rétrovirus ne sont pas les seuls virus à petit génome qui ont apporté des fonctions aux organismes : c’est aussi le cas des densovirus, des virus dépourvus d’enveloppe, apparentés aux parvovirus des vertébrés, et responsables d’épizooties touchant les insectes et les crustacés [
25
]. Leur génome est constitué d’un ADN simple-brin linéaire.
Récemment, l’équipe de Jeniffer Brisson (université de Rochester) a montré que chez le puceron cendré du pommier (
Dysaphis plantaginea
), des gènes de densovirus, intégrés dans le génome de l’insecte, contribuaient à la plasticité de réponse à une forte densité populationnelle [
26
]. La reproduction des pucerons s’effectue le plus souvent de manière clonale, sous la forme d’individus dépourvus d’ailes. L’apparition d’individus ailés, capables de se disperser, se fait à la suite d’épisodes de surpopulation sur la plante, mais à une fréquence très variable selon les colonies. Deux gènes, intégrés dans le génome du puceron cendré du pommier, proviennent d’un densovirus. Ils codent des protéines apparentées à NS1, une protéine non structurale agissant, en particulier, comme facteur activateur de la transcription durant l’infection par un parvovirus [
27
]. Parmi des lignées de pucerons sélectionnées pour leur réponse plus ou moins forte à une densité de population élevée, l’expression de ces deux gènes est induite à un niveau plus élevé dans la lignée présentant une forte réponse à la densité de population. Une analyse fonctionnelle, par injection d’ARN viral double-brin dans les pucerons, a montré que la diminution de l’expression des gènes
ns1
du puceron, induite par interférence ARN, réduisait fortement la production des individus ailés [
26
], montrant ainsi l’implication effective de ces gènes dans la plasticité de la réponse aux conditions environnementales. Les infections des pucerons par des densovirus peuvent également causer l’apparition d’individus ailés même en dehors des conditions habituelles, ce qui contribue à disperser le virus. L’intégration génomique du gène du densovirus a permis aux pucerons de tirer parti de cet effet de l’infection, sur la production des ailes, afin de réguler la plasticité du phénotype ailé.
|
Des dizaines de milliers d’espèces de guêpes utilisent un polydnavirus pour réussir leur parasitisme
Les exemples les plus spectaculaires d’utilisation de virus par des animaux concernent des guêpes parasites et les polydnavirus (PDV) pour lesquels un mécanisme de symbiogenèse [
28
] a permis l’appropriation par l’animal non pas d’un seul gène, comme dans les exemples décrits précédemment, mais de toute une machinerie virale utilisée comme vecteur de transfert de gènes [
10
]. Cela correspond, en quelque sorte, à une « domestication » du virus par la guêpe. Les guêpes parasites utilisent leur ovipositeur
2
pour pondre leurs œufs directement dans le corps d’un autre insecte, l’hôte parasité : le plus souvent une chenille. Après l’éclosion des œufs, les larves de guêpes parasites se développent à l’intérieur de la chenille hôte. Ces guêpes sont omniprésentes dans les écosystèmes, où elles jouent un rôle important dans le contrôle des populations d’insectes. Plusieurs dizaines de milliers d’espèces sont porteuses de polydnavirus qui appartiennent à plusieurs groupes. Deux genres associés à certaines familles de guêpes ont été définis au sein des polydnavirus : les ichnovirus, portés par les ichneumonides, et les bracovirus, portés par les braconides [
29
].
Le corps de l’hôte, la chenille, fournit l’alimentation aux larves, mais la réponse immunitaire que l’hôte développe pourrait tout aussi bien tuer ces larves ou détruire les œufs, juste après la ponte. Contre un corps étranger, tel que l’est un œuf de parasite, les chenilles se défendent en formant autour de celui-ci une gangue de cellules immunitaires [
30
]. Plusieurs groupes de parasites ont une stratégie utilisant le polydnavirus pour inhiber ce mécanisme de défense, une sorte de thérapie génique néfaste consistant à introduire des gènes qui s’intègrent dans l’ADN de l’hôte. Ces gènes sont exprimés par les cellules de l’hôte infecté. Leurs produits inhibent l’immunité de l’hôte, mais ils manipulent également son programme de développement (la chenille infectée croît à travers plusieurs stades larvaires, mais elle ne se métamorphose jamais en papillon), ainsi que de nombreux autres aspects de sa physiologie, mais toujours au bénéfice des larves de guêpes (
Figure 3
).
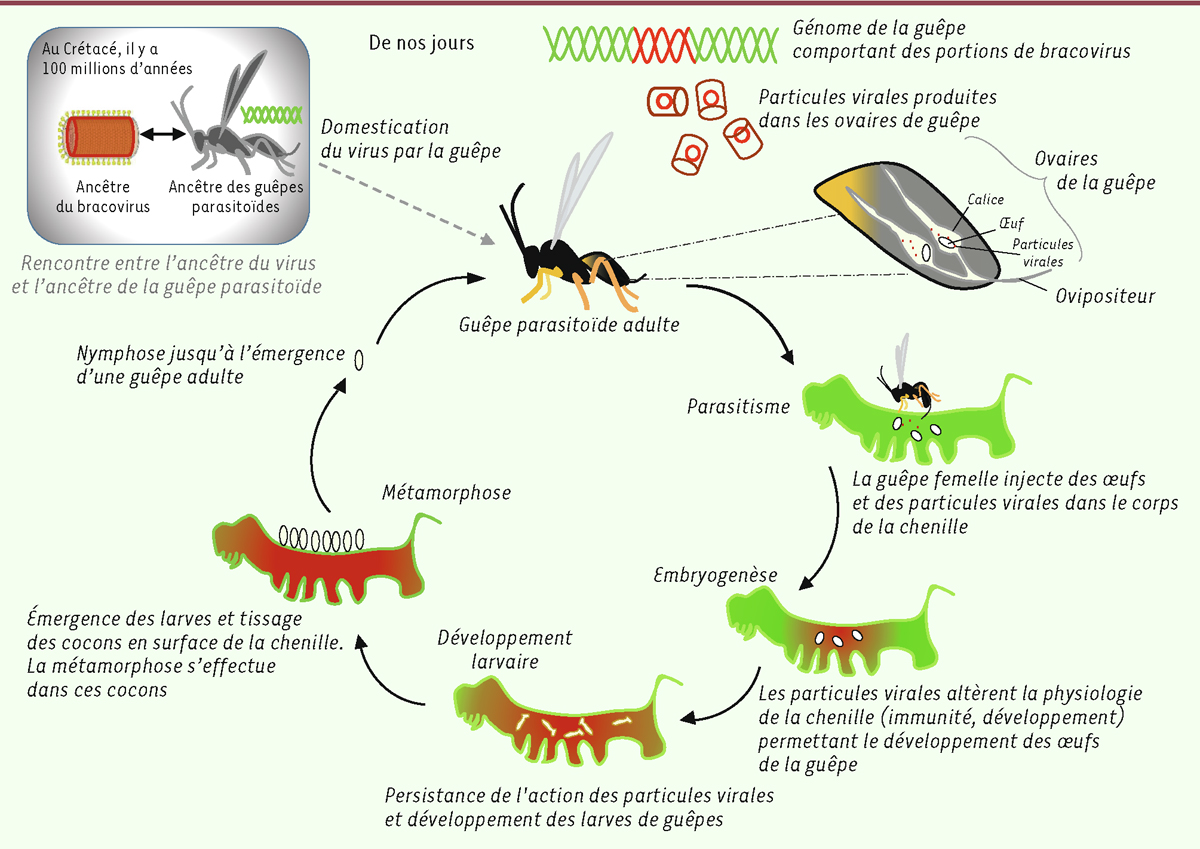
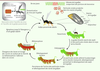 | Figure 3.
Cycle de vie des hyménoptères parasites associés au bracovirus.
Les guêpes parasites (dites parasitoïdes quand le parasitisme entraîne la mort de l’hôte), qui possèdent aujourd’hui des fragments domestiqués de bracovirus au sein de leur génome, se servent des particules produites dans leurs ovaires pour permettre aux œufs qu’elles injectent dans le corps de la chenille d’éclore et aux larves de se développer dans corps de l’hôte. L’expression des gènes viraux altère la réponse immunitaire, modifie le métabolisme et affecte le développement de la chenille qui ne devient jamais un papillon.
|
|
Les particules de bracovirus sont produites dans les ovaires des guêpes parasites à partir de gènes nudiviraux
L’étude des bracovirus (BV) a permis de démontrer que les polydnavirus avaient bien une origine virale, ce qui n’était jusque-là que suggéré par l’aspect des particules observées en microscopie électronique. Ces particules virales s’assemblent (
Figure 4
) dans le noyau des cellules du calice, une région de l’ovaire de la guêpe. La lyse ultérieure des cellules productrices les libère ensuite dans l’oviducte, où elles s’accumulent, prêtes à être co-injectées lors de la ponte de l’œuf de la guêpe. La production est massive, ce qui implique une très forte expression des gènes viraux dans les ovaires.
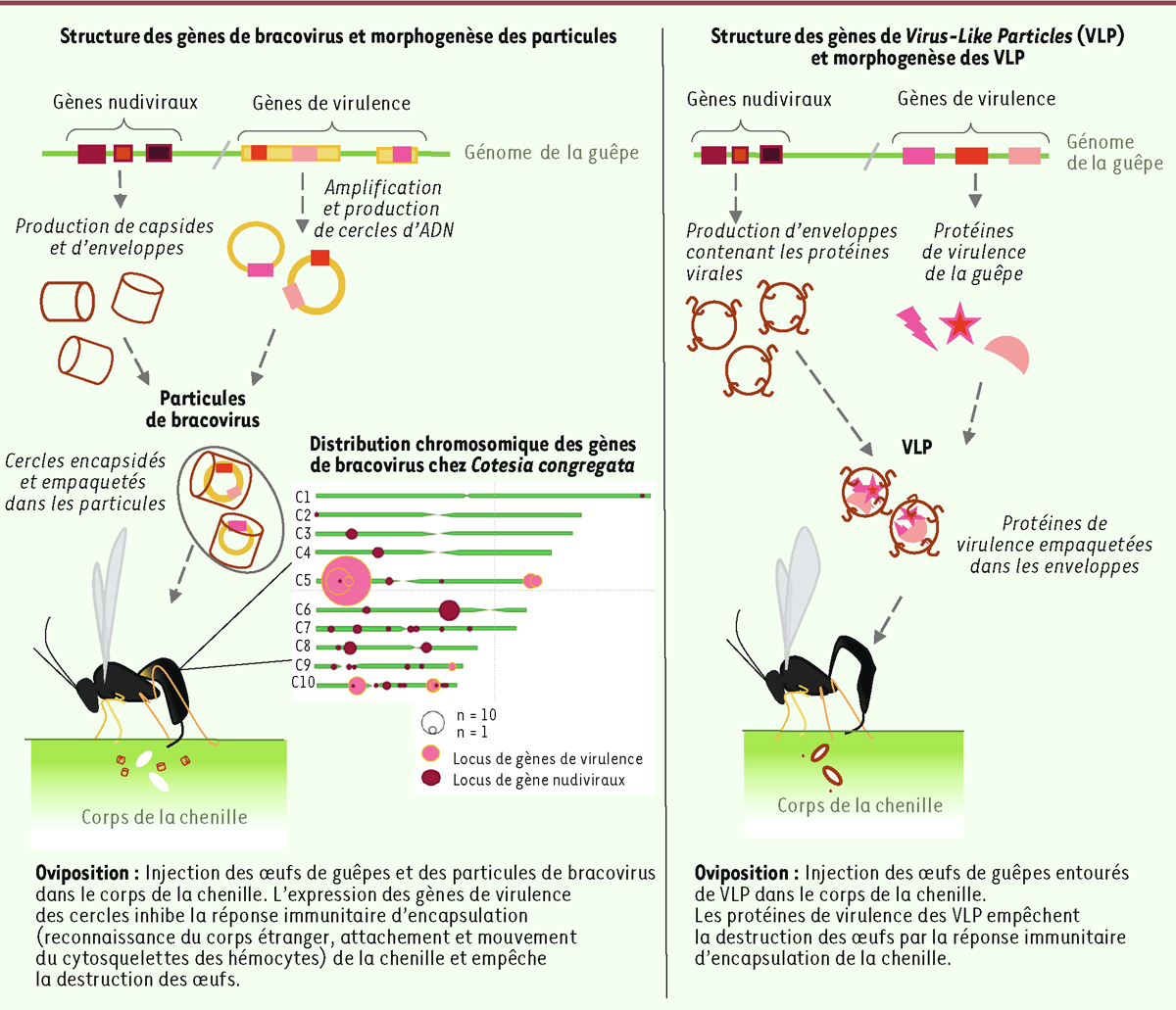
 | Figure 4.
Comparaison de la structure génétique et de la morphogenèse des bracovirus et des VLP (
virus-like particles
).
Les séquences du bracovirus dispersées dans le génome de la guêpe sont constituées de gènes nudiviraux impliqués dans la production des capsides et des enveloppes constituant les particules, ainsi que de gènes de virulence incorporés dans les nucléocapsides. Ces derniers, introduits lors de l’oviposition de la guêpe, sont exprimés par les cellules infectées de la chenille et altèrent sa physiologie afin d’éviter la destruction des œufs de guêpe. Certains des gènes de virulence ont une origine « guêpe » (ils ressemblent à des gènes connus d’hyménoptères) mais la plupart sont d’origine inconnue. Au contraire des bracovirus qui contiennent de l’ADN, les VLP n’ont pas de capsides ; elles sont constituées d’enveloppes virales qui contiennent des protéines de virulence. Ces protéines ont une origine « guêpe ».
|
Nous avons mené une étude dans notre laboratoire (IRBI, CNRS/université de Tours) en collaboration avec le laboratoire de Béatrice Lanzrein (université de Berne) et le Génoscope (CEA, Évry), sur deux espèces de guêpes éloignées phylogénétiquement,
Cotesia congregata
et
Chelonus inanitus
. Une approche de séquençage des ARN messagers exprimés dans les ovaires des insectes, a montré que de nombreux gènes viraux, caractéristiques des nudivirus, étaient exprimés dans les deux espèces [
31
]. Une analyse de protéomique a ensuite prouvé que les produits de ces gènes étaient les composants structuraux des particules de bracovirus. Ces résultats montraient que les particules de bracovirus étaient produites par des gènes de nature nudivirale, et les analyses de l’ADN de l’espèce
Cotesia
indiquaient par ailleurs que ces gènes étaient intégrés dans le génome de la guêpe (
Figure 5
) [
31
]. Les nudivirus infectent des insectes et des crustacés. Ce sont des virus à ADN double-brin circulaire de grande taille, qui constituent un groupe frère des baculovirus, mieux connus, qui sont utilisés en lutte biologique contre les lépidoptères ravageurs [
32
].
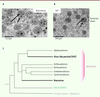 | Figure 5.
Origine nudivirale des bracovirus et des VLP (
virus-like particles
). A.
Observations en microscopie électronique de particules de bracovirus en formation dans le noyau de cellule de calice chez
Cotesia congregata
.
B.
Observation en microscopie électronique à transmission de particules de type VLP dans le noyau de cellule de calice chez
Venturia Canescens
. (Crédits photos, M. Leobold).
C.
Arbre phylogénétique des nudivirus (d’après [
32
]). Les branches correspondant aux virus endogènes produisant les particules de bracovirus et les VLP de l’espèce
Venturia canescens
sont représentées en gras. Les virus originellement capturés et domestiqués appartiennent à deux clades différents de nudivirus.
|
Les études phylogénétiques avaient montré que les guêpes associées aux bracovirus constituaient un groupe monophylétique, dérivant d’un ancêtre commun [
33
]. L’ensemble de ces données a permis de formuler le scénario évolutif selon lequel un nudivirus s’est intégré dans le génome de cet ancêtre commun, une guêpe parasite braconide vivant au Crétacé, il y a environ 100 millions d’années [
31
]. Les multiples évènements de spéciation qui se sont produits dans la descendance de cette guêpe on conduit à la grande diversité des espèces de guêpes actuelles (leur nombre est estimé à 50 000), qui comportent, chacune, leur propre bracovirus, fruit de l’évolution particulière du nudivirus endogène dans chaque lignée.
Les séquences virales ont colonisé tous les chromosomes des guêpes
Les études portant sur l’évolution des génomes de bracovirus et la conservation de leurs fonctions virales ont reposé principalement sur les espèces de guêpes
Cotesia congregata
et
Microplitis demolitor
, cette dernière étant étudiée dans le laboratoire de Mike Strand (Université de Géorgie). Ces espèces appartiennent à deux lignées de la même sous-famille de guêpes braconides, séparées depuis 50 millions d’années [
33
].
Les génomes de nudivirus pathogènes sont constitués d’un ADN circulaire de 100 à 200 kilobases, codant entre 90 et 150 protéines ; le nudivirus originellement capturé se situait probablement dans ces limites [
32
]. Plus d’une trentaine de ces protéines, codées par les gènes « cœurs » du génome nudiviral, sont essentielles au cycle viral et sont conservées chez tous les nudivirus pathogènes.
Le génome de
Cotesia congregata
a été obtenu récemment et a été assemblé jusqu’à l’échelle des chromosomes. Son analyse montre que les gènes « cœurs » du nudivirus ancestral ont été conservés (
Figure 4
: gènes nudiviraux), à l’exception du gène codant l’ADN polymérase, mais qu’ils sont aujourd’hui dispersés sur tous les chromosomes de l’insecte. Initialement, ils ont très probablement été acquis par l’intégration de l’ensemble du génome nudiviral ancestral en un seul point du génome de la guêpe. Ils se sont ensuite dispersés du fait des remaniements qui se produisent au fil du temps dans les génomes, modifiant l’ordre des gènes [
34
]. Bien que dispersés, ces gènes sont toujours exprimés de manière coordonnée et les protéines qu’ils produisent interagissent pour fabriquer les particules du bracovirus. Il s’agit en particulier des gènes codant les différentes sous-unités de l’ARN polymérase nudivirale, qui assure l’expression d’une série d’autres gènes nudiviraux, comme cela se produit au cours d’une infection par un nudivirus pathogène [
35
,
47
]. Vingt-cinq gènes nudiviraux restent localisés dans une même région de 100 kilobases du chromosome 7
,
nommée « cluster nudiviral », extrêmement bien conservée entre les deux génomes de guêpes séquencés (
Figure 4
) [
34
,
36
]. Cette région correspondrait à un fragment très peu remanié du virus originellement intégré. Son maintien dans un contexte général de dispersion génique suggère une forte contrainte évolutive, probablement liée au fait que le cluster nudiviral est amplifié (mais non incorporé dans les capsides) et code les protéines majeures des capsides qui doivent être produites en grande quantité pour fabriquer les particules qui sont injectées massivement dans les chenilles parasitées.
Le second type d’unités fonctionnelles du génome du bracovirus correspond à l’ensemble des régions génomiques de la guêpe contenant les gènes de virulence qui servent de matrice pour produire les cercles d’ADN incorporés dans les particules. Ces régions, les locus proviraux, sont situées dans des positions similaires dans les deux espèces de guêpe ; leur organisation est restée remarquablement stable dans le génome des guêpes sur plus de 50 millions d’années [
34
]. En revanche, le contenu en gènes de virulence varie. Certains proviennent du fond génétique des hyménoptères mais, étonnamment, aucun ne montre une origine nudivirale [
37
]. Les gènes viraux d’origine ont donc été perdus par le génome viral encapsidé. Cela s’explique probablement par le fait qu’il n’y a pas de production de particules dans les chenilles parasitées, donc pas de pression de sélection agissant pour le maintien de ces gènes dans l’ADN introduit dans les chenilles. Ainsi, sur le très long terme, seule leur présence dans le génome de la guêpe est maintenue. Le mécanisme de réplication de l’ADN viral est complexe et incomplètement élucidé, en particulier l’ADN polymérase qui s’est substituée à celle du nudivirus n’a pas été identifiée [
38
,
39
]. Des séquences régulatrices bornant les régions amplifiées sont probablement d’origine nudivirale [
34
], suggérant une réalisation conjointe de la réplication de l’ADN viral par la guêpe et par le virus, qui procure probablement à la guêpe l’assurance contre l’émergence d’un virus pathogène à partir des gènes viraux qui sont présents dans son génome.
|
Les ichnovirus sont produits par un virus endogène d’une famille inconnue sous forme libre
Plusieurs milliers d’espèces appartenant à deux sous-familles des Ichneumonidae (Campopleginés, Banchinés) produisent également des particules dont la morphologie diffère de celle des bracovirus. Les études de l’évolution de ces autres polydnavirus, nommés ichnovirus (IV), ont été conduites en parallèle à celle des bracovirus, mais leur nature virale s’est révélée plus difficile à établir. L’approche initiale d’analyse du transcriptome des ovaires de guêpes par l’équipe de Nathalie Volkoff (INRAE /université de Montpellier) impliquait l’espèce de guêpe
Hyposoter didymator
, qui produit un ichnovirus. Mais aucun ensemble d’ARNm typiques d’un virus n’avait été identifié dans les ovaires d’
Hyposoter
[
31
]. En revanche, nous avions remarqué que les gènes surexprimés spécifiquement dans le tissu produisant les particules ne comportaient pas d’introns (comme les gènes nudiviraux des bracovirus) et étaient densément concentrés dans des régions particulières du génome. L’analyse protéomique des particules d’ichnovirus a ensuite révélé que ces gènes codaient les composants structurels de ces particules [
40
]. L’organisation et la spécialisation fonctionnelle de ces régions rappellent celles du cluster nudiviral des bracovirus, suggérant qu’elles correspondent également à des séquences virales intégrées dans le génome des guêpes. Les ichnovirus seraient donc également issus de l’intégration d’un virus ancestral. L’absence de similarités des gènes d’ichnovirus avec des gènes viraux répertoriés dans les banques de données pourrait être expliquée par notre connaissance encore non exhaustive de la diversité des virus d’insectes. Il est aussi possible que la famille des ancêtres des ichnovirus soit éteinte et que seules subsistent les séquences intégrées dans le génome des guêpes. Le séquençage du génome d’
H. didymator
et d’une autre guêpe campoplégine
3
,
Campoletis sonorensis
, publié en 2020, a révélé que le contenu et l’ordre des gènes produisant les particules sont conservés entre espèces, tandis que les locus proviraux contenant des gènes de virulence sont très dispersés et dans des positions non conservées entre espèces proches [
41
]. Au contraire des séquences provirales des bracovirus, celles des ichnovirus bougent donc assez fréquemment dans le génome (un peu comme des éléments transposables), ce qui reflète peut-être des propriétés différentes des virus ancestraux.
|
Certains nudivirus endogènes produisent des particules sans ADN qui véhiculent des protéines
Les études des nudivirus endogènes des guêpes
Venturia canescens
et
Fopius arisanus
révèlent une autre manipulation de la physiologie de l’hôte parasité [
42
,
43
]. Dans celle-ci, les VLP (
virus-like particles
) (
Figure 4
) [
44
] ne contiennent pas d’acides nucléiques, mais des protéines de virulence qui sont produites par la guêpe. Ces associations avec les nudivirus sont beaucoup plus récentes (de l’ordre de quelques millions d’années) que celles impliquant les bracovirus (qui datent de 100 millions d’années) ; elles permettent de mieux comprendre les phases plus précoces de la domestication d’un nudivirus (
Figure 5
). Le génome de
Venturia canescens
montre que les gènes qui produisent l’ARN polymérase et les protéines d’enveloppe nudivirales ont été conservés. Par contre, les gènes codant la capside et l’ADN polymérase sont des pseudogènes dont le cadre de lecture initial est très altéré [
45
]. L’identification de ces pseudogènes, chez
Venturia
indique que les remaniements du génome viral après son intégration génomique, ne se font pas principalement par la perte de grands blocs de gènes, comme pour les endosymbiotes bactériens
4,
, associés à de nombreuses espèces d’insectes, mais gène par gène, par l’accumulation de mutations interrompant le cadre de lecture, lorsqu’un gène ne contribue plus à la nouvelle fonction des particules. Ces génomes montrent également que les séquences virales sont beaucoup moins dispersées chez
Venturia
et
Fopius
que celles des bracovirus, ce qui confirme l’hypothèse selon laquelle la dispersion des gènes viraux dans le génome de l’hôte s’effectue par un processus de brassage progressif opérant dans tous les génomes [
42
,
43
].
|
La connaissance des microbiotes animaux et végétaux est en plein essor, en grande partie parce qu’il est désormais possible d’inventorier les espèces présentes par des analyses moléculaires. Les code-barres moléculaires (
barcodes
), obtenus par séquençage à haut-débit, y sont pour beaucoup, pour identifier les protagonistes et pour suivre l’évolution des microbiotes. S’il existe de nombreux virus au sein des microbiotes, leur étude reste plus difficile. Les méthodes de code-barre moléculaire reposent sur des locus conservés entre espèces, comme l’ADN ribosomal, amplifiables à l’aide d’amorces spécifiques d’un groupe donné. C’est ce qu’empêche l’extraordinaire diversité des génomes viraux, qui n’ont aucun gène en commun et qui, pour les virus à ARN, nécessitent une rétro-transcription préalable en ADN. S’il est possible, par un séquençage massif de l’ensemble du matériel génétique, de déterminer la diversité virale d’un échantillon, la chose reste peu routinière et exige de gros efforts d’analyses bioinformatiques. Faute de moyen simple pour la décrire, notre connaissance de la diversité virale et de ses rôles reste limitée et est dans l’état où en était la connaissance des microbiotes bactériens et fongiques dans les années 1970-1980. Mais ce retard se comble, notamment grâce à des approches alternatives permettant de mieux accéder à la diversité virale au sein des microbiotes, comme la transcriptomique ciblée sur les ARN viraux et la métagénomique ciblée sur les virus, comportant une étape de purification partielle des particules virales avant l’analyse.
D’ores et déjà, les quelques exemples que nous avons mentionnés suggèrent que la symbiose virale est bien une réalité. Les captures cytosoliques ou génomiques de virus se sont produites de manière récurrente au cours de l’évolution. L’identification plus globale de la contribution virale à l’évolution des plantes et des animaux nécessitera cependant une meilleure connaissance de la biodiversité virale, comme le montre l’exemple des ichnovirus, dont la batterie de gènes caractéristiques est inconnue chez les autres familles virales décrites. Les études récentes de métagénomes viraux ont permis d’identifier des séquences de virus correspondant à des familles complètement nouvelles. Elles indiquent le chemin restant à parcourir pour caractériser la diversité du monde viral. Gageons que la masse et la qualité croissante des données de séquençage produites aujourd’hui combleront ces lacunes. Dans les prochaines années, de très nombreux génomes de plantes et d’animaux seront accessibles grâce à des grands projets, comme l’Atlas de Génomes de Références Européen (ERGA). Nous espérons que les exemples que nous avons cités inciteront les chercheurs travaillant sur les génomes d’organismes variés à identifier de nouveaux exemples de capture de séquences virales pour évaluer, de manière plus systématique, l’apport des virus dans l’évolution des plantes et des animaux. |
Les auteurs déclarent n’avoir aucun lien d’intérêt concernant les données publiées dans cet article.
|
Footnotes |
1.
Roossinck
MJ
.
Evolutionary and ecological links between plant and fungal viruses.
.
New Phytol.
2019;
;
221
:
:86.
–
92
.
2.
Safari
M
,
Ferrari
MJ
,
Roossinck
MJ
.
Manipulation of Aphid Behavior by a Persistent Plant Virus.
.
J Virol.
2019;
;
93
:
:e01781.
–
e01718
.
3.
Marquez
LM
,
Redman
RS
,
Rodriguez
RJ
,
Roossinck
MJ
.
A virus in a fungus in a plant: three-way symbiosis required for thermal tolerance.
.
Science.
2007;
;
315
:
:513.
–
515
.
4.
Gonzalez
R
,
Butkovic
A
,
Escaray
FJ
,
et al.
Plant virus evolution under strong drought conditions results in a transition from parasitism to mutualism.
.
Proc Natl Acad Sci U S A.
2021;
;
118
:
:e2020990118.
.
5.
Chiba
S
,
Kondo
H
,
Tani
A
,
et al.
Widespread endogenization of genome sequences of non-retroviral RNA viruses into plant genomes.
.
PLoS Pathog.
2011;
;
7
:
:e1002146.
.
6.
Nakatsukasa-Akune
M
,
Yamashita
K
,
Shimoda
Y
,
et al.
Suppression of root nodule formation by artificial expression of the TrEnodDR1 (coat protein of White clover cryptic virus 1) gene in Lotus japonicus.
.
Mol Plant Microbe Interact.
2005;
;
18
:
:1069.
–
1080
.
7.
Katzourakis
A
,
Gifford
RJ
.
Endogenous viral elements in animal genomes.
.
PLoS Genet.
2010;
;
6
:
:e1001191.
.
8.
Frank
JA
,
Feschotte
C
.
Co-option of endogenous viral sequences for host cell function.
.
Curr Opin Virol.
2017;
;
25
:
:81.
–
89
.
9.
Suzuki
Y
,
Baidaliuk
A
,
Miesen
P
,
et al.
Non-retroviral Endogenous Viral Element Limits Cognate Virus Replication in Aedes aegypti Ovaries.
.
Curr Biol.
2020;
;
30
:
:3495.
–
506 e6
.
10.
Drezen
JM
,
Leobold
M
,
Bézier
A
,
et al.
Endogenous viruses of parasitic wasps: variations on a common theme.
.
Curr Opin Virol.
2017;
;
25
:
:41.
–
48
.
11.
Malik
HS
,
Henikoff
S
,
Eickbush
TH
.
Poised for contagion: evolutionary origins of the infectious abilities of invertebrate retroviruses.
.
Genome Res.
2000;
;
10
:
:1307.
–
1318
.
12.
Anggono
V
,
Huganir
RL
.
Regulation of AMPA receptor trafficking and synaptic plasticity.
.
Curr Opin Neurobiol.
2012;
;
22
:
:461.
–
469
.
13.
Bi
R
,
Kong
LL
,
Xu
M
,
et al.
The Arc Gene Confers Genetic Susceptibility to Alzheimer’s Disease in Han Chinese.
.
Mol Neurobiol.
2018;
;
55
:
:1217.
–
1226
.
14.
Pastuzyn
ED
,
Day
CE
,
Kearns
RB
,
et al.
The Neuronal Gene Arc Encodes a Repurposed Retrotransposon Gag Protein that Mediates Intercellular RNA Transfer.
.
Cell.
2018;
;
172
:
:275.
–
88 e18
.
15.
Ashley
J
,
Cordy
B
,
Lucia
D
,
et al.
Retrovirus-like Gag protein Arc1 binds RNA and traffics across synaptic boutons.
.
Cell.
2018;
;
172
:
:262.
–
74 e11
.
16.
Hantak
MP
,
Einstein
J
,
Kearns
RB
,
Shepherd
JD
.
Intercellular Communication in the Nervous System Goes Viral.
.
Trends Neurosci.
2021;
;
44
:
:248.
–
59
.
17.
Albagli
O
,
Pelczar
H
.
La protéine neuronale Arc : une capside de rétrotransposon recyclée pour des fonctions clés dans les synapses.
.
Med Sci (Paris).
2020;
;
36
:
:980.
–
3
.
18.
Touret
F
,
Guiguen
F
,
Terzian
C
.
Un retrovirus endogene actif et infectieux : le cas gypsy chez Drosophila melanogaster.
.
Virologie (Montrouge).
2012;
;
16
:
:381.
–
389
.
19.
Lavialle
C
,
Cornelis
G
,
Dupressoir
A
,
et al.
Paleovirology of ‘syncytins’, retroviral env genes exapted for a role in placentation.
.
Philos Trans R Soc Lond B Biol Sci.
2013;
;
368
:
:20120507.
.
20.
Dupressoir
A
,
Heidmann
T
.
Les syncytines des protéines d’enveloppe rétrovirales capturées au profit du développement placentaire.
.
Med Sci (Paris).
2011;
;
27
:
:163.
–
169
.
21.
Blond
JL
,
Lavillette
D
,
Cheynet
V
,
et al.
An envelope glycoprotein of the human endogenous retrovirus HERV-W is expressed in the human placenta and fuses cells expressing the type D mammalian retrovirus receptor.
.
J Virol.
2000;
;
74
:
:3321.
–
3329
.
22.
Dupressoir
A
,
Vernochet
C
,
Bawa
O
,
et al.
Syncytin-A knockout mice demonstrate the critical role in placentation of a fusogenic, endogenous retrovirus-derived, envelope gene.
.
Proc Natl Acad Sci U S A.
2009;
;
106
:
:12127.
–
12132
.
23.
Cornelis
G
,
Heidmann
O
,
Degrelle
SA
,
et al.
Captured retroviral envelope syncytin gene associated with the unique placental structure of higher ruminants.
.
Proc Natl Acad Sci U S A.
2013;
;
110
:
:E828.
–
E837
.
24.
Cornelis
G
,
Funk
M
,
Vernochet
C
,
et al.
An endogenous retroviral envelope syncytin and its cognate receptor identified in the viviparous placental Mabuya lizard.
.
Proc Natl Acad Sci U S A.
2017;
;
114
:
:E10991.
–
EE1000
.
25.
Gosselin Grenet
AS
,
Salasc
F
,
Francois
S
,
et al.
Les densovirus : une « massive attaque » chez les arthropodes.
.
Virologie (Montrouge).
2015;
;
19
:
:19.
–
31
.
26.
Parker
BJ
,
Brisson
JA
.
A Laterally Transferred Viral Gene Modifies Aphid Wing Plasticity.
.
Curr Biol.
2019;
;
29
:
:2098.
–
103 e5
.
27.
Xu
P
,
Zhou
Z
,
Xiong
M
,
et al.
Parvovirus B19 NS1 protein induces cell cycle arrest at G2-phase by activating the ATR-CDC25C-CDK1 pathway.
.
PLoS Pathog.
2017;
;
13
:
:e1006266.
.
28.
Federici
BA
,
Bigot
Y
.
Origin and evolution of polydnaviruses by symbiogenesis of insect DNA viruses in endoparasitic wasps.
.
J Insect Physiol.
2003;
;
49
:
:419.
–
432
.
29.
Lorenzi
A
,
Volkoff
AN
.
[Polydnaviruses, a unique example of viral machinery domesticated by parasitoid wasps].
.
Virologie (Montrouge).
2020;
;
24
:
:113.
–
25
.
30.
Strand
MR
,
Pech
LP
.
Immunological basis for compatibility in parasitoid-host relationships.
.
Annu Rev Entomol.
1995;
;
40
:
:31.
–
56
.
31.
Bézier
A
,
Annaheim
M
,
Herbinière
J
,
et al.
Polydnaviruses of braconid wasps derive from an ancestral nudivirus.
.
Science.
2009;
;
323
:
:926.
–
930
.
32.
Petersen
JM
,
Bézier
A
,
Drezen
JM
,
van Oers
MM
.
The naked truth: An updated review on nudiviruses and their relationship to bracoviruses and baculoviruses.
.
J Invertebr Pathol.
2022;
;
189
:
:107718.
.
33.
Murphy
N
,
Banks
JC
,
Whitfield
JB
,
Austin
AD
.
Phylogeny of the parasitic microgastroid subfamilies (Hymenoptera: Braconidae) based on sequence data from seven genes, with an improved time estimate of the origin of the lineage.
.
Mol Phylogenet Evol.
2008;
;
47
:
:378.
–
395
.
34.
Gauthier
J
,
Boulain
H
,
van Vugt
J
,
et al.
Chromosomal scale assembly of parasitic wasp genome reveals symbiotic virus colonization.
.
Commun Biol.
2021;
;
4
:
:104.
.
35.
Burke
GR
,
Thomas
SA
,
Eum
JH
,
Strand
MR
.
Mutualistic polydnaviruses share essential replication gene functions with pathogenic ancestors.
.
PLoS Pathog.
2013;
;
9
:
:e1003348.
.
36.
Burke
GR
,
Walden
KK
,
Whitfield
JB
,
et al.
Widespread genome reorganization of an obligate virus mutualist.
.
PLoS Genet.
2014;
;
10
:
:e1004660.
.
37.
Espagne
E
,
Dupuy
C
,
Huguet
E
,
et al.
Genome sequence of a polydnavirus: insights into symbiotic virus evolution.
.
Science.
2004;
;
306
:
:286.
–
289
.
38.
Burke
GR
,
Simmonds
TJ
,
Thomas
SA
,
Strand
MR
.
Microplitis demolitor bracovirus proviral loci and clustered replication genes exhibit distinct DNA amplification patterns during replication.
.
J Virol.
2015;
;
89
:
:9511.
–
9523
.
39.
Louis
F
,
Bézier
A
,
Periquet
G
,
et al.
The bracovirus genome of the parasitoid wasp Cotesia congregata is amplified within 13 replication units, including sequences not packaged in the particles.
.
J Virol.
2013;
;
87
:
:9649.
–
9660
.
40.
Volkoff
AN
,
Jouan
V
,
Urbach
S
,
et al.
Analysis of virion structural components reveals vestiges of the ancestral ichnovirus genome.
.
PLoS Pathog.
2010;
;
6
:
:e1000923.
.
41.
Legeai
F
,
Santos
BF
,
Robin
S
,
et al.
Genomic architecture of endogenous ichnoviruses reveals distinct evolutionary pathways leading to virus domestication in parasitic wasps.
.
BMC Biol.
2020;
;
18
:
:89.
.
42.
Pichon
A
,
Bézier
A
,
Urbach
S
,
et al.
Recurrent DNA virus domestication leading to different parasite virulence strategies.
.
Sci Adv.
2015;
;
1
:
:e1501150.
.
43.
Burke
GR
,
Simmonds
TJ
,
Sharanowski
BJ
,
Geib
SM
.
Rapid Viral Symbiogenesis via Changes in Parasitoid Wasp Genome Architecture.
.
Mol Biol Evol.
2018;
;
35
:
:2463.
–
2474
.
44.
Pichon
A
,
Bézier
A
,
Barbe
V
,
et al.
Les guêpes parasites ont domestiqué des virus à plusieurs reprises au cours de leur évolution.
.
Med Sci (Paris).
2016;
;
32
:
:699.
–
703
.
45.
Leobold
M
,
Bézier
A
,
Pichon
A
,
et al.
The Domestication of a Large DNA Virus by the Wasp Venturia canescens Involves Targeted Genome Reduction through Pseudogenization.
.
Genome Biol Evol.
2018;
;
10
:
:1745.
–
1764
.
46.
Martin
JL
,
Cao
S
,
Maldonado
JO
,
et al.
Distinct Particle Morphologies Revealed through Comparative Parallel Analyses of Retrovirus-Like Particles.
.
J Virol.
2016;
;
90
:
:8074.
–
8084
.
47.
Cerqueira de Araujo
A
,
Leobold
M
,
Bézier
A
,
et al.
Conserved viral transcription plays a key role in virus-like particle production of the parasitoid Wasp Venturia canescens.
.
J Virol.
2022;
;
96
:
:1.
–
21
.
|