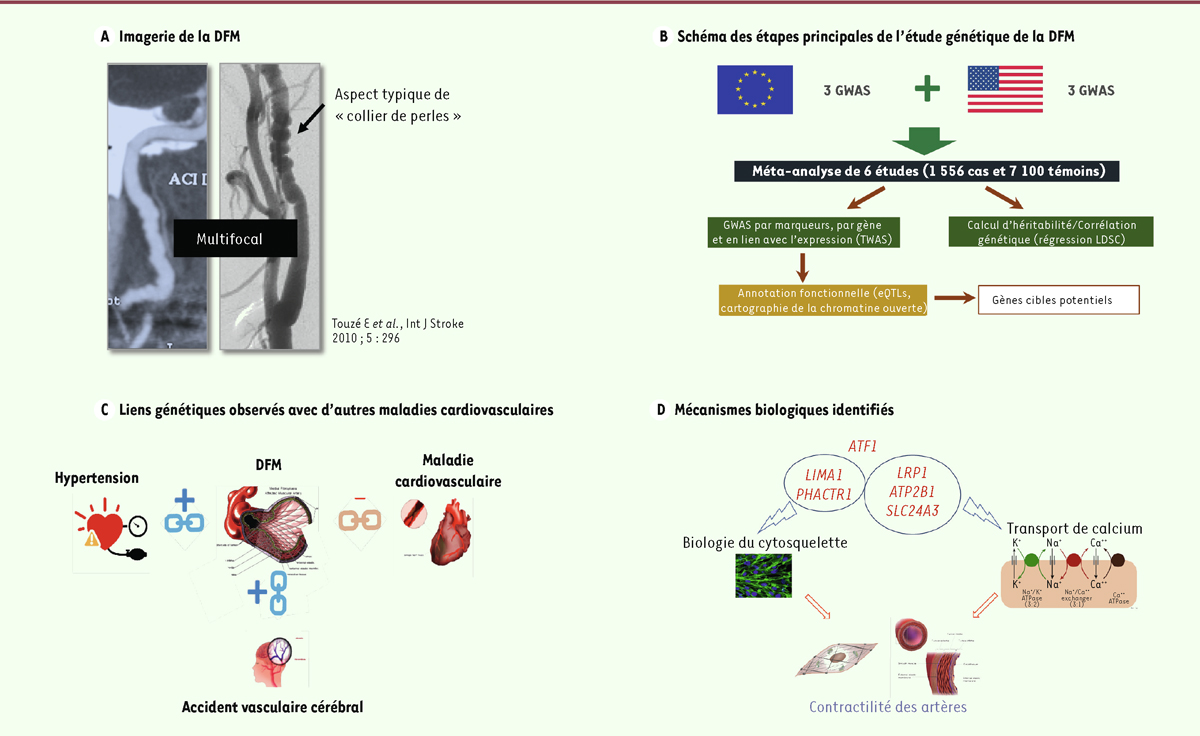
La dysplasie fibromusculaire (DFM) artérielle est une maladie caractérisée par la présence de sténoses et d’anévrismes des artères musculaires 1, comme les artères rénales ou celles issues des troncs supra-aortiques 2 . La forme la plus fréquente (> 80 % des cas) est multifocale : présence de sténoses multiples de l’artère affectée, donnant l’aspect caractéristique « en collier de perles » visible à l’angiographie ou la tomodensitométrie [ 1 ] ( Figure 1A ). La DFM est diagnostiquée principalement chez des femmes d’âge moyen (environ 80 % des cas), avec peu ou pas de facteurs de risque cardiovasculaire comme l’obésité ou la dyslipidémie. Le diagnostic est souvent fortuit lors d’un examen radiologique dans le cadre d’un don de rein pour une greffe, de la recherche de la cause d’une hypertension artérielle atypique ou résistante au traitement, ou encore d’un accident vasculaire cérébral en l’absence de facteur de risque cardiovasculaire associé.
| Figure 1.
Analyse génétique de la dysplasie fibromusculaire artérielle. A.
Imagerie par tomodensitométrie (à gauche) ou angiographie par produit de contraste (à droite), montrant l’aspect typique en « collier de perles » d’une artère atteinte par la maladie.
B.
Schéma des étapes principales de l’étude génétique et méthodes analytiques appliquées.
C.
Illustrations des liens génétiques obtenus pas la méthode LDSR (
linkage disequilibrium score regression
), notamment le lien positif avec l’hypertension artérielle, avec la dissection cervicale et avec l’anévrisme intracrânien, et le lien négatif avec la coronaropathie.
D..
Liste des gènes et illustration des mécanismes biologiques liés à la contractilité des artères, dans lesquels sont impliqués les gènes identifiés comme cibles potentielles de régulation par les locus de prédisposition à la DFM.
|
La recherche génétique sur la DFM a longtemps été infructueuse, par manque d’un modèle génétique et de grandes familles comportant plusieurs sujets atteints permettant une recherche de liaison génétique. Plus récemment, l’analyse d’association génétique de variants fréquents de type SNP ( single nucleotide polymorphism ) avait impliqué un locus de prédisposition à la maladie situé dans le gène PHACTR1 ( phosphatase and actin regulator 1 ) [ 2 ]. Ce locus contient un SNP (rs9349379) fréquent (0,60) qui est associé avec le risque de plusieurs maladies vasculaires, y compris la DFM, les coronaropathies, la migraine et l’hypertension artérielle [ 3 ]. À la suite de ce résultat, nous avons réalisé la première étude d’association pangénomique ( genome-wide association study , GWAS) à la recherche d’autres locus de prédisposition à la DFM.
Nous avons étudié les génotypes d’environ 5,5 millions de SNP fréquents (fréquence de l’allèle mineur > 0,01) dans une méta-analyse de six GWAS (trois réalisées en Europe, dont l’étude française ARCADIA, et trois réalisées aux États-Unis), concernant au total environ 1 550 sujets atteints de la forme multifocale de DFM et environ 7 100 témoins [ 4 ] ( Figure 1B ).
Nous avons d’abord exploré les bases génétiques de la DFM en analysant le génome entier par la méthode de régression du score de déséquilibre de liaison ( linkage disequilibrium score regression , LDSR) [ 5 ], qui permet de quantifier séparément les contributions des effets polygéniques et de divers facteurs de confusion, tels que la stratification de la population 3 . Nous avons ainsi estimé l’héritabilité de la DFM à environ 43 %, ce qui confirme qu’il s’agit d’une maladie polygénique. La méthode LDSR nous a également permis de comparer la DFM avec plusieurs autres maladies auxquelles elle est souvent associée ( Figure 1C ). Nous avons ainsi mis en évidence des corrélations génétiques (notées rg) positives entre la DFM et les valeurs de la pression artérielle systolique (rg = 0,43 ; p = 2 × 10 -9 ) et diastolique (rg = 0,37 ; p = 1 × 10 -8 ). Ce résultat, compatible avec le Tableau clinique, indique qu’une part importante de la base génétique de la DFM serait commune avec celle de la pression artérielle. D’autre part, nous avons identifié des corrélations génétiques positives avec la migraine, décrite chez 25 à 69 % des individus atteints de DFM, avec une forme rare d’accident vasculaire cérébral lié à la dissection d’une artère cervicale, et avec l’anévrisme intracrânien, également plus fréquent chez les femmes. Enfin, étonnamment, nous avons trouvé une corrélation génétique négative entre la DFM et les coronaropathies (rg = – 0,31 ; p = 5 × 10 -5 ), dont les mécanismes génétiques et biologiques restent difficiles à cerner.
Nous nous sommes ensuite intéressés à l’implication fonctionnelle des quatre locus validés par l’association statistique dans l’étude GWAS et à celle d’un gène identifié grâce à l’approche TWAS 4, à partir de données du projet GTEx ( genotype-tissue expression , https://gtexportal.org/home/ ) ( Tableau I ). La totalité des SNP associés à la maladie étant situés dans des séquences non codantes du génome, nous avons émis l’hypothèse qu’ils avaient un effet sur la régulation de gènes voisins. Les données disponibles du projet GTEx indiquaient par exemple que le variant rs9349379 identifié est corrélé avec l’expression de PHACTR1 dans les artères. Grâce à la colocalisation Bayésienne 5 [ 6 ], nous avons trouvé une très forte probabilité ( p proche de 1) que l’allèle à risque associé à la DFM soit le déterminant majeur de l’expression de ce gène dans les artères, ce qui serait la cause de l’augmentation du risque pour la DFM. Ce fut le cas également pour le SNP rs11172113 dans LRP1 ( low-density lipoprotein receptor-related protein-1 ; p proche de 1) , et pour le SNP rs2681492 dans ATP2B1 ( ATPase plasma membrane Ca 2+ transporting 1 , p = 0,98). Pour le locus correspondant au SNP rs 7301566, qui contient notamment les gènes LIMA1 ( LIM domain and actin-binding protein 1 ) et ATF1 ( activating-transcription factor 1 ), nous n’avons pas réussi à identifier le gène cible impliqué dans la maladie, la corrélation avec l’expression de ces gènes dans les artères n’ayant pas impliqué un SNP unique et commun avec l’association à la DFM. À l’aide de cartes génomiques de chromatine ouverte 6 , nous avons confirmé que plusieurs SNP associés à la DFM sont situés dans des éléments régulateurs spécifiques des cellules musculaires lisses, qui sont fonctionnels dans celles des artères, mais pas dans les cellules endothéliales. La confirmation expérimentale, dans des modèles cellulaires dérivés de cellules pluripotentes, de l’hypothèse d’effets régulateurs, sur les gènes voisins, des SNP identifiés est en cours.
Tableau I.
Liste des locus de prédisposition à la DFM identifiés et gènes candidats correspondants.
p
quantifie la significativité statistique de l’association des SNP ou du gène
SLC24A3
à la maladie, mise en évidence par la méthode GWAS ou TWAS.
|
Il convient de noter que les gènes que nous avons identifiés comme des cibles probables des SNP associés à la DFM sont tous impliqués dans des mécanismes biologiques pertinents d’un point de vue physiopathologique ( Figure 1D ). PHACTR1 est impliqué dans l’assemblage des fibres de contrainte de l’actine et interviendrait dans la motilité cellulaire nécessaire pour un alignement optimal des cellules musculaire lisses , qui est perturbé dans les artères affectées par la DFM [ 7 ]. LIMA1, sous réserve de la confirmation de son implication dans la maladie, code également une protéine de liaison à l’actine, et jouerait également un rôle dans le maintien de l’alignement des cellules musculaires lisses dans la paroi des artères. Quant à LRP1 , il code une protéine dont le rôle a été très étudié dans de multiples processus cellulaires, comme le remodelage de la matrice extracellulaire et la migration des cellules musculaires lisses vasculaires [ 8 ]. D’autre part, ATP2B1 [ 9 ] et SLC24A3 (solute carrier family 24 member 3) [ 10 ] codent respectivement un transporteur du calcium impliqué dans l’exportation de cet ion vers l’espace extracellulaire et un échangeur transmembranaire Na + /K + /Ca 2+ . Il s’agit d’acteurs de l’homéostasie du calcium intracellulaire, un élément régulateur majeur de la fonction de vasoconstriction et vasodilatation, ce qui souligne l’importance de ce mécanisme vasculaire du maintien de l’intégrité artérielle dans la survenue des lésions dysplasiques.
En résumé, cette première étude GWAS portant sur la DFM a permis d’établir la carte génétique la plus complète jusqu’à présent pour cette maladie, et d’identifier des locus de prédisposition et des gènes cibles potentiels. Ces découvertes fournissent de nouvelles pistes pour comprendre les mécanismes biologiques de la sténose artérielle en l’absence d’athérosclérose. Des études moléculaires approfondies sont en cours, qui portent sur la régulation des gènes candidats et tentent d’évaluer leur potentiel thérapeutique.