| |
| Med Sci (Paris). 38(10): 839–841. doi: 10.1051/medsci/2022126.Scoop : les mutations « synonymes » ne sont pas neutres ! Chroniques génomiques Bertrand Jordan1* 1Biologiste, généticien et immunologiste, président d’Aprogène (Association pour la promotion de la génomique)
,
13007Marseille
,
France MeSH keywords: Évolution biologique, Humains, Mutation, Mutation faux-sens, Mutation ponctuelle |
Lorsqu’une mutation ponctuelle se produit au sein d’une séquence de nucléotides codant une protéine, on a trois possibilités : mutation non sens faisant apparaître un codon stop et conduisant à la production d’une protéine tronquée, généralement non fonctionnelle ; mutation faux sens, modifiant la nature de l’acide aminé qui sera inséré à cet endroit dans la chaîne polypeptidique, la protéine sera alors plus ou moins fonctionnelle selon les cas ; et enfin, mutation synonyme (ou « silencieuse ») qui ne change pas l’acide aminé (plusieurs codons pouvant spécifier le même acide aminé) et donc ne modifie pas la structure et les propriétés de la protéine. On s’attend donc à ce que les mutations synonymes n’aient aucun effet physiologique et soient neutres du point de vue de l’évolution [
7
] (
→
).
(→) Voir la Synthèse de O. Tenaillon et I. Matic, page 777 de ce numéro
C’est presque un dogme sur lequel reposent, par exemple, la détection de séquences fortement sélectionnées (du fait qu’elles contiennent plus de mutations synonymes que de mutations faux sens ou non sens), comme nous l’avons vu récemment pour les Sébastes [
1
](
→
) ou encore la notion d’horloge moléculaire qui suppose que ces mutations silencieuses s’accumulent au fil du temps sans subir de pression sélective. Depuis quelques années, ce dogme a été remis en cause [
2
] (
→
) et l’on a notamment montré que, dans certains cas, une mutation synonyme pouvait, chez l’homme, être la cause d’une affection génétique. On suppose que de telles mutations pourraient perturber l’épissage du préARN messager, ou modifier sa conformation, ou encore provoquer l’apparition d’un codon synonyme mais rarement utilisé dans l’organisme en cause
1
. Une nouvelle étude, parue en juin 2022 dans la revue
Nature
[
3
], et son commentaire [
4
] examinent cette question de manière systématique et exhaustive. En bref, ses auteurs ont choisi 21 gènes non essentiels chez la levure
Saccharomyces cerevisiae
, ont introduit (presque) toutes les mutations possibles dans une zone de chaque séquence codante, et ont mesuré de manière précise la valeur adaptative (
fitness
) de chacun de ces mutants par rapport à la souche « sauvage ». Ils ont ainsi comparé près de dix mille mutants à la souche d’origine et ont constaté que les mutations silencieuses diminuaient autant la valeur adaptative que les mutations faux sens – résultat indiscutable car la méthodologie est impeccable même s’il s’agit bien sûr d’un organisme particulier et d’un choix de gènes non essentiels. C’est un résultat important qui, s’il est confirmé dans d’autres systèmes, va remettre en question nombre de certitudes.
(→) Voir la Chronique génomique de B. Jordan,
m/s
n° 2, février 2022, page 218
(→) Voir la Chronique génomique de B. Jordan,
m/s
n° 4, avril 2012, page 435
|
Les paramètres de l’étude
Le génome de la levure porte un peu plus de six mille gènes, mais seuls environ mille d’entre eux sont nécessaires à la croissance en milieu riche [
5
], comme l’ont montré des expériences de délétion systématique. Les gènes non essentiels peuvent participer à la croissance mais ne sont pas indispensables dans ces conditions. L’idée est donc de choisir un jeu de gènes non essentiels (mais dont la délétion ralentit la croissance), intervenant dans une large variété de processus (métabolisme, transcription, traduction, synthèse de la paroi, etc.) et présentant une large gamme de niveaux d’expression (couvrant un facteur 1 000). Pour chacun d’eux, on choisit (au hasard) une séquence de 150 nucléotides située dans la première moitié de la séquence codante et on synthétise les 450 (150 x 3) variants possibles, qui sont ensuite utilisés (en masse) pour introduire les mutations correspondantes dans la levure par la technique CRISPR-Cas9. On séquence enfin le mélange de mutants pour vérifier la présence des mutations. Ce processus est répété pour chacun des 21 gènes et devrait, en théorie, fournir 9 450 (21 x 450) mutants ; on en détecte en fait un total de 8 341, ce qui est déjà un très bon résultat. On va ensuite cultiver le mélange de l’ensemble des mutants obtenus (pour un gène donné) avec un aliquote de la souche sauvage, et mesurer par séquençage la fréquence de chaque mutant (et de la souche sauvage) au départ et après une trentaine de générations (doublements du nombre de cellules). Si une mutation diminue la valeur adaptative d’une souche, sa fréquence par rapport au témoin sauvage aura baissé entre le début et la fin de l’expérience. En pratique, le mélange de mutants pour un gène avec le témoin sauvage est cultivé à 30 °C en milieu riche et des aliquotes sont prélevés pour effectuer un séquençage au début et après 48 h (ce qui correspond à 29 générations). Le rapport des concentrations pour un mutant entre début et fin, comparé au même rapport pour le témoin, permet de calculer sa valeur adaptative (
fitness
), définie comme l’augmentation du nombre de cellules du mutant en une génération par rapport au même paramètre chez le témoin. Une mutation totalement neutre donnerait donc une valeur égale à 1.
|
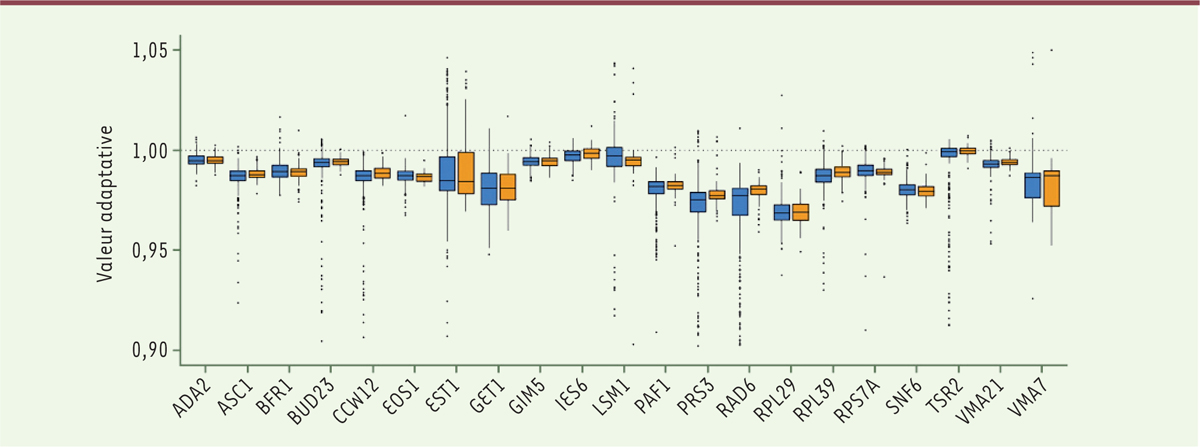
Comme on pouvait s’y attendre
(
Figure 1
)
, la valeur adaptative est inférieure à 1 pour presque tous les mutants (bien que quelques-uns l’augmentent, étant donc « avantageux » dans les conditions de culture utilisées). Pour les mutants non sens (qui sont au nombre de 169) la médiane de cette valeur est de 0,94, un chiffre relativement proche de 1 dû au fait qu’il s’agit de gènes non essentiels. Notons qu’en 29 générations, cela aboutit à une concentration relative de 0,166 (0,94 puissance 29) : la multiplications des levures au cours de la culture exacerbe les différences entre mutants et témoin et permet de mesurer précisément de faibles écarts de valeur adaptative. Pour les 6 306 mutants faux sens, cette valeur est de 0,989, logiquement plus proche de 1 que pour les non sens. Mais la surprise vient des 1 866 mutants synonymes dont la valeur adaptative médiane est de …0,988, soit quasiment la même que pour les mutants faux sens, alors que l’on s’attendrait à un chiffre nettement plus proche de 1. Bien entendu ces valeurs varient selon le gène et le mutant
(
Figure 1
)
, mais faux sens et synonymes restent toujours très proches.
 | Figure 1.
Valeurs adaptatives pour les mutants faux sens (en bleu) et synonymes (en bistre) pour chacun des 21 gènes étudiés
(voir
www.yeastgenome.org
pour leur description). Les mutants qui donnent des valeurs extrêmes sont représentés par des points en dehors de la cartouche qui regroupe la grande majorité des valeurs. On voit que ces valeurs varient selon les mutants (et qu’elles sont supérieures à 1 pour quelques-uns), que leur médiane (barre au milieu de la cartouche) diffère selon les gènes, mais que les valeurs sont, sauf exception, quasiment identiques pour les mutations faux sens et synonymes. Les mutations silencieuses ne sont donc pas « plus neutres » que les faux sens (extrait partiel et remanié de la figure 2 de [
3
]).
|
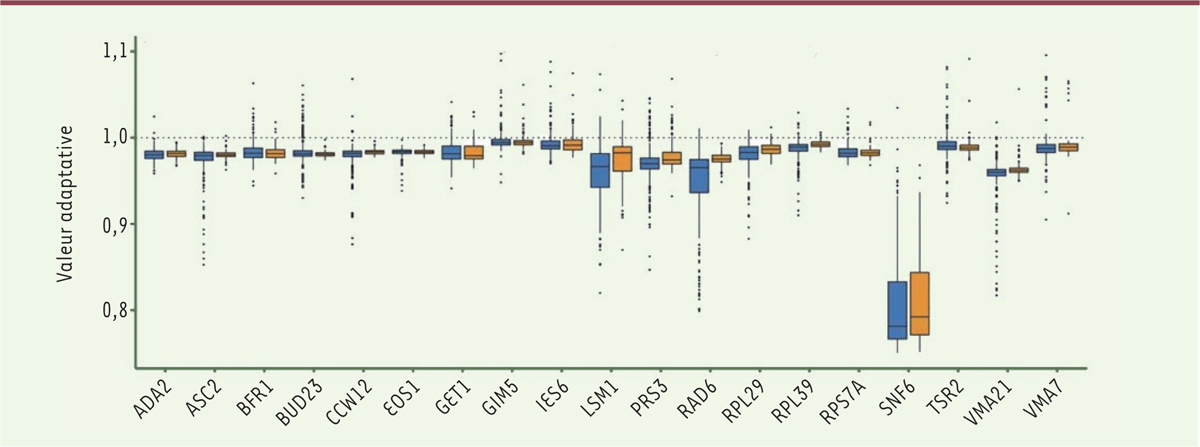
Les auteurs ont voulu examiner l’effet des conditions de culture sur ces résultats. Ils ont notamment remplacé le milieu YPD utilisé (1 % extrait de levure [Y], 2 % peptone [P], 2 % dextrose ou D-glucose [D]) par le milieu YPE (le glucose est remplacé par de l’éthanol [E]). Ils obtiennent alors des résultats assez différents pour les chiffres de valeur adaptative
(
Figure 2
)
, mais toujours quasiment identiques pour les mutations faux sens et synonymes. D’autres milieux donnent le même type de résultat, qui n’est donc pas un artefact lié aux conditions de culture.
 | Figure 2.
Légende identique à celle de la Figure 1, mais les levures sont ici cultivées en milieu YPE au lieu de YPD.
Les chiffres de valeur adaptative sont assez différents (notamment pour le gène
SNF6
, impliqué dans le remaniement de la chromatine), mais restent quasiment identiques pour les mutations faux sens (en bleu) et synonymes (en bistre) (extrait partiel et remanié de la figure 8 des données supplémentaires [
extended data
] de [
3
]).
|
|
Des limites, mais aussi des implications importantes
On ne peut qu’être admiratif devant la qualité de ce très joli travail, qui comporte aussi de nombreuses expériences complémentaires pour vérifier la reproductibilité des données et éliminer les artéfacts. Un
design
ingénieux amplifie l’effet de petites différences de valeur adaptative, le système CRISPR-Cas9 est utilisé à bon escient pour saturer en mutations une région de chaque gène, et la lecture du résultat par séquençage « en masse » du mélange de mutants permet d’amasser un important ensemble de données. Les multiples contrôles (non rapportés ici) confirment la fiabilité des résultats. La démonstration, sur un effectif important, que les mutations synonymes (ou silencieuses) ne sont pas plus neutres que les mutations non sens est imparable, du moins dans les limites du système utilisé. Limites qui tiennent essentiellement au choix de gènes non essentiels, comme le confirme la faible baisse de valeur adaptative pour les mutations non sens : de 1 à 0,94, alors que pour un gène essentiel, la valeur adaptative d’une mutation non sens est
a priori
proche de zéro puisque le mutant n’est pas viable. On peut donc se demander si la différence entre mutations faux sens et mutations synonymes ne serait pas plus nette si les gènes mutés étaient essentiels – on ne voit pas,
a priori
, ce qui interdirait de répéter une telle étude sur un jeu de gènes indispensables à la croissance.
Reste que ce travail montre que, au moins dans certaines conditions, on ne peut plus partir du principe qu’une mutation synonyme est neutre. C’est bien ennuyeux car cette neutralité supposée joue un rôle essentiel en génétique évolutive, que ce soit pour identifier des gènes ayant fait l’objet de sélection positive – grâce à leur richesse relative en mutations synonymes – ou pour calibrer une « horloge moléculaire » fondée sur l’accumulation de mutations silencieuses (donc en principe neutres) censées indiquer le taux de mutation en l’absence de sélection [
6
]. C’est ennuyeux, mais « seule la vérité est révolutionnaire » ! Il est donc important d’examiner si les résultats de Shen
et al
[
3
] sont généralisables à des gènes essentiels, à d’autres systèmes biologiques, auquel cas il faudrait réviser de nombreux raisonnements en génétique évolutive…
|
L’auteur déclare n’avoir aucun lien d’intérêt concernant les données publiées dans cet article.
|
Footnotes |
1.
Jordan
B
.
La longévité des sébastes.
.
Med Sci (Paris).
2022;
;
38
:
:218.
–
21
.
2.
Jordan
B.
The sound of silence. Med Sci (Paris).
2012;
;
28
:
:435.
–
437
.
3.
Shen
X
,
Song
S
,
Li
C
,
Zhang
J
.
Synonymous mutations in representative yeast genes are mostly strongly non-neutral.
.
Nature.
2022;
;
606
:
:725.
–
31
.
4.
Sharp
N
.
Mutations matter even if proteins stay the same.
.
Nature.
2022;
;
606
:
:657.
–
9
.
5.
Svensson
JP
,
Pesudo
LQ
,
Fry
RC
,
et al.
Genomic phenotyping of the essential and non-essential yeast genome detects novel pathways for alkylation resistance.
.
BMC Syst Biol.
2011;
;
5
:
:157.
.
6.
Morgan
GJ
.
Emile Zuckerkandl, Linus Pauling, and the molecular evolutionary clock.
.
J Hist Biol.
1998;
;
31
:
:155.
–
178
.
7.
Tenaillon
O
,
Matic
I
.
L’impact des mutations neutres sur l’évolvabilité et l’évolution des génomes.
.
Med Sci (Paris).
2022;
;
38
:
:777.
–
85
.
|