La régulation de l’expression des gènes comprend une multitude de mécanismes pouvant cibler chacune des étapes depuis la transcription des ARN à partir de l’ADN génomique et leur maturation, jusqu’à la traduction des ARN messagers (ARNm) en protéines. Au cœur de ces mécanismes, les ARN non-codants (ARNnc), longs ou petits, jouent très souvent un rôle crucial [ 1 ] ( → ).
(→) Voir la Synthèse de Y. Abel et al ., m/s n° 3, mars 2014, page 297
Les microARN (miARN), de taille comprise entre 19 et 25 nucléotides, constituent une classe majeure de petits ARN impliqués dans la régulation post-transcriptionnelle de l’expression des gènes [ 2 ] ( → ) : les miARN, au sein du complexe RISC ( RNA induced silencing complex ), en s’appariant à la région 3’ non-traduite (3’-UTR) des ARNm ciblés, inhibent leur traduction et provoquent ensuite leur dégradation [ 3 ] ( → ).
(→) Voir la Synthèse de L. Fressigné et M.J. Simard et al ., m/s n° 2, février 2018, page 137
(→) Voir le Repères de F. Dautry et C. Ribet, m/s n° 8-9, août-septembre 2004, page 815
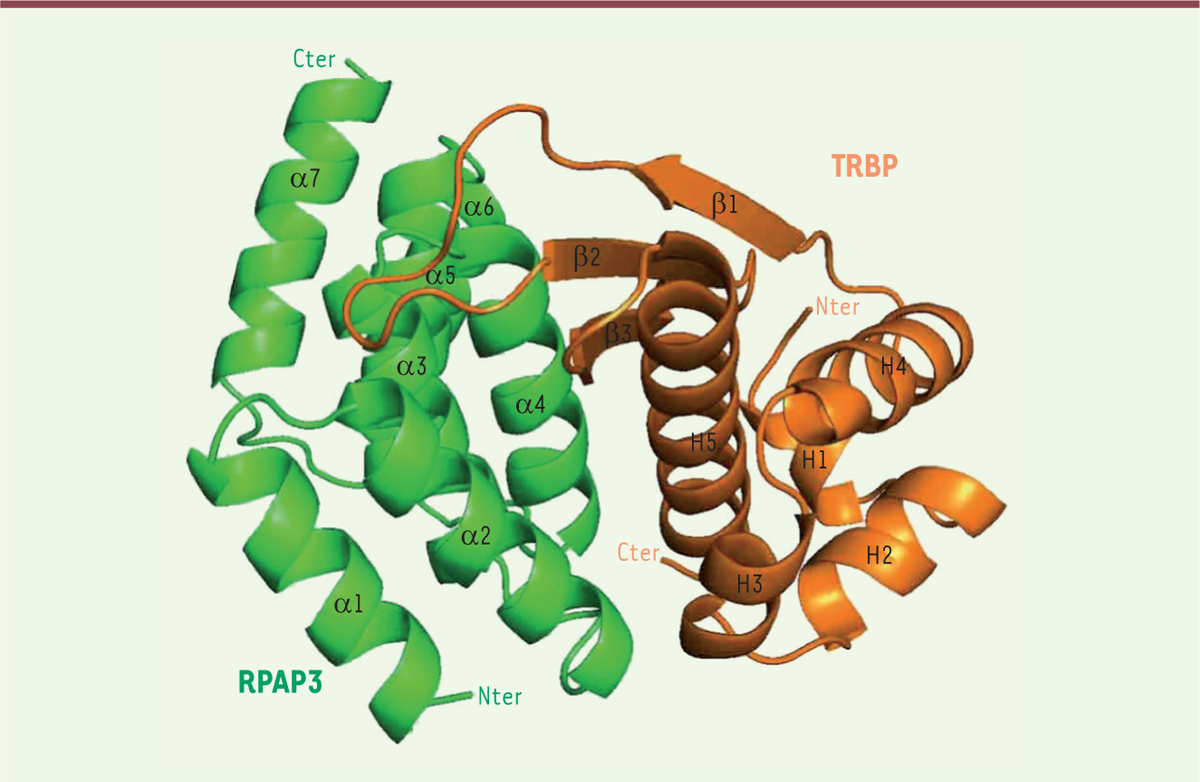
La protéine TRBP ( TAR-RNA binding protein ) joue un rôle essentiel dans la biogenèse des miARN [ 4 ]. En effet, une fois le transcrit primaire clivé par la ribonucléase nucléaire (RNase) de type III Drosha accompagnée de son cofacteur DGCR8 ( DiGeorge syndrome critical region 8 ), le miARN précurseur ou pré-miARN résultant est exporté dans le cytoplasme, où il sera clivé par la RNase de type III, Dicer, accompagnée de l’un de ses cofacteurs, PACT (ou PRKRA : protein activator of the double-stranded RNA-dependent kinase-PKR ) ou TRBP (ou TARBP2) [ 2 ]. Ce second clivage produit le miARN double brin mature, dont l’un des brins sera chargé au sein du complexe effecteur de l’ARN interférence RISC, composé notamment d’une protéine de la famille Argonaute (AGO). Or, la nature du duplex produit à l’issue du clivage par Dicer dépend du cofacteur associé à cette dernière. En effet, TRBP et PACT, malgré leur similarité de séquence, conduisent à des spécificités différentes de clivage des ARN double-brin substrats par Dicer, ce qui peut produire des miARN matures de taille variable [ 4 , 5 ].
TRBP, en plus de son rôle au cours de la biogenèse des miARN, est impliquée dans la réplication du virus de l’immunodéficience humaine (VIH) [ 6 ]. Comme son acronyme le laisse entendre, TRBP a d’abord été caractérisée par son interaction avec l’élément ARN transactivateur du VIH (TAR RNA pour trans activating response RNA ), une structure en tige boucle de l’ARN viral [ 6 ]. La formation de cette structure particulière, située dans la partie 5’ LTR ( long terminal repeat ) du génome viral, limite la traduction de ce dernier. La fixation de TRBP sur l’élément TAR déstabilise la structure en tige boucle, ce qui facilite alors la traduction [ 6 ]. Ainsi, TRBP contribue à l’infection virale. Récemment, il a même été suggéré que c’est le clivage de l’élément TAR par Dicer, associée à TRBP, qui permettrait de déstabiliser l’élément TAR, produisant par la même occasion des miARN viraux favorisant la multiplication virale en limitant l’apoptose des cellules infectées [ 7 ]. TRBP agit également sur d’autres aspects de la réplication virale en inhibant directement la protéine kinase R (PKR), activée en présence d’ARN exogènes, ou en l’inhibant indirectement, en empêchant son interaction avec l’une de ses protéines activatrice, la protéine PACT [ 6 ]. Il a été montré récemment que TRBP pouvait en outre recruter la 2’- O -méthyltransférase FTSJ3 ( FtsJ RNA 2’-O-methyltransferase 3 ) pour méthyler l’ARN viral, limitant ainsi son caractère immunogène [ 8 ].