| |
| Med Sci (Paris). 38(6-7): 600–607. doi: 10.1051/medsci/2022079.Le virus SARS-CoV-2 n’a pas « d’origine » Roger Frutos,1* Laurent Gavotte,2 and Christian A. Devaux3,4 1Cirad, UMR 17, Intertryp
,
Montpellier
,
France 2Espace-Dev, université de Montpellier
,
Montpellier
,
France 3Microbes, évolution, phylogénie et infection (MEPHI), Aix-Marseille université, IRD, AP-HM, IHU-Méditerranée infection
,
Marseille
,
France 4CNRS
,
Marseille
,
France |
Vignette (© CDC/Alissa Eckert, MSMI ; Dan Higgins, MAMS).
En janvier 2020, le monde entier a entendu parler pour la première fois d’une nouvelle maladie, la COVID-19 (
coronavirus disease 2019
), causée par un nouveau bétacoronavirus, le virus SARS-CoV-2 (
severe acute respiratory syndrome-coronavirus-2
) [
1
–
4
]. Depuis son apparition, l’origine de ce virus a fait l’objet d’une vive polémique. La pandémie de COVID-19 a officiellement débuté le 8 décembre 2019 au marché de gros de Huanan (le
Huanan seafood wholesale market
, HSWM) à Wuhan, en République Populaire de Chine [
3
,
4
]. Cependant, cela ne correspond qu’à la date et au lieu officiels, des études phylogénétiques ont en effet montré que le virus circulait certainement déjà à l’automne 2019 [
5
–
8
].
|
Être ou ne pas être… un virus pandémique
Le SARS-CoV-2 est un virus pandémique ; mais que signifie « être un virus pandémique » ? Les virus pandémiques émergents sont, par définition, des virus multi-hôtes, c’est-à-dire qu’ils sont naturellement capables d’infecter des individus d’espèces différentes. Ces virus ont la particularité de reconnaître des récepteurs cellulaires ubiquistes, c’est-à-dire présents chez plusieurs espèces. Cela leur permet de survivre même si une ou plusieurs de ses espèces hôtes disparaissent. Ces récepteurs sont des protéines essentielles pour l’organisme hôte et sont conservés. Le récepteur reconnu par le SARS-CoV-2 est l’ACE2 (
angiotensin I converting enzyme 2
) [
9
], un récepteur impliqué dans la régulation de la pression artérielle et l’homéostasie cardiaque et rénale chez les vertébrés. C’est une protéine présente chez tous les vertébrés et absolument vitale pour l’hôte. La fixation du virus à son récepteur peut être, initialement, de faible affinité, le virus évoluant par la suite en s’adaptant à son nouvel hôte avec l’acquisition d’une plus forte affinité pour ce récepteur. Un autre paramètre important de l’évolution du virus est sa capacité à éviter les défenses immunitaires de l’hôte. Pour être pandémique, un virus doit présenter une pathogénicité relativement faible avec une période d’incubation importante favorisant ainsi sa transmission au sein de la population. C’est exactement ce que l’on voit avec le SARS-CoV-2. La COVID-19 est une maladie présentant la plupart du temps des symptômes légers ou modérés, avec un taux de mortalité actuellement estimé entre 1 et 2 % [
10
], mais certainement moindre, car beaucoup de cas asymptomatiques ne sont pas pris en compte dans le nombre total d’individus infectés. Le nombre élevé de décès qui a été observé est en fait la conséquence de la taille, énorme, de la population humaine [
11
]. La mortalité n’est le plus souvent pas directement liée à un effet pathogène du virus, mais plutôt à la faiblesse des patients qui sont décédés et qui étaient en général âgés et/ou atteints de comorbidités majeures.
Pour comprendre comment émerge une nouvelle maladie infectieuse, il est nécessaire de faire la distinction entre les concepts de
cas primaire
et de
cas index
, ou plutôt
des cas index
. Le cas primaire est la première personne à être infectée par un virus donné : il correspond à la primo-infection dans la population humaine. Le cas index est, quant à lui, défini comme la première personne à avoir été diagnostiquée avec la nouvelle maladie. Ce sont deux notions très différentes. L’identification du cas index est souvent rétrospective, et il faut d’ailleurs distinguer le
cas index réel
, c’est-à-dire la première personne ayant effectivement présenté les symptômes de la maladie, mais qui est souvent mal diagnostiquée et inconnue, du
cas index officiel
qui est la personne présentant ces symptômes et qui a été officiellement reconnue comme la première. Cette distinction est importante du point de vue de la dynamique de la maladie. Entre la survenue du cas primaire et celle du cas index officiel, le virus se propage, alors indétecté (faute de diagnostic établi), dans la population humaine. Le cas index réel peut parfois être identifié, mais le cas primaire n’est jamais retrouvé. C’est très exactement ce qui se passe pour la COVID-19 : il existe un cas index officiel, daté du 8 décembre 2019, qui a été déclaré comme tel par décision administrative, mais qui n’est pas le cas index réel. Le cas primaire, lui, n’est pas connu et ne sera probablement jamais identifié.
Une autre caractéristique des virus pandémiques est leur capacité à évoluer dans l’hôte par un processus de « quasi-espèce »
1
, qui est propre aux virus à ARN [
12
–
14
]. La grande majorité des virus zoonotiques, qui se transmettent à l’homme à partir d’animaux infectés, sont d’ailleurs des virus à ARN [
15
] et ce n’est pas une coïncidence. Le mode d’évolution « quasi-espèce » correspond à la génération d’une multitude de variants couvrant toutes les possibilités de mutations du génome viral. La quasi-totalité de ces variants ne sont pas viables et sont par conséquent éliminés, mais certains d’entre eux portent des mutations avantageuses, augmentant leur affinité pour leur récepteur cellulaire, ou leur permettant d’éviter les défenses immunitaires, ou augmentant leur capacité d’exploitation de la machinerie de la cellule hôte. La séquence de leurs génomes mutés sera alors le point de départ d’une nouvelle génération de variants et, progressivement, une séquence optimale sera sélectionnée, sous la pression de l’hôte lui-même. Cette évolution conduira parfois à des mutations rendant les virus très hautement transmissibles. Cela représente bien sûr un net avantage sélectif et les virus porteurs de ces mutations seront rapidement sélectionnés. Ce mécanisme d’évolution et d’adaptation chez l’hôte par le biais d’un processus de « quasi-espèce » est continuel et, si l’hôte peut lutter contre le virus
via
ses défenses immunitaires, celui-ci s’adaptera à son tour en produisant de nouveaux variants capables de leur échapper. C’est ce que nous voyons aujourd’hui dans le cas de la COVID-19 avec l’émergence et la diffusion régulières de nouveaux variants remplaçant les précédents, comme les variants Delta ou Omicron, qui échappent, au moins partiellement, à la protection induite par une primo-infection avec un autre variant, ou, même, par les vaccins conçus à partir du virus initial (virus inactivé ou protéine recombinante) ou sur la base de la séquence de ce dernier (pour les vaccins à ARN). De nouveaux variants continueront donc à apparaître à cause de ces différents processus. Le SARS-CoV-2 lui-même peut ainsi être vu comme un variant très transmissible à l’homme ayant évolué à partir d’une forme antérieure du même virus circulant déjà chez l’homme mais qui était insuffisamment transmissible pour déclencher une maladie, et que l’on pourrait alors appeler SARS-CoV-2-AD pour «
ante disease
».
|
Une confusion entre infection et maladie Une maladie infectieuse émergente est une maladie et donc, par définition, un problème médical. Hélas, elle n’est vue que comme un problème exclusivement médical. Considérer une pandémie, telle que celle de la COVID-19, uniquement comme une maladie est à l’origine de nombreuses confusions. Maladie et infection sont en fait souvent confondues. Une maladie est un concept médical défini par la présence d’un ensemble spécifique de symptômes, un syndrome, que les médecins peuvent reconnaître et auxquels un nom est donné. L’art du médecin est donc d’identifier l’ensemble de ces symptômes, de nommer la maladie, puis de prescrire le traitement adapté. La COVID-19 est simplement la maladie qui a été identifiée à Wuhan. À l’inverse, une infection est un concept biologique qui correspond à la multiplication d’un agent pathogène au sein d’un hôte, mais qui ne suppose pas obligatoirement l’apparition de symptômes, donc d’une maladie. Les cas asymptomatiques jouent ainsi un rôle majeur dans la dynamique d’expansion d’un agent pathogène, mais ils ne sont pas visibles pour les médecins qui se concentrent uniquement sur les symptômes. La maladie n’est que le point final d’un processus qui a commencé bien avant avec l’infection du premier cas humain, la primo-infection. Une maladie infectieuse émergente totalement nouvelle comme la COVID-19 est, par définition, une maladie sans syndrome connu ni aucun nom. C’est l’origine du problème. Les médecins ne réalisent la présence d’une maladie inconnue que lorsqu’ils sont confrontés à un flux de patients présentant le même ensemble atypique de symptômes. La détection de l’épidémie est ainsi décalée et ne se produit qu’après que le virus se soit propagé dans la population et qu’il ait été identifié. Il est à ce stade trop tard pour essayer d’endiguer la dispersion du virus, il s’est déjà dispersé. Des actions nationales n’ont été envisagées qu’après que l’Organisation mondiale de la santé (OMS) ait déclaré la COVID-19 comme une pandémie, en mars 2020. Il était trop tard pour la prévention et la seule option restante était de soigner les malades. La situation mondiale actuelle, avec la COVID-19, montre ainsi clairement qu’il ne faut pas se focaliser sur l’origine de la maladie (le cas index) mais plutôt sur l’origine de la primo-infection (le cas primaire). |
Traquer le virus dans la nature
La COVID-19 étant considérée comme une « maladie zoonotique », c’est-à-dire une maladie provenant d’animaux, la recherche de l’origine du SARS-CoV-2 s’est immédiatement orientée vers la faune sauvage. Cette recherche des ancêtres sauvages du SARS-CoV-2 suit la théorie du «
Spillover
» (du débordement, en français). Cette théorie stipule qu’il doit exister un « réservoir » et une « espèce animale intermédiaire » augmentant l’exposition au même virus que celui à l’origine de l’épidémie et facilitant sa diffusion chez l’homme [
16
]. Cette théorie est à l’origine de la recherche d’espèces intermédiaires et de projets de criblage afin d’identifier de potentiels virus zoonotiques circulant dans la nature. Dans le contexte de la COVID-19, cette espèce intermédiaire était censée faire le lien entre les chauves-souris, réservoir originel putatif du virus [
17
,
18
], et l’homme, hôte receveur final. Cependant, aucune des prédictions du modèle «
Spillover
» n’a jamais été confirmée à ce jour, et l’hypothèse du rôle du pangolin comme espèce intermédiaire a été invalidée [
19
–
21
]. Aucun intermédiaire, réservoir, ni aucun virus SARS-CoV-2 (de séquence identique) n’ont été signalés dans la nature. Les virus pandémiques ne représentent que l’évolution dans l’espèce humaine d’un groupe de virus qui, chez d’autres hôtes, évoluent différemment, donnant des virus apparentés mais cependant différents. Le SARS-CoV-2 n’est donc qu’une forme humaine de la métapopulation de virus SARS, le SARS-CoV, l’agent causal du SARS, apparu en 2002, en étant une autre. À ce jour, seuls des virus apparentés au SARS-CoV-2 ont été trouvés dans la nature, mais jamais le SARS-CoV-2 lui-même. Une exception concerne les animaux en élevage ou en captivité qui ont été contaminés par l’homme et qui peuvent contaminer l’homme en retour [
22
]. Les séquences les plus proches du SARS-CoV-2 trouvées dans la nature ont été obtenues chez des virus de chauves-souris, en particulier RaTG13 (qui a 96 % d’identité avec le SARS-CoV-2) qui a été la première séquence identifiée à partir d’une chauve-souris (
Rhinolophus affinis
) dans une mine de cuivre abandonnée à Tong Guan (canton de Mojiang, Yunnan, République Populaire de Chine) [
23
,
24
]. D’autres séquences ont été obtenues par la suite : RmYN02 (93,3 % d’identité), également identifiée dans la province du Yunnan chez une autre espèce de chauve-souris,
Rhinolophus malayanus
[
25
], RshSTT182 (92,6 % d’identité) et RshSTT200 (92,6 % d’identité), chez des spécimens de
Rhinolophus shameli
capturés en 2010 au Cambodge [
26
], ou encore RacCS203 (95,86 % d’identité) chez
Rhinolophus acuminatus,
capturée en Thaïlande [
27
].
|
Les narratifs de l’accident de laboratoire
L’impossibilité de trouver un réservoir, une espèce intermédiaire ou un virus SARS-CoV-2 dans la nature a conduit au développement de ce que l’on appelle le narratif, ou plutôt les narratifs, de l’accident de laboratoire [
28
,
29
]. Il faut bien parler des narratifs au pluriel car il s’agit en réalité de l’association de plusieurs récits contradictoires. Ce récit s’appuie sur le fait que Wuhan, où la COVID-19 a été décrite pour la première fois, abrite l’Institut de virologie de Wuhan (WIV), qui gère un laboratoire de haut niveau de sécurité (P4/L4) et où des travaux de recherche avaient été menés auparavant sur le SARS-CoV et d’autres coronavirus [
30
]. Une équipe du WIV a collecté des échantillons de chauves-souris dans la mine de cuivre de Tong Guan, où un incident impliquant six mineurs tombés malades, dont trois sont décédés, avait eu lieu en 2012. La séquence RaTG13 [
23
,
24
] dont nous avons parlé ci-dessus vient de l’un de ces échantillons.
Le récit du possible accident survenu dans ce laboratoire est composite : le SARS-CoV-2 proviendrait de la mine de Mojiang où des membres du WIV ont été contaminés ; le SARS-CoV-2 aurait évolué dans les poumons des mineurs hospitalisés ; le SARS-CoV-2 se serait accidentellement échappé du laboratoire du WIV ; et le SARS-CoV-2 aurait été conçu au WIV et s’est accidentellement échappé ou a été volontairement libéré. Ces narratifs sont souvent considérés ensemble. Pourtant, ils sont contradictoires et s’excluent mutuellement. Si le SARS-CoV-2 est un virus naturel présent dans la mine de Tong Guan, il ne peut provenir du génie génétique et
vice versa
. Si le SARS-CoV-2 a été propagé par des membres du personnel du WIV qui se sont infectés dans la mine de Tong Guan, il ne peut provenir d’un accident de laboratoire et
vice versa
. Si le SARS-CoV-2 a évolué dans les poumons des mineurs hospitalisés à Kunming en 2012, il ne peut ni avoir contaminé les agents du WIV qui se sont rendus dans la mine un an plus tard en 2013, ni être sorti d’un laboratoire situé à 1 500 km de là, à Wuhan, sept ans plus tard. RaTG13 n’est par ailleurs qu’une séquence virtuelle incomplète (les extrémités 3’ et 5’ sont manquantes) et non un virus. C’est une séquence qui n’existe que dans un ordinateur et aucun virus « RaTG13 » n’a jamais été cloné ni mis en culture. Il ne peut donc ni avoir fait l’objet de passages en culture de cellules, ni d’ingénierie génétique, ni s’être échappé d’un laboratoire. RaTG13 est la seule séquence relativement proche du SARS-CoV-2 trouvée à Tong Guan. L’analyse des dossiers médicaux des six mineurs par des cliniciens et des radiologues, a permis de rejeter l’hypothèse d’une infection par le SARS-CoV-2, sur la base de nombreux symptômes incompatibles avec la COVID-19 [
31
]. Il n’y a actuellement strictement aucun élément concret permettant de soutenir l’hypothèse d’une fuite de laboratoire. Ce sont donc des suppositions qui se contredisent. Il y a aussi, et c’est beaucoup plus grave, de fausses informations et de faux résultats, comme, par exemple, les tests montrant l’existence d’une réponse anticorps IgG positive contre le SARS-CoV chez les quatre mineurs testés, relayé par Rahalkar et Bahulikar [
29
]. Aucun article ne mentionne des tests permettant la détection d’IgG sériques spécifiques, et encore moins des résultats positifs, et les références données par Rahalkar and Bahulikar [
29
], en soutien à cette affirmation, ne mentionnent aucun test anticorps. L’un des mineurs, présenté comme « IgG positif pour SARS-CoV », portait d’ailleurs la mention de test SARS-CoV négatif dans son dossier médical. Finalement, aucun test de ce type n’a été réalisé en 2012, sur les 13 échantillons issus des quatre mineurs hospitalisés à Kunming et reçus par le WIV. Ces mineurs ont effectivement été testés, mais par PCR (
polymerase chain reaction
) ciblant l’ARN polymérase ARN dépendante (RdRp) de trois virus : Ebola, Nipah et SARS, et les résultats ont tous été négatifs [
32
]. Ce genre de fausse information décrédibilise donc les narratifs sur l’accident de laboratoire, qui ne reposaient déjà sur aucune preuve.
|
La lutte de deux narratifs
Le contexte médiatique actuel quant à l’origine du SARS-CoV-2, se résume, pour l’essentiel, à la lutte entre deux narratifs qui, l’un comme l’autre, ne repose sur aucun élément concret, ni sur aucune preuve scientifique, médicale ou matérielle. Il s’agit donc d’une joute oratoire, une lutte d’idées. Le concept du débordement (
Spillover
) a été initialement développé par Daszak
et al.
[
33
], non pas pour expliquer l’origine d’une zoonose, mais pour caractériser la propagation de maladies infectieuses dans la faune sauvage, à partir d’animaux domestiques. Le mécanisme inverse était qualifié de «
Spillback
». Power et Mitchell [
16
] ont donné au terme son sens actuel de contamination de la population humaine à partir d’une épizootie (épidémie chez une espèce animale). Depuis, le sens de «
Spillover
» a été altéré pour le rendre finalement totalement synonyme de « contamination » [
34
]. Pour ces auteurs, quel que soit le mode de contamination, il s’agit d’un «
Spillover
», d’un débordement de l’animal à l’homme. Ce glissement sémantique vient d’une incompréhension majeure présentant les microorganismes comme des éléments passifs, parfois qualifiés de simples « propagules »
2
, dans l’action de transmission. Pourtant, ce sont en fait les microorganismes qui représentent la partie active du processus de transmission. Ils ont, au cours de l’évolution, développé des mécanismes actifs pour infecter les hôtes, tromper leurs défenses et même les manipuler pour assurer leur transmission. L’exemple de manipulation le plus connu est celui du virus de la rage qui génère chez l’hôte infecté (en l’occurrence le chien ou le renard) un comportement agressif, permettant la transmission du virus par la salive lors d’une morsure. Les prérequis du modèle «
Spillover
», c’est-à-dire la présence du virus humain (avec une séquence identique) dans une espèce réservoir et une espèce intermédiaire, n’ont jamais été démontrés, que ce soit pour le SARS-CoV-2, le SARS-CoV, le MERS-CoV (coronavirus du syndrome respiratoire du Moyen-Orient), des souches d’HCoV (coronavirus humains), HCoV-HKU1 et HCoV-NL63, le virus de l’hépatite C, le virus de la poliomyélite ou le virus Ebola [
35
]. Cette opposition de deux constructions intellectuelles, «
Spillover
» et fuite de laboratoire, ne peut ainsi en aucun cas permettre la compréhension de l’émergence de maladies infectieuses.
|
Humanisation et socialisation
Avant d’aborder l’explication de l’émergence du SARS-CoV-2, il est important de considérer la question de l’évolution et de l’adaptation d’un virus chez son hôte. Tous les virus évoluent et s’adaptent, et les virus à ARN sont particulièrement efficaces pour ce faire. Les virus multi-hôtes, qui infectent des hôtes différents, évoluent en s’adaptant en permanence. Lorsqu’ils s’installent chez l’homme, cela conduit à un processus qualifié d’ « humanisation ». Chez une autre espèce, le virus évoluera différemment. Le virus s’adapte en permanence à son hôte humain, en affinant l’adéquation de ses protéines à celles de son hôte et en surmontant ses défenses. Cela conduit à la génération de nouvelles lignées virales qui remplacent les précédentes. Ce processus a conduit, avec le SARS-CoV-2, à l’apparition de nouvelles lignées plus adaptées, les variants Beta, Delta, Omicron, etc. C’est un processus continu qui ne s’arrêtera pas. Le gain par évolution de mutations qui modifient, par exemple, le site de clivage de la protéine Spike par la furine, une enzyme présente à la membrane des cellules de l’hôte, nécessaire à l’entrée du virus, ou la substitution D614G (l’aspartate [D] en position 614 remplacé par une glycine [G]) dans la séquence de Spike, qui affecte et semble accroître son affinité pour ACE2, était essentiel pour l’augmentation de la transmissibilité et l’émergence du SARS-CoV-2 [
36
,
37
]. Les mutations des nouveaux variants jouent le même rôle. Par définition, faute de pouvoir se multiplier seuls, les virus utilisent leurs hôtes pour se propager. Ils profitent, en particulier, de l’écologie et du comportement de leurs hôtes pour se disséminer par des contacts intra- ou inter-espèces. Le même processus se produit avec le SARS-CoV-2 chez l’homme. Cependant, les sociétés humaines sont régies par des règles sociétales spécifiques. Les moteurs de la dissémination du virus à l’intérieur ou à l’extérieur de la population humaine sont donc majoritairement sociétaux. Il est essentiel de comprendre, qu’en conséquence, le SARS-CoV-2 s’est
socialisé
en même temps qu’il s’est humanisé, et que son expansion est régie par nos règles sociétales.
|
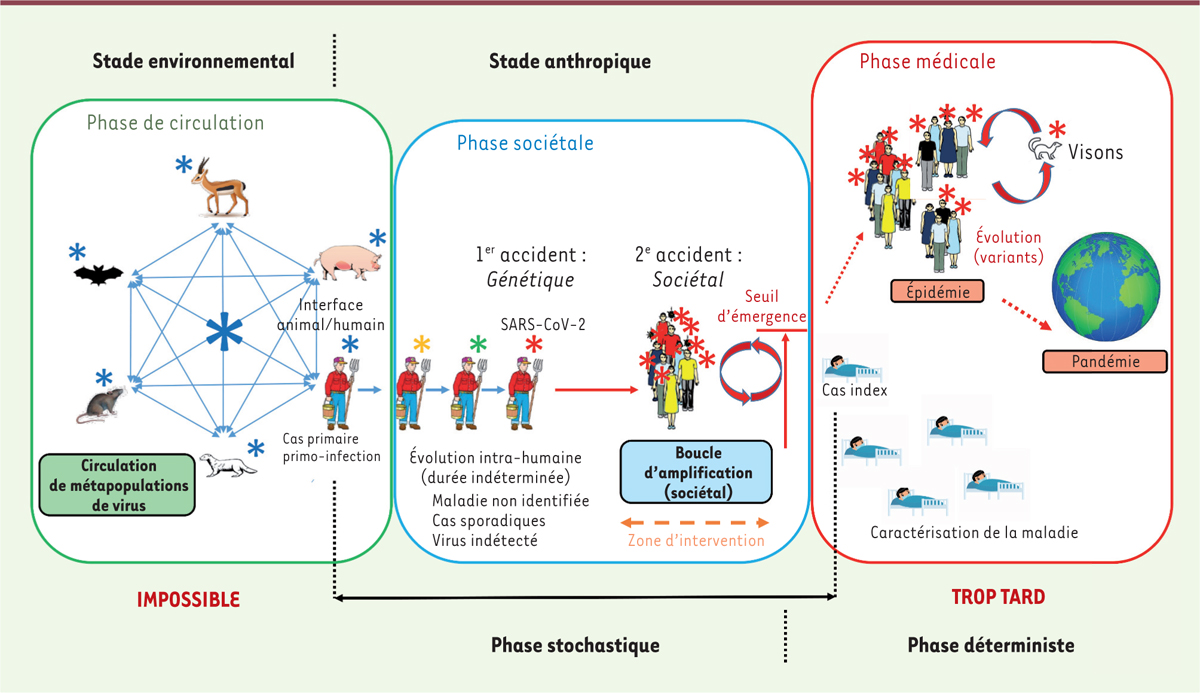
De l’origine du SARS-CoV-2 par sélection naturelle, ou la préservation des variants privilégiés dans la lutte pour sa survie
Le processus d’émergence d’une pandémie peut être divisé en trois phases : une phase de circulation, une phase sociétale et une phase médicale
(
Figure 1
)
. Les phases « sociétale » et « médicale » forment ensemble un stade anthropique (dû à l’homme). La phase de circulation correspond au stade environnemental et est caractérisée par une circulation multidirectionnelle permanente de virus multi-hôtes, simplement par contact. Les hommes font partie de ce vaste processus de circulation, acquérant des virus d’autres espèces et transmettant des virus à d’autres espèces. Une circulation de virus ne signifie absolument pas qu’il y ait des épidémies. Il ne faut surtout pas confondre infection et maladie. Cette circulation se fait à bas bruit. Ainsi, l’étude des viromes
3
chez des sujets sains, y compris humains, montre la présence de nombreux virus sans qu’il y ait eu d’épidémie [
38
]. Les lieux les plus adéquats pour que les hommes acquièrent des virus multi-hôtes sont les environnements ruraux anthropisés
4
[
39
,
40
], en particulier les fronts pionniers ou fronts de peuplement. Ces habitats ruraux fournissent nourriture et abri à divers animaux et offrent des possibilités de contacts et de transmission de virus qui n’existent pas dans la nature [
39
–
41
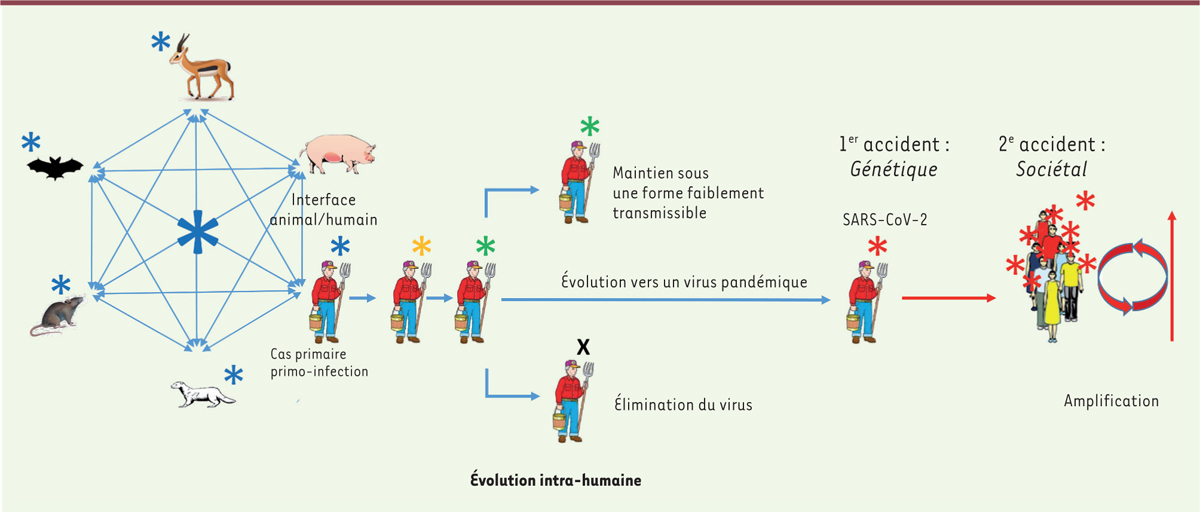
]. Une fois la primo-infection établie, le virus entre dans la phase sociétale : son évolution et sa transmission ont maintenant lieu au sein de la population humaine ; le virus évolue alors à bas bruit au sein de cette population suivant le processus de « quasi-espèce ». Cependant, cette phase est stochastique et de nombreux événements peuvent se produire, le virus pouvant être éliminé, rester sous une forme à faible transmissibilité, ou encore muter en une forme à haute transmissibilité
(
Figure 2
)
. C’est ce dernier cas de figure qui s’est réalisé avec le SARS-CoV-2. Il n’est pas possible de déterminer combien de temps ce processus peut durer, mais, dans le cas du SARS-CoV-2, il a laissé des traces de sélection positive, c’est-à-dire de sélection dirigée par l’hôte, dans le génome viral [
42
,
43
]. L’évolution vers une forme hautement transmissible correspond à un premier accident,
génétique
. Un deuxième accident,
sociétal
, doit alors se produire pour assurer l’amplification de la population virale jusqu’au niveau du seuil d’émergence. Une épidémie ne commence jamais avec un seul individu infecté ; c’est un processus probabiliste, c’est-à-dire qu’il faut qu’il y ait un nombre minimal d’individus infectés pour déclencher une épidémie : c’est ce qui est appelé seuil d’émergence [
44
]. Cela nécessite une forte densité humaine dans une zone limitée pendant un temps limité et dans des conditions permettant la transmission du virus entre individus. Cela correspond à une boucle d’amplification, un processus permettant d’accroître rapidement la population virale jusqu’au niveau du seuil d’émergence. Un
cluster
est la représentation statique et la boucle d’amplification, la représentation dynamique du même processus. Cela correspond à des événements sociétaux et explique pourquoi les maladies pandémiques émergent dans les villes. La probabilité qu’un événement sociétal à forte densité de population se produise est en effet significativement plus élevée en milieu urbain qu’en milieu rural. Les événements sociétaux conduisant à l’amplification de la population virale sont divers et peuvent dépendre du type de société. Une fois le seuil d’émergence atteint, le virus entre dans la phase déterministe qui caractérise une épidémie. À ce stade, le processus entre dans la dernière phase, la phase médicale. La maladie n’est pas encore identifiée lorsque la population virale atteint le seuil d’émergence. Il y a un retard dû au fait qu’une maladie émergente est par définition inconnue et que le syndrome spécifique n’est pas caractérisé. Les médecins ne détectent la survenue d’une maladie inconnue que lorsqu’ils sont confrontés à un flux de patients souffrant du même syndrome inconnu. Le cas index ainsi que l’agent causal peuvent alors être identifiés à ce stade, permettant ainsi le développement de tests de détection et de dépistage dans la population. Or, le temps de franchir toutes ces étapes, le virus s’est déjà largement propagé dans la population, déclenchant une épidémie, qui peut, du fait de la forte mobilité internationale qui caractérise notre époque, se transformer rapidement en pandémie. Tout ce processus correspond à ce qui a été décrit comme le modèle
circulation
[
6
,
19
,
45
]. Ce modèle circulation a été récemment confirmé pour le virus Ebola [
46
]. En effet, le virus responsable de l’épidémie d’Ebola de 2020-2021 en Guinée circulait au sein de la population humaine depuis plusieurs années sans provoquer d’épidémie et sans qu’aucune propagation depuis un hôte sauvage, c’est à dire un «
Spillover
», ne se soit produite [
46
].
 | Figure 1.
Représentation schématique du processus de circulation et d’émergence de virus pandémiques dans la population humaine
(figure adaptée de [
6
]).
|
 | Figure 2.
Différentes voies d’évolution d’un virus à ARN dans une population humaine
(figure adaptée de [
6
]).
|
Le 24 novembre 1859, Charles Darwin a publié un ouvrage majeur intitulé «
De l’origine des espèces par sélection naturelle ou la préservation des races favorisées dans la lutte pour la vie
». Il faudrait de nos jours interpréter le terme « race » dans le titre original de l’œuvre de Darwin comme signifiant « populations », ce qui correspond d’ailleurs au sens donné par Darwin au mot « race » au
xix
e
siècle. Dans cet ouvrage fondateur, Darwin démontre qu’il n’y a pas « d’origine » déterminée aux chats, aux rorquals bleus, aux éléphants, aux humains, aux kangourous ou aux pins d’Alep. Il existe un processus permanent d’évolution, d’adaptation et de sélection dans lequel le hasard et l’environnement jouent un rôle majeur, processus qui conduit au développement d’espèces adaptées. De même, il n’y a pas « d’origine » déterminée pour le SARS-CoV-2 ou d’autres virus, mais un processus permanent d’évolution, d’adaptation et de sélection façonné par le hasard et l’environnement, qui donne naissance à de nouvelles lignées, c’est-à-dire des variants. Un virus se comporte de la même manière avant et après émergence : il se diffuse par contact et évolue sous la pression de l’hôte sous forme de variants successifs comme le montre le SARS-CoV-2.
|
Perception et compréhension Charles Darwin nous a donné les clefs il y a 163 ans, mais il est encore extrêmement difficile pour beaucoup d’accepter que l’émergence d’une maladie ne soit que le résultat de l’évolution, d’événements stochastiques, de la société humaine et du comportement humain. Les populations veulent des réponses à la question sur « l’origine » de toute maladie infectieuse et plus précisément « quand », « qui », « d’où » et « pourquoi ». Ces questions n’ont en fait pas de réponse parce qu’elles n’ont pas de réalité. La seule question qui existe véritablement et à laquelle une réponse peut être apportée est « comment », c’est-à-dire, quel est le processus conduisant à l’émergence d’une maladie infectieuse. Ce processus est simplement une évolution, une adaptation et une sélection comme pour tout autre organisme vivant sur Terre. Ce sont les paroles de sagesse que Darwin nous a apportées en 1859. Pourquoi ne sont-elles pas comprises aujourd’hui ? Un premier facteur est l’ignorance. Beaucoup de gens, y compris dans les milieux médicaux et scientifiques, ne sont pas familiers avec les concepts et mécanismes de l’évolution et imaginent un processus de transmission simple et linéaire avec une origine, éventuellement un intermédiaire et un destinataire final. Ce modèle linéaire est très simple et attractif mais, hélas, faux car l’émergence d’une maladie est le résultat de l’évolution et de la survenue d’événements stochastiques. Il ne faut pas non plus négliger les attitudes corporatistes conduisant à favoriser certaines théories dans un but de profit ou de pouvoir, ni la manipulation à des fins politiques, idéologiques ou commerciales. Il est essentiel de ne fonder des conclusions que sur des preuves scientifiques et de s’éloigner des opinions et des narratifs. |
Un plaidoyer pour une approche rationnelle D’autres pandémies surviendront dans le futur, ce n’est qu’une question de temps. La probabilité d’occurrence étant directement liée au nombre de contacts et aux opportunités d’amplification, la croissance permanente de la population humaine rend cette probabilité plus élevée chaque année. La question essentielle est donc : peut-on prévenir une autre pandémie ? Syndromique par essence, la médecine arrive trop tard dans le processus et ne peut empêcher ni l’émergence d’une pandémie, ni sa propagation, et son objectif reste de soigner les malades. Le dépistage des virus dans la nature et la surveillance ne peuvent rien non plus puisque, par définition, il est impossible de détecter et surveiller ce qui n’est pas encore connu et les virus pandémiques évoluant chez l’homme ne préexistent pas dans la nature. Il est également impossible de dire quel virus parmi la multitude en circulation pourrait un jour infecter les hommes et, pire encore, lequel pourrait donner lieu à une pandémie. Si nous avions détecté et séquencé le virus SARS-CoV-2, en 2018 par exemple, personne n’aurait été capable de déterminer son potentiel pandémique, cela aurait juste été une séquence anonyme parmi une multitude d’autres. La solution n’est pas de cibler la maladie ou l’agent causal putatif mais plutôt de cibler le processus d’émergence de la maladie lui-même. Le processus d’émergence de la maladie étant d’origine sociale, la seule approche fiable pour prévenir une émergence est une approche sociétale. Il faut organiser les activités de façon à éviter les boucles d’amplification et empêcher le virus d’atteindre le seuil d’émergence. Quel que soit le virus, il devra passer par ces boucles d’amplification. C’est l’effort que l’humanité doit consentir. Notre société a su s’organiser pour faire face à d’autres menaces et dangers majeurs, comme le nucléaire ou le changement climatique. Pourquoi ne pourrait-elle pas en faire de même pour les pandémies ? |
Les auteurs déclarent n’avoir aucun lien d’intérêt concernant les données publiées dans cet article.
|
Footnotes |
1.
Zhu
N
,
Zhang
D
,
Wang
W
,
et al.
A Novel Coronavirus from Patients with Pneumonia in China, 2019.
.
N Engl J Med.
2019;
;
382
:
:727.
–
733
.
2.
Guan
W
,
Ni
Z
,
Hu
Y
,
et al.
Characteristics Clinical of Coronavirus Disease 2019 in China.
.
New. N Engl J Med.
2020;
;
58
:
:711.
–
712
.
3.
Huang
C
,
Wang
Y
,
Li
X
,
et al.
.
Clinical features of patients infected with 2019 novel coronavirus in Wuhan, China.
.
Lancet.
2022;
;
395
:
:497.
–
506
.
5.
Frutos
R
,
Lopez-Roig
M
,
Serra-Cobo
J
,
Devaux
C.A.
COVID-19: The conjunction of events leading to the pandemic and lessons to learn for future threats.
.
Front Med.
2020;
:
7
:
:223.
.
6.
Frutos
R
,
Pliez
O
,
Gavotte
L
,
Devaux
CA
.
There is no “origin” to SARS-CoV-2.
.
Environ Res.
2022;
;
207
:
:112173.
.
7.
Li
Q
,
Guan
X
,
Wu
P
,
et al.
Early transmission dynamics in Wuhan, China, of novel coronavirus-infected pneumonia.
.
N Engl J Med.
2020;
;
382
:
:1199.
–
207
.
8.
Lai
A
.
Early phylogenetic estimate of the effective reproduction number of SARS-CoV-2.
.
J Med Virol.
2020;
;
92
:
:675.
–
9
.
9.
Cao
Y
,
Li
L
,
Feng
Z
,
et al.
Comparative genetic analysis of the novel coronavirus (2019-nCoV/SARS-CoV-2) receptor ACE2 in different populations.
.
Cell Discov.
2020;
;
6
:
:1.
–
4
.
11.
Coelho
MTP
,
Rodrigues
JFM
,
Medina
AM
,
et al.
Global expansion of COVID-19 pandemic is driven by population size and airport connections.
.
PeerJ.
2020;
;
8
:
:e9708.
.
https://doi.org/10.7717/peerj.9708
.
12.
Andino
R
,
Domingo
E
.
Viral quasispecies.
.
Virology.
2015;
;
479
:
:46.
–
51
.
13.
Song
HD
,
Tu
CC
,
Zhang
GW
,
et al.
Cross-host evolution of severe acute respiratory syndrome coronavirus in palm civet and human.
.
Proc Natl Acad Sci U S A.
2005;
;
102
:
:2430.
–
2435
.
14.
Karamitros
T
,
Papadopoulou
G
,
Bousali
M
,
et al.
SARS-CoV-2 exhibits intra-host genomic plasticity and low-frequency polymorphic quasispecies.
.
J Clin Virol.
2020;
;
131
:
:104585.
.
15.
Woolhouse
ME
,
Adair
K
,
Brierley
L
.
RNA viruses: a case study of the biology of emerging infectious diseases.
.
Microbiol Spectr.
2013;
;
1
:
:10.1128.
.
16.
Power
AG
,
Mitchell
CE
.
Pathogen spillover in disease epidemics.
.
Am Nat.
2004;
;
164
:
:S79.
–
S89
.
17.
Boni
MF
,
Lemey
P
,
Jiang
X
,
et al.
Evolutionary origins of the SARS-CoV-2 sarbecovirus lineage responsible for the COVID-19 pandemic.
.
Nat Microbiol.
2020;
;
5
:
:1408.
–
17
.
18.
Burki
T
.
The origin of SARS-CoV-2.
.
Lancet Infect Dis.
2020;
;
20
:
:1018.
–
9
.
19.
Frutos
R
,
Serra-Cobo
J
,
Chen
T
,
Devaux
CA
.
COVID-19: Time to exonerate the pangolin from the transmission of SARS-CoV-2 to humans.
.
Infect Genet Evol.
2020;
;
84
:
:104493.
.
20.
Lee
J
,
Hughes
T
,
Lee
MH
,
et al.
No evidence of coronaviruses or other potentially zoonotic viruses in Sunda pangolins (Manis javanica) entering the wildlife trade via Malaysia.
.
Ecohealth.
2020;
;
17
:
:406.
–
18
.
21.
Tang
X
,
Wu
C
,
Li
X
,
et al.
On the origin and continuing evolution of SARS-CoV-2.
.
Natl Sci Rev.
2020;
;
7
:
:1012.
–
23
.
22.
Munnink
BBO
,
Sikkema
RS
,
Nieuwenhuijse
DF
,
et al.
Transmission of SARS-CoV-2 on mink farms between humans and mink and back to humans.
.
Science.
2021;
;
371
:
:172.
–
7
.
23.
Ge
XY
,
Wang
N
,
Zhang
W
,
et al.
Coexistence of multiple coronaviruses in several bat colonies in an abandoned mineshaft.
.
Virol Sin.
2016;
;
31
:
:31.
–
40
.
24.
Zhou
P
,
Yang
XL
,
Wang
XG
,
et al.
A pneumonia outbreak associated with a new coronavirus of probable bat origin.
.
Nature.
2020;
;
579
:
:270.
–
3
.
25.
Zhou
H
,
Chen
X
,
Hu
T
,
et al.
A novel bat coronavirus closely related to SARS-CoV-2 contains natural insertions at the S1/S2 cleavage site of the spike protein.
.
Curr Biol.
2021;
;
30
:
:2196.
–
203.e3
.
26.
Delaune
D
,
Hul
V
,
Karlsson
EA
,
Hassanin
A
,
et al.
A novel SARS-CoV-2 related coronavirus in bats from Cambodia.
.
Nat Commun.
2021;
;
12
:
:6563.
.
27.
Wacharapluesadee
S
,
Tan
CW
,
Maneeorn
P
,
et al.
Evidence for SARS-CoV-2 related coronaviruses circulating in bats and pangolins in Southeast Asia.
.
Nat Commun.
2021;
;
12
:
:972.
.
28.
Domingo
JL
.
What we know and what we need to know about the origin of SARS-CoV-2.
.
Environ Res.
2021;
;
200
:
:111785.
.
29.
Rahalkar
MC
,
Bahulikar
RA
.
Lethal pneumonia cases in Mojiang miners (2012) and the mineshaft could provide important clues to the origin of SARS-CoV-2.
.
Front Public Health.
2020;
;
8
:
:581569.
.
30.
Menachery
VD
,
Yount
BL
,
Debbink
K
,
et al.
A SARS-like cluster of circulating bat coronaviruses shows potential for human emergence.
.
Nat Med.
2015;
;
21
:
:1508.
–
1513
.
31.
Frutos
R
,
Javelle
E
,
Barberot
C
,
et al.
Origin of COVID-19: Dismissing the Mojiang mine theory and the laboratory accident narrative.
.
Environ Res.
2022;
;
204
:
:112141.
.
32.
Zhou
P
,
Yang
XL
,
Wang
XG
,
et al.
Addendum: a pneumonia outbreak associated with a new coronavirus of probable bat origin.
.
Nature.
2020;
;
588
:
:E6.
.
33.
Daszak
P
,
Cunningham
AA
,
Hyatt
AD
.
Emerging infectious diseases of wildlife–threats to biodiversity and human health.
.
Science.
2020;
;
287
:
:443.
–
9
.
34.
Plowright
RK
,
Parrish
CR
,
McCallum
H
,
et al.
Pathways to zoonotic spillover.
.
Nat Rev Microbiol.
2017;
;
15
:
:502.
–
510
.
35.
Holmes
EC
,
Goldstein
SA
,
Rasmussen
AL
,
et al.
The origins of SARS-CoV-2: A critical review.
.
Cell.
2021;
;
184
:
:4848.
–
56
.
36.
Xia
S
,
Lan
Q
,
Su
S
,
et al.
The role of furin cleavage site in SARS-CoV-2 spike protein-mediated membrane fusion in the presence or absence of trypsin.
.
Signal Transduct Target Ther.
2020;
;
5
:
:92.
.
37.
Zhou
B
,
Thao
TTN
,
Hoffmann
D
,
et al.
SARS-CoV-2 spike D614G change enhances replication and transmission.
.
Nature.
2021;
;
592
:
:122.
–
7
.
38.
Kumata
R
,
Ito
J
,
Takahashi
K
,
et al.
A tissue level atlas of the healthy human virome.
.
BMC Biol.
2020;
;
18
:
:55.
.
39.
Afelt
A
,
Lacroix
A
,
Zawadzka-Pawlewska
U
,
et al.
Distribution of bat-borne viruses and environment patterns.
.
Infect Genet Evol.
2017;
;
58
:
:181.
–
191
.
40.
Afelt
A
,
Frutos
R
,
Devaux
C
.
Bats, coronaviruses and deforestation: towards the emergence of novel infectious diseases?
.
Front Microbiol.
2018;
;
9
:
:702.
.
41.
Plowright
RK
,
Eby
P
,
Hudson
PJ
,
et al.
Ecological dynamics of emerging bat virus spillover.
.
Proc Biol Sci.
2015;
;
282
:
:20142124.
.
42.
MacLean
OA
,
Lytras
S
,
Weaver
S
,
et al.
Natural selection in the evolution of SARS-CoV-2 in bats created a generalist virus and highly capable human pathogen.
.
PLoS Biol.
2021;
;
19
:
:e3001115.
.
43.
Frutos
R
,
Gavotte
L
,
Devaux
CA
.
Unravelling the origin of SARS-CoV-2: is the model good?
New Microbes New Infect.
2021;
;
43
:
:100918.
.
44.
Hartfield
M
,
Alizon
S
.
Introducing the outbreak threshold in epidemiology.
.
PLoS Pathog.
2013;
;
9
:
:e1003277.
.
45.
Frutos
R
,
Gavotte
L
,
Devaux
CA
.
Understanding the origin of COVID-19 requires to change the paradigm on zoonotic emergence from the spillover to the circulation model.
.
Infect. Genet Evol.
2021;
;
95
:
:104812.
.
46.
Keita
AK
,
Koundouno
FR
,
Faye
M
,
et al.
Resurgence of Ebola virus in 2021 in Guinea suggests a new paradigm for outbreaks.
.
Nature.
2021;
;
597
:
:539.
–
43
.
|