| |
| Med Sci (Paris). 38(6-7): 532–536. doi: 10.1051/medsci/2022070.Une sous-population de macrophages hépatiques impliquée dans la régulation du métabolisme Camille Blériot1,2* and Florent Ginhoux1,2,3,4** 1Inserm U1015, Institut Gustave Roussy, bâtiment de médecine moléculaire
,
114 rue Édouard Vaillant
,
94800Villejuif
,
France 2
Singapore Immunology Network (SIgN), Agency for Science, Technology and Research (A
*
STAR)
,
8A Biomedical Grove, Immunos Building #3-4
, Biopolis,
Singapore138648
,
Singapore 3Shanghai Institute of Immunology, Shanghai JiaoTong University School of Medicine
,
280 South Chongqing Road
,
Shanghai200025
,
Chine 4Translational Immunology Institute, SingHealth Duke-NUS Academic Medical Centre
,
Singapore169856
,
Singapore MeSH keywords: Humains, Foie, Macrophages, métabolisme |
Les macrophages résidents constituent une population hétérogène de cellules immunitaires caractérisées par des identités spécifiques aux tissus dans lesquels elles résident et présentant un large éventail de fonctions s’étendant bien au-delà du système immunitaire [
1
]. Par exemple, les macrophages du cerveau, appelés microglie, sont impliqués dans la transmission neuronale en façonnant les terminaisons synaptiques en fonction des stimulations externes [
2
].
Le foie héberge une population de macrophages résidents, appelés cellules de Kupffer (KC), qui tapissent les sinusoïdes hépatiques et sont notamment impliqués dans la détoxification du sang provenant de l’intestin
via
la veine porte et qui contient des sous-produits toxiques de la digestion et parfois même des agents pathogènes [
3
]. Comme la plupart des macrophages résidents, les KC dérivent de précurseurs ayant colonisé le foie pendant le développement embryonnaire, bien avant la naissance et la mise en place de l’hématopoïèse définitive dans la moelle osseuse [
1
]. Ainsi, contrairement à ce qui a été longtemps considéré comme un fait acquis, les KC sont indépendantes des monocytes circulant dans le sang et se renouvellent de manière autonome dans le foie par un mécanisme de lent auto-renouvèlement. Bien que les KC constituent la population de macrophages la plus abondante dans le foie, d’autres populations, comme les macrophages capsulaires, ont également été identifiées, mais celles-ci ont une localisation, des fonctions et des origines complètement différentes [
3
].
Au sein des KC, l’existence de sous-populations distinctes a déjà été proposée [
4
,
5
] mais cette hypothèse s’est bien souvent heurtée à des difficultés pour discerner de façon concluante ces différentes populations de macrophages hépatiques, celles-ci possédant des marqueurs de surface très similaires. En effet, rien ne ressemble plus à un macrophage qu’un autre macrophage et l’utilisation de marqueurs tels que F4/80 chez la souris ou CD68 chez l’homme se révèle trop limitée pour appréhender l’hétérogénéité de ces cellules. Dans l’étude que nous présentons [
6
], nous avons combiné des approches à haut débit sur cellules uniques, pour profiler les KC, avec des modèles murins spécifiques, permettant d’identifier génétiquement très précisément les KC. Nous avons identifié deux sous-populations majeures de KC qui diffèrent notamment par leur expression du marqueur CD206, le récepteur du mannose des macrophages (MMR ou Mrc1,
mannose receptor C-type
1), ainsi que par l’expression d’une signature métabolique avec notamment le transporteur d’acides gras CD36.
|
Une approche à haut débit non biaisée sur cellules uniques pour révéler l’hétérogénéité des cellules de Kupffer
Afin de cartographier précisément les KC, nous avons tout d’abord utilisé des approches à haut débit, comme le séquençage d’ARN sur cellules uniques et la cytométrie de masse (CyTOF). L’analyse bio-informatique des données ainsi obtenues permet d’obtenir une résolution inégalée et d’esquisser une cartographie précise des cellules présentes dans un organe donné. En restreignant l’analyse aux cellules immunitaires qui expriment toutes la molécule CD45, nous avons ainsi pu identifier les différentes populations : les lymphocytes B ou les lymphocytes T, les cellules dendritiques, les monocytes et, bien entendu, les macrophages. Nous avons ensuite centré l’analyse sur les KC qui expriment de manière spécifique des gènes/protéines, comme CLEC4F (
C-type lectin domain family 4 member F
) et TIM4 (
T-cell membrane protein 4
) et avons observé que cette population pouvait être scindée en deux sous-populations, différant notamment par l’expression de CD206. La mise en évidence de ce marqueur par cette approche non biaisée nous a ensuite permis de l’utiliser pour étudier les deux sous-populations par des approches plus conventionnelles comme la cytométrie en flux
(
Figure 1A
)
.
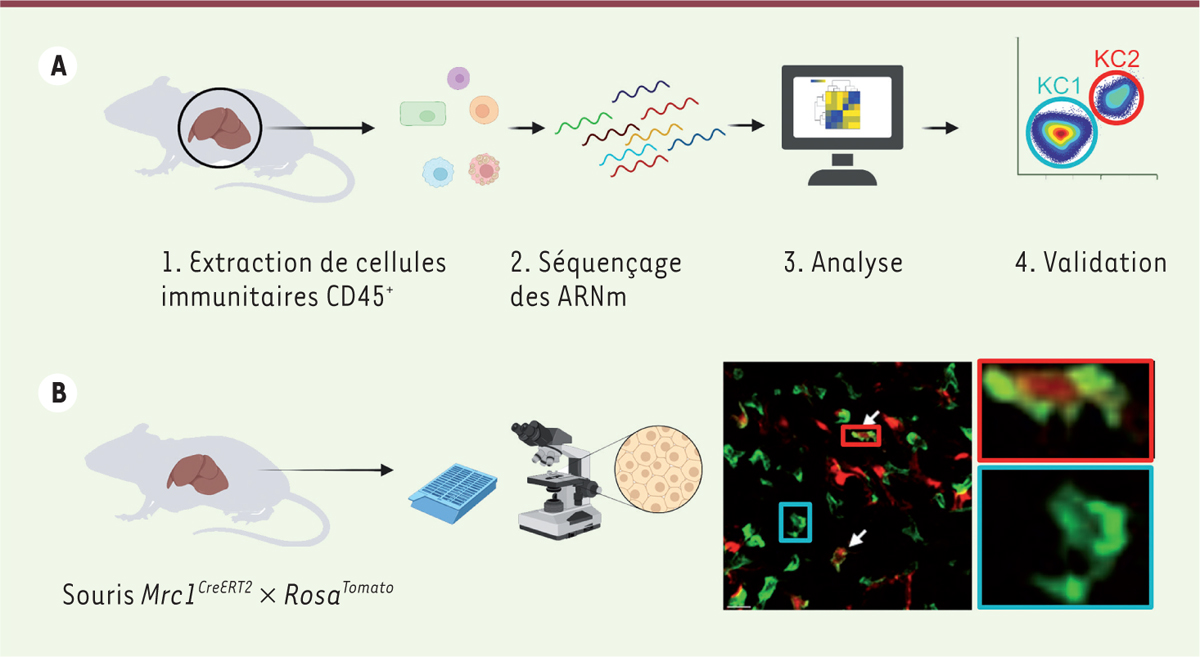
 | Figure 1.
Hétérogénéité des cellules de Kupffer (KC). A.
Analyse, chez la souris, du foie par séquençage d’ARN sur cellules uniques et validation par cytométrie en flux. Nous avons utilisé deux approches complémentaires. Tous les leucocytes (CD45
+
) ont été extraits de foies sains puis analysés en utilisant la technique Chromium 10X. Cette analyse sur un grand nombre de cellules, mais avec une profondeur de séquençage limitée, permet de dresser une cartographie des cellules immunitaires du foie. Nous avons également trié les KC puis les avons séquencées à haute résolution grâce au protocole SMARTseq2, ce qui a permis de révéler les deux sous-populations. Nous avons ensuite intégré ces deux jeux de données et les avons validés en utilisant la cytométrie en flux.
B.
Observation en microscopie à fluorescence de lames de foies fixés provenant de souris
Mrc1
CreERT2
x
Rosa
Tomato
. Dans ce modèle, les cellules qui expriment la protéine CD206 codée par le gène
Mrc1
sont marquées en rouge par la protéine fluorescente Tomato. Les macrophages sont marqués par un anticorps anti-F4/80 en vert. Cela permet de visualiser
in situ
les deux sous-populations de KC : KC1 CD206
-
F4/80
+
en vert, et KC2 CD206
+
F4/80
+
en vert et rouge. Les KC2 (flèche blanche) représentent 10 à 15 % des cellules totales.
|
|
Validation de l’approche non biaisée par des outils conventionnels
Bien que l’approche à haut débit que nous avons utilisée autorise un profilage très efficace d’un échantillon donné, les hypothèses qu’elles permet d’avancer doivent être confirmées. Pour cela, nous avons utilisé les biomarqueurs mis en évidence dans cette première étape, comme CD206 et ESAM (
endothelial cell-selective adhesion molecule
), et ainsi pu valider la présence de deux sous-populations cellulaires au sein des KC : les cellules ESAM
-
CD206
-
ou KC1 et les cellules ESAM
+
CD206
+
ou KC2. Nous avons par ailleurs développé un nouveau modèle murin fondé sur l’expression spécifique de CD206 par les KC2. Les souris
Mrc1
CreERT2
x
Rosa
Tomato
permettent le marquage des cellules exprimant le gène
Mrc1
codant CD206
in vivo.
Dans ce modèle, nous avons ainsi observé que certaines KC, correspondant aux cellules KC2 que nous avions identifiées, étaient spécifiquement marquées
(
Figure 1B
)
.
De nombreuses études ont établi que la plupart des macrophages résidents, dont les KC, dérivent de précurseurs myeloïdes qui colonisent les organes durant le développement embryonnaire et sont, de ce fait, indépendants des monocytes circulants adultes [
1
]. Nous avons développé un modèle murin fondé sur l’expression du gène
Ms4a3
(
membrane spanning 4-domains A3
) qui est spécifiquement exprimé par les précurseurs de monocytes de la moelle osseuse adulte, ou GMP (
granulocyte-monocyte precursors
). Ce modèle
Ms4a3
Cre
x
Rosa
Tomato
permet de différencier très précisément les macrophages embryonnaires des macrophages dérivant de monocytes [
6
].
Nous avons donc utilisé ce modèle de souris et observé que les deux sous-populations de KC étaient marquées avec la même efficacité, certes très faiblement, car les KC dérivent très majoritairement de précurseurs embryonnaires, mais ce marquage confirmait leur origine commune. Nous avons également utilisé de nombreux autres modèles murins rapporteurs, connus pour marquer sélectivement les macrophages, comme, par exemple, les modèles
Csf1r
GFP
et
Mafb
GFP
1
. Dans ces modèles, les deux sous-populations, KC1 et KC2, étaient marquées avec une efficacité similaire, attestant que ces deux populations appartiennent bien à une même lignée de macrophages.
|
Établissement de signatures propres aux deux sous-populations de KC
Nous avons ensuite utilisé ces marqueurs pour trier ces sous-populations et approfondir leur analyse. Comme attendu, les deux sous-populations expriment à un niveau comparable les gènes caractéristiques des KC comme
Clec4f
,
Lyz2
(
lysozyme 2)
2
ou
Vsig4
(
V-set and immunoglobulin domain-containing 4
). Nous avons également généré des données de « translatomique », grâce à la stratégie « RiboTag ». Cette approche s’appuie sur le modèle murin
Lyz2
Cre
x
Rpl22
HA
(
ribosomal protein L22
), qui permet d’isoler, par immunoprécipitation, les ribosomes des cellules exprimant
Lyz2
. Seuls les ARN associés à ces ribosomes sont ensuite séquencés, ce qui permet d’exclure les ARN éventuellement ingérés par les macrophages ayant phagocyté d’autres cellules. Enfin, nous avons déterminé le protéome des deux sous-populations de KC par spectrométrie de masse. Nous avons ainsi pu générer une signature multi-omique robuste pour les deux sous-populations
(
Figure 2A
)
.
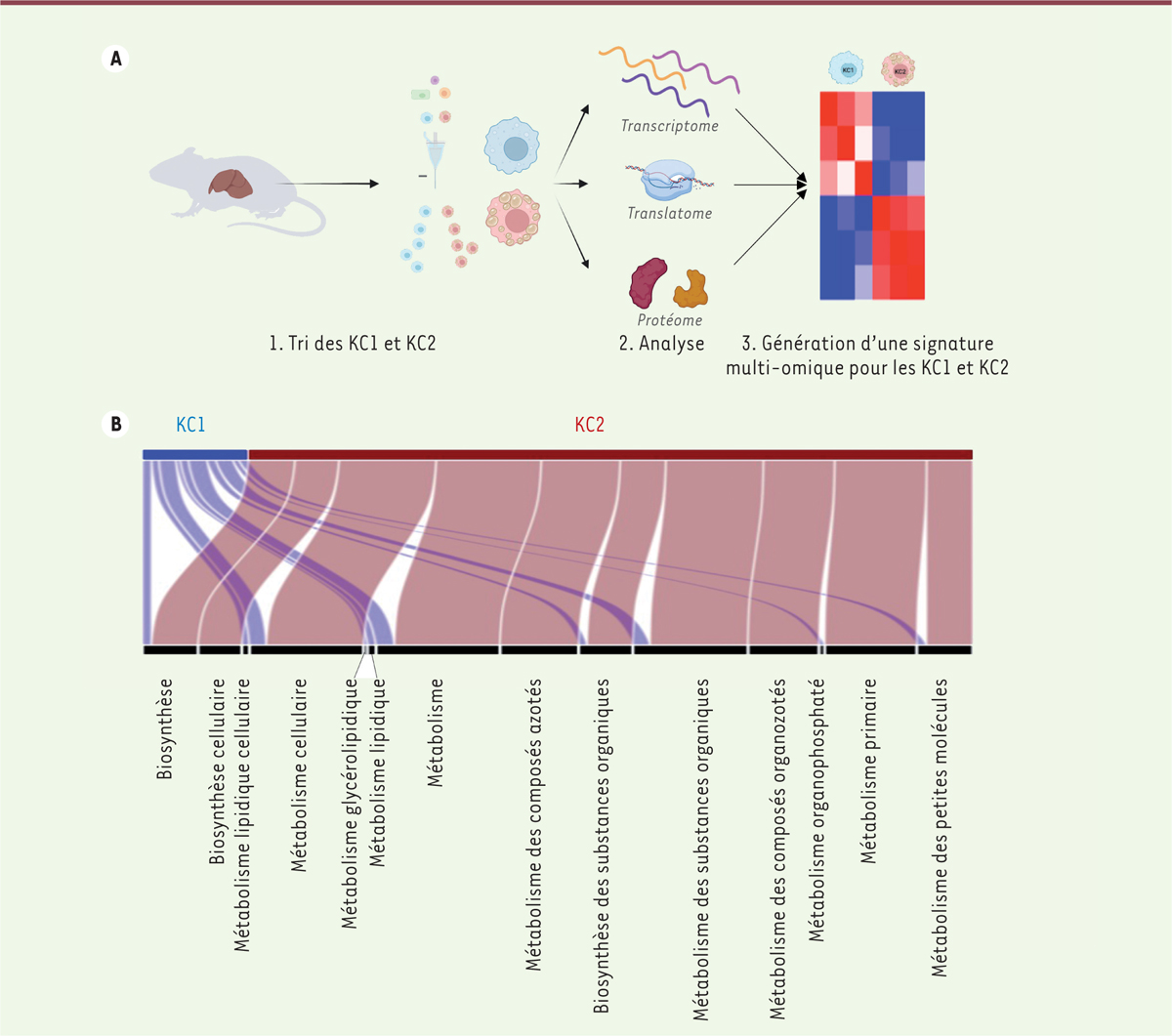
 | Figure 2.
La sous-population CD206
+
ESAM
+
KC présente une signature métabolique caractéristique. A.
Analyse des transcriptomes, translatomes et protéomes des KC1 et KC2 triées, et génération d’une signature multi-omique mettant en évidence les fonctions métaboliques des KC2.
B.
Voies de signalisation métabolique exprimées dans les deux sous-populations de KC. L’épaisseur des traits est proportionnelle au nombre de gènes impliqués.
|
Au-delà de la signature « KC-macrophage » partagée par les deux sous-populations, l’analyse de ces signatures a en particulier révélé la prédominance de fonctions métaboliques pour la population KC2
(
Figure 2B
)
, avec, notamment, une expression accrue du gène
Cd36
qui code un transporteur d’acides gras.
|
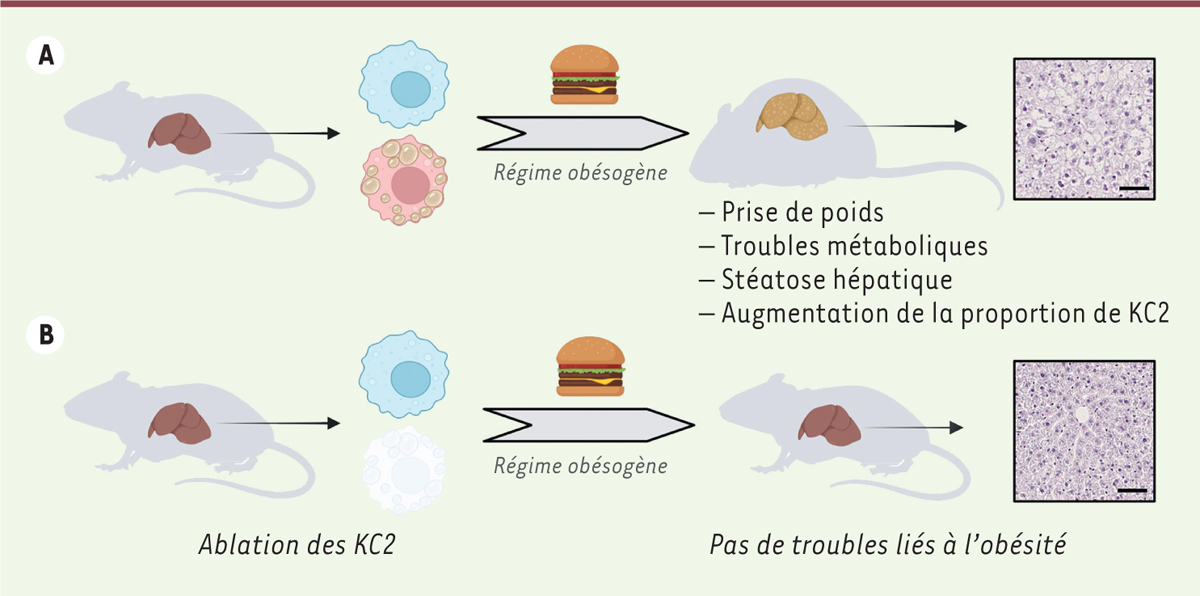
L’ablation des CD206+ ESAM+ KC2 module les troubles associés à l’obesité
Pour étudier ces fonctions métaboliques observées dans les KC2, nous avons utilisé un régime alimentaire obésogène très riche en lipides (ou régime HFD pour
high fat diet
) et avons étudié ses effets sur les KC. Les animaux soumis à ce type de régime gagnent rapidement du poids et développent une stéatose hépatique prononcée après plusieurs semaines
(
Figure 3A
)
. En revanche, les animaux chez lesquels la population KC2 a été éliminée, se révèlent protégés de la prise de poids et des complications liées à l’obésité, comme la dérégulation de la glycémie et la stéatose hépatique
(
Figure 3B
)
.
 | Figure 3.
Le ciblage des CD206
+
ESAM
+
KC module le métabolisme. A.
Les souris soumises à un régime obésogène riche en lipides développent des troubles associés à l’obésité, comme une prise de poids et une stéatose hépatique.
B.
Lorsque la population KC2 est éliminée, les animaux soumis au même régime sont protégés de ces troubles.
|
|
Dans cette étude, nous avons étudié l’hétérogénéité des KC, une question posée depuis plusieurs décennies mais ayant fourni des résultats contradictoires, notamment en raison de la difficulté à discerner les KC des autres macrophages dérivés de monocytes recrutés dans le foie. Grâce à notre approche « multi-omique » à haut débit sur cellules uniques et à l’utilisation de modèles murins spécifiques, nous avons notamment identifié une sous-population de KC impliquée dans la régulation du métabolisme. Bien que cette population de KC2 soit naturellement présente dans le foie sain, nos données montrent une augmentation de leur présence (au moins en proportion), lorsque les souris sont placées sous régime obésogène. Une perspective de ce travail est la mise en évidence et la caractérisation de cette population chez l’homme et l’étude de ses rôles dans le développement des maladies métaboliques. Outre l’obésité, d’autres maladies, comme la stéatose hépatique non-alcoolique (NASH pour
non-alcoholic steatohepatitis
), deviennent des problèmes de santé publique majeurs. La prise en compte de cette population de KC2 est ainsi importante à considérer dans l’étude de ces maladies [
7
]. Des macrophages dérivés de monocytes circulants sont mobilisés dans le contexte de la NASH [
8
,
9
], mais il semble qu’une fraction de KC natives pourrait également être impliquée dans cette maladie dont l’étiologie reste méconnue.
Une question restée sans réponse dans notre étude, est celle de la localisation des deux types de KC. Le foie est un organe arborant une zonation prononcée. Il a ainsi été montré que les hépatocytes au contact de la veine porte avaient une identité assez différente de ceux proches des veines centro-lobulaires [
10
]. Nous n’avons pas observé de répartition spatiale particulière pour les deux sous-populations de KC, ce qui suggère que la niche des KC2 pourrait être façonnée par des interactions très locales, notamment avec des cellules de leur environnement proche, comme les cellules endothéliales ou les cellules étoilées, deux populations qui interagissent fortement avec les KC. Une meilleure compréhension de l’hétérogénéité de ces populations du parenchyme hépatique pourrait donc se révéler déterminante pour éclaircir les raisons de l’émergence de ces deux profils de KC.
Parallèlement à notre étude, nous avons examiné la capacité des KC2 à présenter les antigènes. Dans un modèle d’hépatite induite par l’HBV (virus de l’hépatite B), nous avons pu établir que les KC2 répondaient à l’interleukine 2 et participaient à la mise en place de la réponse immunitaire adaptative contre le virus HBV [
11
]. Ces observations ouvrent ainsi de nouvelles perspectives, mais elles valident aussi notre approche et interrogent sur la nécessaire analyse de l’hétérogénéité des macrophages au sein d’un même tissu afin d’identifier leurs fonctions.
|
Les auteurs déclarent n’avoir aucun lien d’intérêt concernant les données publiées dans cet article.
|
Footnotes |
1.
Bleriot
C
,
Chakarov
S
,
Ginhoux
F
.
Determinants of Resident Tissue Macrophage Identity and Function.
.
Immunity.
2020;
;
52
:
:957.
–
70
.
2.
Squarzoni
P
,
Oller
G
,
Hoeffel
G
,
et al.
Microglia modulate wiring of the embryonic forebrain.
.
Cell Rep.
2014;
;
8
:
:1271.
–
1279
.
3.
Bleriot
C
,
Ginhoux
F
.
Understanding the Heterogeneity of Resident Liver Macrophages.
.
Front Immunol.
2019;
;
10
:
:2694.
.
4.
David
BA
,
Rezende
RM
,
Antunes
MM
,
et al.
Combination of Mass Cytometry and Imaging Analysis Reveals Origin, Location, and Functional Repopulation of Liver Myeloid Cells in Mice.
.
Gastroenterology.
2016;
;
151
:
:1176.
–
1191
.
5.
Klein
I
,
Cornejo
JC
,
Polakos
NK
,
et al.
Kupffer cell heterogeneity: functional properties of bone marrow derived and sessile hepatic macrophages.
.
Blood.
2007;
;
110
:
:4077.
–
4085
.
6.
Liu
Z
,
Gu
Y
,
Chakarov
S
,
et al.
Fate Mapping via Ms4a3-Expression History Traces Monocyte-Derived Cells.
.
Cell.
2019;
;
178
:
(1509–25)
:e19.
.
7.
Kanwal
F
,
Shubrook
JH
,
Younossi
Z
,
et al.
Preparing for the NASH Epidemic: A Call to Action.
.
Gastroenterology.
2021;
;
161
:
:1030.
–
42 e8
.
8.
Guilliams
M
,
Bonnardel
J
,
Haest
B
,
et al.
Spatial proteogenomics reveals distinct and evolutionarily conserved hepatic macrophage niches.
.
Cell.
2022;
;
185
:
:379.
–
96 e38
.
9.
Devisscher
L
,
Scott
CL
,
Lefere
S
,
et al.
Non-alcoholic steatohepatitis induces transient changes within the liver macrophage pool.
.
Cell Immunol.
2017;
;
322
:
:74.
–
83
.
10.
Halpern
KB
,
Shenhav
R
,
Matcovitch-Natan
O
,
et al.
Single-cell spatial reconstruction reveals global division of labour in the mammalian liver.
.
Nature.
2017;
;
542
:
:352.
–
356
.
11.
De Simone
G
,
Andreata
F
,
Bleriot
C
,
et al.
Identification of a Kupffer cell subset capable of reverting the T cell dysfunction induced by hepatocellular priming.
.
Immunity.
2021;
;
54
:
:2089.
–
100 e8
.
|