| |
| Med Sci (Paris). 38(5): 425–427. doi: 10.1051/medsci/2022050.La chromatine bivalente préserve la plasticité transcriptionnelle Léa Baudre,1 Céline Vallot,1 and Justine Marsolier1* 1CNRS UMR3244, Département de recherche translationnelle, Institut Curie, Université PSL, Paris, France MeSH keywords: Chromatine, Épigenèse génétique, Histone, Humains, génétique, métabolisme |
Toutes les cellules d’un organisme possèdent la même information génétique, et pourtant, les cellules différenciées de tissus différents ont des caractéristiques morphologiques et des fonctions distinctes. Ces caractéristiques cellulaires sont le résultat de l’activation ou de la répression de groupes de gènes spécifiques au cours de la différenciation des cellules souches embryonnaires issues de la masse cellulaire interne du blastocyste1,. Ce contrôle précis des programmes transcriptionnels repose en partie sur l’organisation du génome des cellules eucaryotes sous la forme de chromatine. En effet, la chromatine est constituée de nucléosomes2, et cette configuration chromatinienne, qui module finement l’accessibilité de l’ADN à la machinerie de transcription des gènes, participe donc à leur activation ou à leur répression. La méthylation de l’ADN et les modifications post-traductionnelles des histones sont deux mécanismes épigénétiques qui, en contrôlant la compaction de l’ADN et les programmes transcriptionnels, contrôlent le destin des cellules, notamment au cours du développement [1]. La méthylation de l’ADN est une marque épigénétique stable, héritable lors de la mitose, qui concerne préférentiellement les régions du génome riches en dinucléotides CpG3,, et qui est associée à la répression transcriptionnelle irréversible des gènes associés [1]. Les histones, quant à elles, peuvent être soumises à de nombreuses modifications post-traductionnelles incluant la méthylation ou l’acétylation [2]. Certaines de ces modifications, comme la tri-méthylation de la lysine en position 4 de l’histone H3 (H3K4me3), sont associées à une activation de l’expression génique, alors que d’autres, comme la tri-méthylation de la lysine en position 27 de l’histone H3 (H3K27me3), sont associées à la répression transcriptionnelle des gènes. Cependant, une structure chromatinienne atypique, bivalente, caractérisée par un co-enrichissement de H3K27me3 et H3K4me3 à certains emplacements du génome des cellules souches embryonnaires, a été identifiée [3]. Il a été proposé que dans les cellules souches embryonnaires, les gènes jouant un rôle décisionnel dans l’acquisition d’une identité cellulaire seraient dans cette configuration chromatinienne afin de permettre leur activation transcriptionnelle rapide durant l’embryogenèse [4, 5], mais cette hypothèse élégante reste à prouver. |
Promoteurs bivalents et activation transcriptionnelle Grâce à l’analyse des statuts chromatiniens et transcriptionnels des gènes lors de la différenciation de cellules souches embryonnaires murines en cellules de type épiblastique (EpiLC pour post-implantation epiblast-like cells) in vitro, Kumar et ses collègues ont montré que les gènes possédant un promoteur bivalent (« gènes bivalents ») et qui sont réprimés dans les cellules souches embryonnaires ne s’activent pas plus vite que les autres. Parmi les gènes fortement exprimés dans les cellules EpiLC, la présence ainsi que l’enrichissement de H3K4me3 au niveau des gènes ayant un promoteur bivalent dans les cellules souches embryonnaires (dont les cellules EpiLC sont issues) n’entraînent pas une activation plus rapide ou plus forte des promoteurs correspondants par rapport à d’autres gènes ne présentant pas un tel enrichissement de H3K4me3 [6]. Ce résultat a également été confirmé lors d’étapes plus tardives de la différenciation des cellules souches embryonnaires humaines pour former les trois feuillets embryonnaires (ectoderme, mésoderme et endoderme), après la gastrulation [6]. L’étude du potentiel transcriptionnel de l’ARN polymérase II (ARNPII) pour les gènes présentant une structure chromatinienne bivalente permet aux auteurs de proposer que la plupart des promoteurs bivalents ne présentent pas une ARNPII engagée transcriptionnellement, mais plutôt « prête à s’engager ». Lorsqu’elle est engagée dans la transcription, l’ARN-PII est phosphorylée sur le résidu sérine en position 5 (S5p) par le complexe TFIIH (transcription factor II H), tandis que lorsqu’elle est prête à s’engager sur les promoteurs bivalents, la phosphorylation S5p de l’ARNPII est effectuée par la kinase MAPK1 (mitogen-activated protein kinase 1), qui est préférentiellement présente sur les gènes cibles du complexe de répression transcriptionnelle PRC2 (polycomb repressive complex 2), responsable de la tri-méthylation de H3K27. La présence, sur certains gènes bivalents, de cette RNAPII « prête pour la transcription » ne permet pas pour autant une activation transcriptionnelle plus rapide par rapport aux gènes bivalents qui en sont dépourvus. |
Établissement de la chromatine bivalente : un produit de l’activité du complexe PRC2 sur des îlots d’ADN riches en dinucléotides CpG Afin de mieux comprendre le contrôle de la bivalence de la chromatine, Kumar et ses collègues ont étudié les caractéristiques de la séquence de l’ADN dans les régions bivalentes du génome. Ils ont montré que la présence de H3K4me3 dans les régions promotrices bivalentes est fortement corrélée avec la densité en dinucléotides CpG [6]. De plus, une autre équipe avait montré précédemment que les régions enrichies en dinucléotides CpG non méthylés permettent d’établir per se des domaines enrichis en H3K4me3 [7]. En parallèle, Kumar et ses collègues ont aussi observé que la densité des dinucléotides CpG associée à l’enrichissement en H3K27me3 permet de prédire les gènes ayant une configuration bivalente de la chromatine associée, et que le nombre de gènes bivalents identifiés dans différents types cellulaires est corrélé au degré d’expression de EZH2, la sous-unité catalytique du complexe PRC2 [6]. L’ensemble de ces résultats suggèrent que la structure bivalente de la chromatine serait un produit de l’activité du complexe PRC2 sur des séquences d’ADN riches en dinucléotides CpG. |
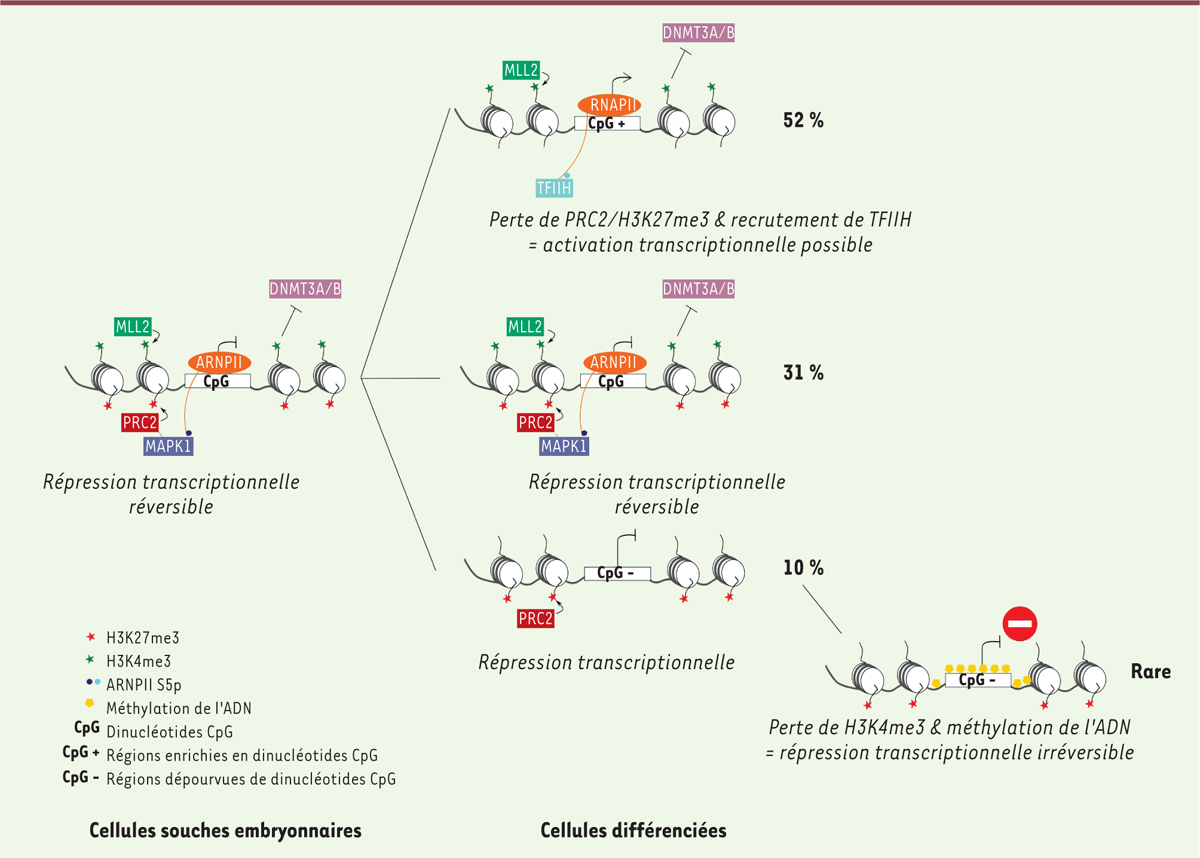
Destin de la chromatine bivalente lors du développement embryonnaire Au cours de la différenciation des cellules souches embryonnaires, il avait été postulé que les gènes bivalents pourraient avoir deux destins : conserver uniquement H3K4me3 ou conserver uniquement H3K27me3, ce qui permettrait respectivement l’expression ou la répression du gène correspondant [4]. Les auteurs de cette nouvelle étude proposent que les gènes ayant une configuration bivalente de la chromatine associée à leurs promoteurs peuvent avoir trois destins différents : rester dans une configuration chromatinienne bivalente (environ 30 % des cas ; répression transcriptionnelle réversible), conserver uniquement H3K4me3 (environ 50 % des cas ; activation transcriptionnelle possible mais non garantie ; préférentiellement dans les régions riches en dinucléotides CpG), ou encore conserver uniquement H3K27me3 (environ 10 % des cas ; répression transcriptionnelle ; préférentiellement dans les régions dépourvues de séquences CpG) (Figure 1). Inversement, certains promoteurs de gènes acquièrent une structure chromatinienne bivalente au cours de la différenciation des cellules souches embryonnaires, et ils correspondent majoritairement à des régions de l’ADN riches en dinucléotides CpG et présentant un enrichissement en H3K4me3 de la chromatine associée dans les cellules souches embryonnaires. Cette dernière observation renforce l’hypothèse selon laquelle la structure bivalente de la chromatine résulte de l’activité du complexe PRC2 et de la tri-méthylation de H3K27 en regard des séquences d’ADN riches en CpG.  | Figure 1.Bivalence de la chromatine et développement. Modèle proposé par Kumar et al. [ 6]. ESC : embryonic stem cells ; ARNPII : ARN polymérise I ; MAPK1 : mitogen-activated protein kinase 1 ; TFIIH : transcription factor II H ; PRC2 : polycomb repressive complex 2 ; DNMT3A/B : DNA methyltransferase 3 α/β ; MLL2/KMT2D : histone-lysine N-methyltransferase 2D. |
|
Chromatine bivalente : une protection contre la méthylation de l’ADN ? La bivalence de la chromatine semble également jouer un rôle dans les cellules tumorales. En effet, il a été montré que deux-tiers des promoteurs de gènes hyperméthylés dans les cellules cancéreuses ont une configuration bivalente de la chromatine associée et sont majoritairement des cibles du complexe PRC2 dans les cellules souches embryonnaires [6]. Cette observation suggère que la bivalence de la chromatine ou la présence du complexe PRC2 pourrait prédisposer ces régions à une méthylation de l’ADN de novo, donc à une répression transcriptionnelle irréversible dans les cellules cancéreuses [8]. Kumar et ses collègues ont testé cette hypothèse dans deux types de cancers : ostéosarcome et cancers colorectaux [6]. Ils proposent au contraire que la configuration bivalente de la chromatine protège les promoteurs des gènes d’une méthylation de l’ADN de novo et d’une répression transcriptionnelle irréversible, et que la perte de H3K4me3, i.e. la perte de la structure bivalente de la chromatine dans ces régions génomiques (préférentiellement riches en dinucléotides CpG), augmente la probabilité d’une méthylation de l’ADN dans les cellules tumorales [6]. Ce mécanisme est également observé au cours de la différenciation des cellules souches embryonnaires en cellules épiblastiques, mais seulement une minorité de gènes bivalents (préférentiellement pauvres en dinucléotides CpG) sont concernés par une hyperméthylation de l’ADN à la suite d’une perte de H3K4me3 (Figure 1). |
La présence d’une structure chromatinienne bivalente dans les régions promotrices de certains gènes a d’abord été décrite dans les cellules souches embryonnaires [4], puis retrouvée dans les cellules différenciées [3, 8], ou plus récemment, dans des promoteurs de gènes clefs pour la résistance de cellules tumorales à la chimiothérapie [9]. Au cours du développement embryonnaire, il semblerait que les gènes bivalents ne s’activent pas plus vite que les autres dans les cellules en voie de différenciation. Cependant, la présence simultanée de H3K27me3 et H3K4me3 maintient ces gènes dans un état de répression transcriptionnelle réversible. Kumar et ses collègues montrent en effet que la bivalence peut notamment protéger l’ADN de la méthylation, donc d’une répression définitive de l’expression génique. La bivalence du promoteur d’un gène ne permettrait pas nécessairement une activation rapide de la transcription, mais préserverait plutôt la possibilité de cette activation. Ainsi les gènes dont le promoteur est bivalent seraient en quelque sorte « actionnables », à l’inverse des gènes réprimés par méthylation de l’ADN par exemple. Dans le cas du cancer du sein, il a été montré que ces gènes bivalents sont activables en effaçant simplement la marque H3K27me3 du génome [9]. La bivalence chromatinienne devra désormais être caractérisée dans les divers tissus et dans différents contextes physiologiques ou pathologiques afin de déterminer son universalité ainsi que sa variation dynamique au cours du temps. |
Les auteures déclarent n’avoir aucun lien d’intérêt concernant les données publiées dans cet article. |
Footnotes |
1.
Allis CD, Jenuwein T. The molecular hallmarks of epigenetic control . Nat Rev Genet. 2016; ; 17 : :487.–500. 2.
Kouzarides T. Chromatin modifications and their function . Cell. 2007; ; 128 : :693.–705. 3.
Mikkelsen TS, Ku M, Jaffe DB, et al. Genome-wide maps of chromatin state in pluripotent and lineage-committed cells . Nature. 2007; ; 448 : :553.–560. 4.
Bernstein BE, Mikkelsen TS, Xie X, et al. A bivalent chromatin structure marks key developmental genes in embryonic stem cells . Cell. 2006; ; 125 : :315.–326. 5.
Voigt P, Tee W-W, Reinberg D. A double take on bivalent promoters . Genes Dev. 2013; ; 27 : :1318.–1338. 6.
Kumar D, Cinghu S, Oldfield AJ, et al. Decoding the function of bivalent chromatin in development and cancer . Genome Res. 2021; gr.275736.121.. 7.
Thomson JP, Skene PJ, Selfridge J, et al. CpG islands influence chromatin structure via the CpG-binding protein Cfp1 . Nature. 2010; ; 464 : :1082.–1086. 8.
Ohm JE, McGarvey KM, Yu X, et al. A stem cell-like chromatin pattern may predispose tumor suppressor genes to DNA hypermethylation and heritable silencing . Nat Genet. 2007; ; 39 : :237.–242. 9.
Marsolier J, Prompsy P, Durand A, et al. H3K27me3 conditions chemotolerance in triple-negative breast cancer . Nat Genet. 2022;; 54 : :459.–68. |