| |
| Med Sci (Paris). 38(4): 398–400. doi: 10.1051/medsci/2022032.Chiens, loups et gènes Chroniques génomiques Bertrand Jordan1* 1Biologiste, généticien et immunologiste, président d’Aprogène (Association pour la promotion de la génomique)
,
13007Marseille
,
France MeSH keywords: Allèles, Animaux, Chiens, Humains, Phylogenèse, Loups, génétique |
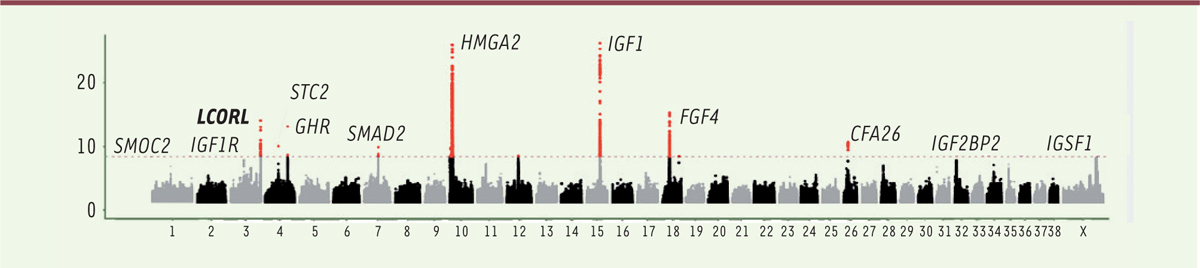
La taille à l’âge adulte est un des caractères les plus héritables chez les mammifères, pour l’homme comme pour d’autres espèces. Ce caractère facilement mesurable a fait l’objet de nombreuses études dès la mise au point des approches de balayage du génome (
GWAS, genome-wide association studies
). Elles ont révélé, dans l’espèce humaine, une hérédité très fortement polygénique : à l’heure actuelle, on a identifié plusieurs centaines de locus impliqués, chacun d’eux pris individuellement ayant un effet très limité sur la taille [
1
]. Plus récemment, ces études ont été étendues à l’espèce canine, qui constitue un cas particulièrement intéressant, compte tenu de la très grande différence de taille et de masse existant entre les diverses races de chiens : de deux kg environ pour le Chihuahua à plus de soixante pour le Terre-Neuve, qui appartient pourtant bien à la même espèce. Les résultats de ces études montrent une image assez différente de celle obtenue pour l’homme, avec un petit nombre de locus (moins de vingt au total) apparaissant dans les études les plus récentes [
2
]. Comme on le voit
(
Figure 1
)
, les deux principaux locus se situent au voisinage des gènes
HMGA2
(
high mobility group AT-hook 2
), un facteur de transcription dont certains mutants sont associés, chez l’homme, à une forme de nanisme (le syndrome de Silver-Russel) [
3
], et
IGF1
(
insulin-like growth factor 1
), qui code un facteur de croissance impliqué dans le développement embryonnaire et la croissance post-natale [
4
].
 | Figure 1.
Balayage du génome chez le chien (39 paires de chromosomes) pour la taille à l’âge adulte.
En ordonnée : niveau de significativité (−Log
10
de la probabilité que le résultat soit dû au hasard). La ligne pointillée rouge indique la limite au-delà de laquelle le résultat est considéré comme significatif. Noter que les pics repérés par un nom (acronyme) de gène ne signifient pas nécessairement que le variant se situe dans le gène : ils désignent une zone du chromosome couvrant quelques dizaines de mégabases (10
6
bases), contenant le gène indiqué mais aussi de l’ADN non codant et éventuellement d’autres gènes (extrait partiel et remanié de la figure 1 de [
2
]).
|
C’est à ce locus
IGF1
, pour lequel il a été montré qu’il était responsable d’environ 15 % de la différence de taille entre races [
2
], que nous allons nous intéresser maintenant. Il fait l’objet d’un article publié début 2022 [
5
], émanant d’une quinzaine d’équipes étatsuniennes, françaises, d’autres pays européens, russes et asiatiques et intitulé «
Sélection naturelle et dirigée portant sur un variant non codant associé à la taille chez les canidés anciens et modernes
»
1
. Vaste programme, qui recouvre en effet un très intéressant ensemble d’études.
|
Un variant fortement associé avec la taille et avec le niveau d’IGF1
Les auteurs ont analysé les génomes de plus de 400 chiens, déjà répertoriés dans les bases de données et provenant majoritairement du projet
Dog10K
, un consortium international visant à séquencer l’ADN de 10 000 canidés en cinq ans [
6
]. Ils y ont ajouté les séquences d’une vingtaine de canidés sauvages (chacals, coyotes, renards). Ils ont bien retrouvé l’association de la taille avec le locus du chromosome 15
(
Figure 1
)
qui contient le gène
IGF-1
, et ont montré que le variant polymorphique le plus significatif (le SNP [
single nucleotide polymorphism
] rs22397284) se situe dans le dernier exon d’un long ARN non codant (lncARN) appelé
IGF1-AS
(AS pour
antisense
) car il est complémentaire de l’ARN messager du gène
IGF1
. Cet ARN antisens doit probablement réguler l’expression d’
IGF1
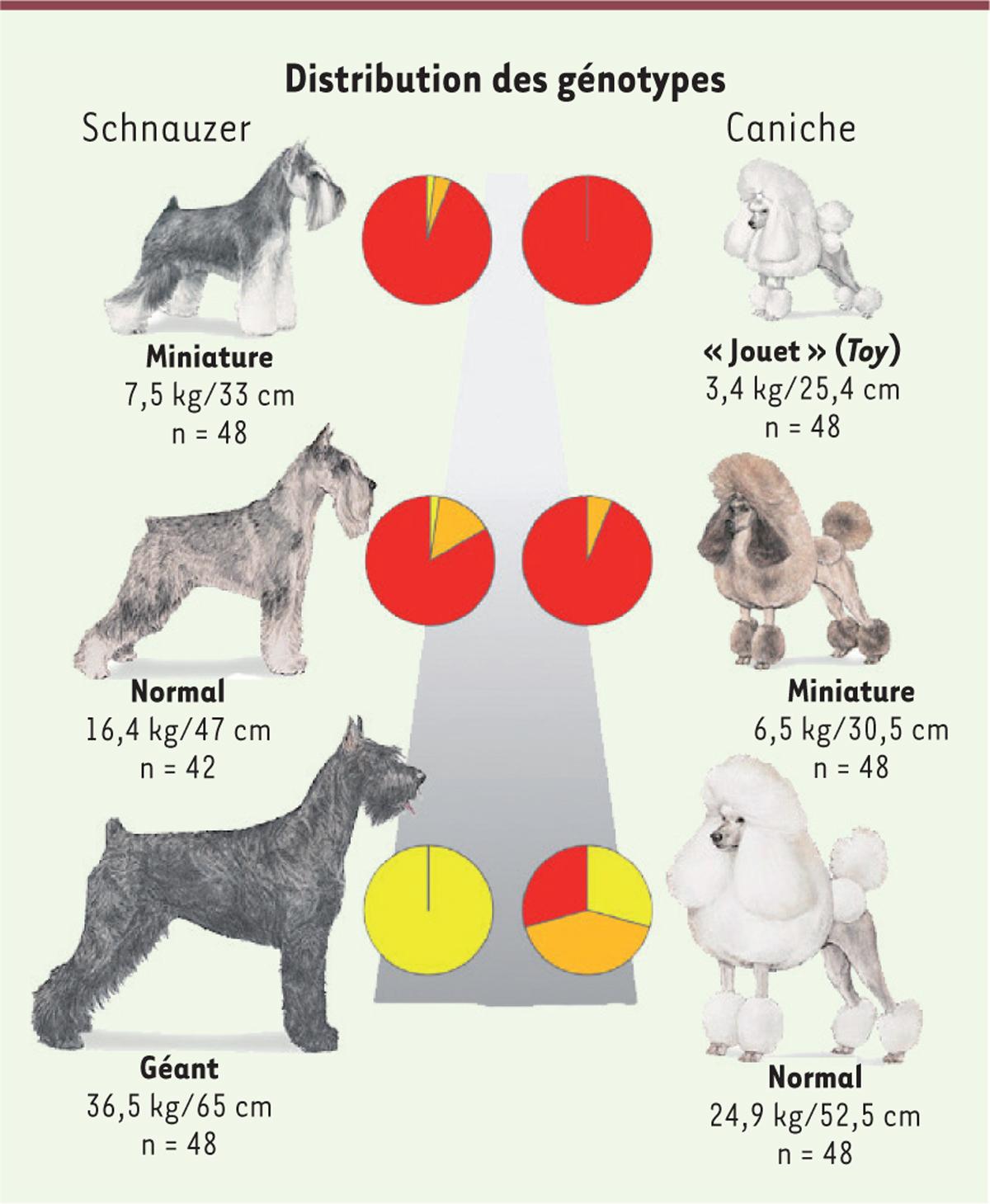
en se fixant sur son ARN messager, modulant ainsi sa traduction : on imagine facilement que le nucléotide polymorphe mis en évidence dans ce variant peut modifier cette régulation. Les deux allèles présents dans la population canine pour ce SNP correspondent à la présence soit d’une cytosine (C), associée à la petite taille, soit d’une thymine (T), associée à la grande taille. La
Figure 2
illustre cette association pour deux races canines existant en petit, moyen ou grand « modèle », le caniche et le schnauzer : on voit que la corrélation est très significative ! Plus le chien est petit, plus son génotype contient une cytosine, et inversement, plus il est grand, plus la thymine est présente. La mesure de la concentration sérique de la protéine IGF1 dans le sang d’une cinquantaine de chiens montre que celle-ci est positivement corrélée avec la masse corporelle des chiens (et avec la présence de l’allèle T). Tout cela laisse supposer que le polymorphisme C/T observé joue sur l’association du transcrit antisens avec l’ARN messager du gène
IGF1,
modifiant sa traduction. Cela change en conséquence la quantité de la protéine correspondante IGF1, un facteur de croissance analogue à l’insuline, comme l’indique son nom, et influe ainsi sur la taille de l’animal. On aimerait néanmoins en savoir plus sur l’origine de ce polymorphisme et sur son histoire évolutive…
 | Figure 2.
Répartition des allèles (C) (petite taille) et (T) (grande taille) du SNP rs22397284 dans le gène IGF1-AS pour deux races de chiens, chacune comportant différentes tailles.
Le poids des chiens varie (de haut en bas de la figure) de cinq à presque dix fois. La proportion des deux allèles est indiquéepar les « camemberts » : en rouge, les homozygotes C/C (ultra-majoritaires chez les plus petits) ; en jaune, les homozygotes T/T ; et en marron, les hétérozygotes C/T. n : nombre d’animaux analysés (extrait partiel et remanié de la figure 1 de [
5
]).
|
|
Histoire évolutive des allèles du gène
IGF1
Les races actuelles de chiens ont été, pour l’essentiel, créées au cours des deux derniers siècles par l’action des éleveurs qui ont exercé une sélection très efficace pour isoler et fixer les caractères recherchés, notamment une taille réduite pour certains chiens de compagnie [
7
], et, selon leurs besoins, pour la chasse (petits chiens pour la chasse au renard, par exemple). Ils ont ainsi sélectionné les porteurs de l’allèle C (petite taille) du gène
IGF1-AS
que l’on retrouve à l’état homozygote chez les petits chiens. Mais quelle est l’histoire de cet allèle au cours des trente ou quarante mille ans pendant lesquels les canidés ont évolué en diverses branches (chacals, renards, coyotes, loups), puis, il y a quinze ou vingt mille ans, lors de la domestication du loup, pour donner naissance aux chiens ? On pourrait penser que cet allèle est apparu récemment au sein de l’espèce canine, et qu’il a été ensuite sélectionné par les éleveurs d’animaux de compagnie. En remontant dans le passé, l’analyse de l’ADN ancien permet d’éclairer cette question : une trentaine de génomes de chiens et de loups datant de mille à plus de cinquante mille ans ont déjà été séquencés et permettent de suivre la fréquence des allèles de
IGF1-AS
, tandis que la taille et la masse de ces animaux peuvent être estimées à partir de leurs restes. Les auteurs y ont ajouté les séquences de canidés génétiquement plus distants restés sauvages (coyotes, chacals, renards). Il en résulte que l’allèle C est généralement présent à l’état homozygote chez les canidés modernes de petite taille, mais qu’il est aussi retrouvé (à l’état hétérozygote, et à basse fréquence) chez les loups anciens et actuels, tandis que sa fréquence chez les chiens anciens est corrélée avec leur taille estimée. À partir de l’ensemble de ces résultats, les auteurs considèrent que l’allèle ancestral du gène
IGF1-AS
chez les canidés est l’allèle C (petite taille). L’allèle T (grande taille) serait ensuite apparu il y a plus de cinquante mille ans, car il est présent à l’état hétérozygote dans le génome de loup le plus ancien retrouvé, provenant de la région de la rivière Tirektyakh en Sibérie orientale et daté à – 52 500 ans [
5
]. Cet allèle serait rapidement devenu majoritaire chez les loups, pour lesquels la grande taille présente un avantage sélectif, sans pour autant que l’allèle C ne disparaisse complètement de la population. Ce dernier serait resté majoritaire chez les canidés de petite taille où il est souvent retrouvé à l’état homozygote. Enfin, la domestication du loup à partir de – 15 000 ans donna naissance à l’espèce canine, au sein de laquelle l’homme préhistorique opère alors une différenciation (chiens de garde, de chasse, d’élevage, etc.), avant que des sélections rigoureuses et systématiques n’aboutissent, au cours de XIX
e
siècle, aux races bien définies que nous connaissons aujourd’hui [
7
], et pour lesquelles la sélection a notamment porté, à travers le choix de la taille, sur les allèles du gène
IGF1-AS
.
|
Fascinant – mais encore incomplet
Ce très joli travail fait appel à des techniques sophistiquées et notamment à l’analyse de l’ADN ancien, une approche qui a révolutionné l’anthropologie et a remis en cause bien des interprétations, en révélant toute la complexité de l’histoire humaine [
8
]. Il illustre comment un allèle d’un gène peut presque disparaître puis redevenir important au sein d’une population en fonction des pressions de sélection qu’elle subit. Reste, néanmoins, qu’on aimerait en savoir plus sur le mécanisme par lequel le simple changement d’une cytosine en thymine au sein de la séquence d’un ARN antisens peut induire un aussi important changement de phénotype. Les auteurs font allusion à des investigations sur l’existence de l’ARN non codant transcrit du gène
IGF1-AS
, et sur son association avec le transcrit du gène
IGF1,
mais il ne semble pas que l’ARN non codant modifie l’expression du gène, ni le niveau de son transcrit (données rapidement évoquées dans la discussion de l’article) : le mécanisme est sans doute plus subtil, et il reste à découvrir. N’oublions pas enfin que – contrairement à l’impression que peut donner une lecture rapide de l’article – l’allèle de
IGF1-AS
n’est pas, et de loin, le seul déterminant de la taille des chiens : selon les analyses GWAS précédemment rapportées [
5
]
(
Figure 1
)
, ce locus ne rend compte que d’environ 15 % de la variation de taille observée entre différentes races de chiens. Les caniches « jouets » de la
Figure 2
doivent donc porter toute une série d’allèles contribuant à leur petite taille, et
IGF1-AS
ne fait que concourir à ce caractère, sans être à proprement parler causal. Cette nuance est souvent absente des présentations journalistiques de ce travail, même lorsqu’elles émanent de revues prestigieuses
2
…
|
L’auteur déclare n’avoir aucun lien d’intérêt concernant les données publiées dans cet article.
|
Footnotes |
1.
Guo
MH
,
Hirschhorn
JN
,
Dauber
A
.
Insights and Implications of genome-wide association studies of height.
.
J Clin Endocrinol Metab.
2018;
;
103
:
:3155.
–
3168
.
2.
Plassais
J
,
Kim
J
,
Davis
BW
,
et al.
Whole genome sequencing of canids reveals genomic regions under selection and variants influencing morphology.
.
Nat Commun.
2019;
;
10
:
:1489.
.
3.
De Crescenzo
A
,
Citro
V
,
Freschi
A
,
et al.
A. A splicing mutation of the HMGA2 gene is associated with Silver-Russell syndrome phenotype.
.
J Hum Genet.
2015;
;
60
:
:287.
–
293
.
4.
Baker
J
,
Liu
JP
,
Robertson
EJ
,
Efstratiadis
A
.
Role of insulin-like growth factors in embryonic and postnatal growth.
.
Cell.
1993;
;
75
:
:73.
–
82
.
5.
Plassais
J
,
vonHoldt
BM
,
Parker
HG
,
et al.
Natural and human-driven selection of a single non-coding body size variant in ancient and modern canids.
.
Curr Biol.
2022
;
S0960–9822(21)01723–1.
.
6.
Ostrander
EA
,
Wang
GD
,
Larson
G
,
et al.
Dog10K Consortium. Dog10K: an international sequencing effort to advance studies of canine domestication, phenotypes and health.
.
Natl Sci Rev.
2019;
;
6
:
:810.
–
24
.
7.
Worboys
M
,
Strange
JM
,
Pemberton
N
.
The invention of the modern dog: breed and blood in Victorian britain.
.
Baltimore:
:
Johns Hopkins University Press;
,
2018
:
:304.
p.
8.
Haber
M
,
Mezzavilla
M
,
Xue
Y
,
Tyler-Smith
C
.
Ancient DNA and the rewriting of human history: be sparing with Occam’s razor.
.
Genome Biol.
2016;
;
17
:
:1.
.
9.
Callaway
E
.
Big dog, little dog: mutation explains range of canine sizes.
.
Nature.
2022;
;
602
:
:18.
.
|