| |
| Med Sci (Paris). 38(2): 168–176. doi: 10.1051/medsci/2022008.L’importance des télomères dans les infections par les Herpèsvirus humains-6A/B Vanessa Collin1 and Louis Flamand1,2* 1Axe des maladies infectieuses et immunitaires, Centre de recherche du CHU de Québec - Université Laval, Québec
,
2705 boulevard Laurier
,
Québec
,
Canada 2Département de microbiologie, maladies infectieuses et immunologie, Faculté de médecine, Université Laval, Québec
,
2705 boulevard Laurier
,
Québec
,
Canada |
Vignette (Photo © Inserm – Damien Sanlaville/Chloé Tessereau).
La famille des
Herpesviridae
fait partie des virus les mieux adaptés pour établir des infections persistantes. Cette famille de virus à ADN double brin (ADNdb) comprend plus de 100 différents virus qui infectent les vertébrés. Chez l’homme, on les subdivise en trois sous-familles selon leurs caractéristiques biologiques : les a
-Herpesvirinae
(
Herpes Simplex Virus
1,
Herpes Simplex Virus 2
,
Varicella Zoster Virus
), les b
-Herpesvirinae
(
Human Herpesvirus-6A et -6B
,
Human Herpesvirus-7
,
Cytomegalovirus
) et les g
-Herpesvirinae
(
Epstein-Barr Virus [EBV]
,
Human Herpesvirus-8
). À la suite de l’infection primaire, les Herpèsvirus maintiennent leur génome dans la cellule hôte sous forme épisomale en exprimant de façon minimale leurs gènes viraux. Ceci leur permet d’établir un état de latence (état silencieux) afin de persister chez les hôtes toute la vie de ces derniers. La latence est un état de quiescence réversible à partir duquel le virus peut se réactiver et conduire à des infections secondaires. L’infection active des Herpèsvirus se définit par la phase lytique au cours de laquelle le génome viral est transcrit puis traduit en protéines afin de mener à sa réplication, aboutissant à la synthèse de nouveaux virions. Pour qu’un virus puisse se répliquer, l’environnement cellulaire doit être favorable : les concentrations des protéines virales et cellulaires, et diverses molécules nécessaires à la réplication virale, doivent être en quantités suffisantes. En l’absence de telles conditions, l’état de latence est favorisé.
Les Herpèsvirus humains-6A/B (HHV-6A/B) sont des
Roseolovirus
ubiquitaires dont la prévalence mondiale est de 97 % dès l’âge de deux ans. L’HHV-6B cause l’
exanthème subit
, une maladie infantile caractérisée par des éruptions cutanées, des détresses respiratoires et dans certains cas, des épilepsies. La pathologie liée à HHV-6A est moins connue, mais l’infection par ce virus pourrait être associée au développement de certaines maladies neurodégénératives [
1
]. Malgré leurs divergences concernant les maladies qu’ils provoquent, HHV6A et HHV6B partagent 94 % d’homologie nucléotidique dans leurs génomes et la capacité unique d’intégrer ceux-ci aux télomères humains [
2
]. L’intégration du génome des HHV-6A/B à cet endroit des chromosomes assure une persistance virale durable. Au cours de ces dernières années, divers travaux ont montré l’implication des télomères, des structures présentes aux extrémités des chromosomes, au cours des infections par les Herpèsvirus. Dans cette revue, nous décrirons d’abord les télomères puis leurs rôles au cours de l’infection par les HHV-6A/B : non seulement les télomères favorisent la réplication de ces virus, mais l’intégration des génomes des HHV-6A/B dans ces structures permet l’établissement d’un état de latence virale.
|
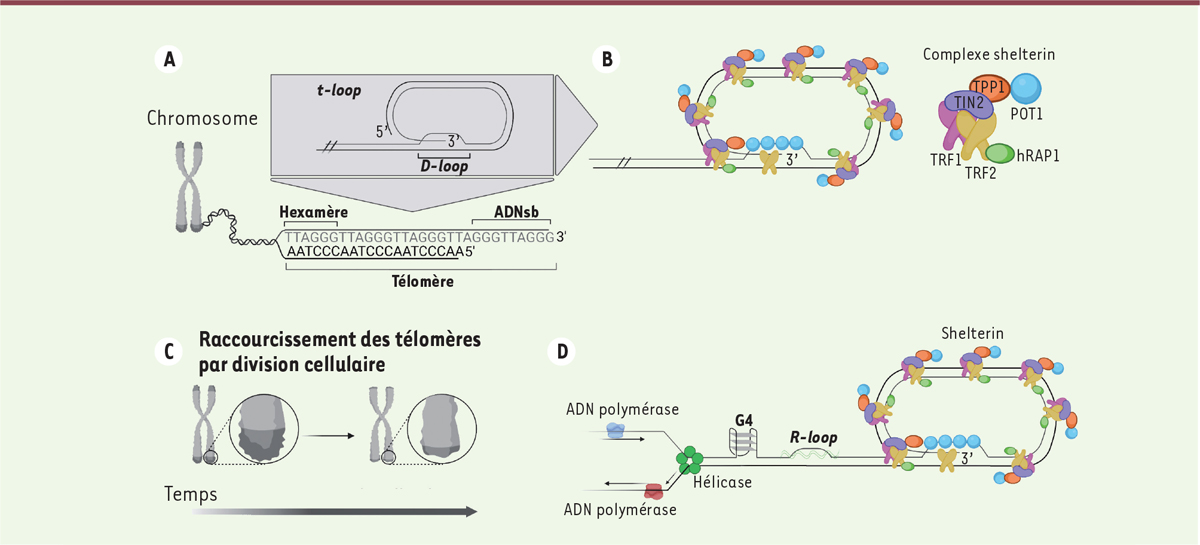
Les extrémités des chromosomes sont composées chacune d’un télomère, une séquence d’ADN TTAGGG répétée, pouvant atteindre des longueurs de 5 à 15 kpb (kilos paires de bases)
(
Figure 1A
)
[
3
]. L’extrémité 3’ de chaque télomère se termine par un ADN simple brin (ADNsb) de 50 à 200 pb (paires de bases) riches en nucléotides G (guanine). Afin d’assurer l’intégrité du télomère, l’ADNsb envahit l’ADNdb télomérique afin de former une structure tridimensionnelle en forme de boucle, la
t-loop
[
4
]. Cette structure, mise en évidence par des techniques de microscopie électronique, est formée et maintenue par les protéines du complexe
shelterin (
Figure 1B
)
[
5
,
6
]. Ce complexe est formé de six protéines : TRF1 (
telomere repeat-binding factor 1
), TRF2 (
telomere repeat-binding factor 2
), POT1 (
protection of telomere 1
), TPP1 (
shelterin complex subunit and telomerase recruitment factor 1
), RAP1 (
TRF2-interacting protein 1
) et TIN2 (
TRF1-interacting nuclear factor 2
).
 | Figure 1.
Compositions nucléiques et protéiques des télomères. A.
Chaque extrémité d’un chromosome possède un télomère (région gris foncé), composé d’hexamères « TTAGGG » dans l’ADNdb et un ADNsb en 3’, riche en nucléotides G. L’ADNsb en 3’ « envahit » l’ADNdb des télomères en formant une structure
D-loop
qui ferme le télomère sous forme de boucle,
t-loop
(encadré gris).
B.
La
t-loop
est formée et maintenue par le complexe
shelterin
, composé des protéines TRF1, TRF2, hRAP1, TIN2, TPP1 et POT1 (voir le texte).
C.
Schéma du raccourcissement des télomères à la suite de plusieurs divisions cellulaires.
D.
Structures secondaires présentes au niveau des télomères, qui bloquent le travail de l’ADN polymérase. ADNdb : ADN double brin ; ADNsb : ADN simple brin ; G4 : quadruplex de guanines ;
R-loop
: hybride ADN:ARN avec un TERRA.
|
Le rôle principal des télomères est de prévenir la perte de l’information génétique du génome. Lors de la réplication de l’ADN génomique, 50 à 100 pb sont en effet perdues, en raison de l’incapacité de l’ADN polymérase (ADN pol) à synthétiser l’extrémité 3’ du brin discontinu du génome. Pour y pallier, les cellules expriment la télomérase, un complexe moléculaire composé de plusieurs protéines, dont l’enzyme transcriptase inverse TERT (
telomerase reverse transcriptase
) qui, une fois active, synthétise les séquences TTAGGG télomériques. L’expression la plus forte de la télomérase est détectée dans les cellules souches embryonnaires, les cellules germinales, les leucocytes ainsi que dans les cellules néoplasiques. Cependant, une fois les cellules différenciées en cellules somatiques saines, l’expression de l’enzyme diminue, voire est complètement réprimée [
7
]. Les séquences télomériques répétées (TMR) servent donc de région tampon pour éviter la perte d’information génétique à long terme. À cette fin, les télomères agissent comme une horloge moléculaire qui dicte les capacités réplicatives d’une cellule. Lorsque la longueur des télomères atteint un seuil critique, environ 13 hexamères, la cellule devient sénescente et cesse de se diviser (un phénomène que l’on nomme limite de Hayflick)
(
Figure 1C
)
[
8
].
En plus d’éviter la perte d’information génétique, les télomères servent à empêcher l’instabilité génomique. En absence de protection, l’ADNdb des télomères est reconnu comme une cassure double brin (CDB) par le mécanisme de réponse aux dommages de l’ADN appelé DDR (
DNA damage response
). Cette reconnaissance est à l’origine de la formation de foyers de signalisation, les TIF (
telomere dysfunction-induced foci
), qui recrutent les protéines impliquées dans la réparation des CDB [
9
]. Cela a comme conséquence la fusion des chromosomes ou la recombinaison des chromosomes entre eux, au niveau des télomères, avec la chromatide sœur ou avec un chromosome proche [
10
]. Ces réparations créent de l’instabilité génomique pouvant être responsable d’une sénescence cellulaire prématurée ou de l’apparition de cellules néoplasiques. Les protéines du complexe
shelterin
sont présentes au niveau des télomères pour bloquer la DDR. En particulier, TRF2 forme des homodimères qui lient directement les séquences télomériques TMR et empêchent la reconnaissance des CDB, en inhibant l’activité de la kinase ATM (
ataxia telangiectasia, mutated
), l’enzyme responsable de la formation des foyers TIF [
6
]. Récemment, une étude du groupe de Titia de Lange (université Rockefeller, New York, États-Unis) a montré l’intérêt de l’utilisation de la méthode STORM (
Stochastic Optical Reconstruction Microscopy
)
1,
pour démontrer l’importance de la présence de TRF2 dans la génération de la
t-loop
[
6
,
11
]. Parmi les diverses fonctions des protéines du complexe
shelterin
, il faut souligner leur capacité à remédier à la présence de structures secondaires qui bloquent l’accès de l’ADN pol aux télomères pour permettre son action
(
Figure 1D
)
. Les télomères sont en effet riches en G, ce qui favorise la formation de quadruplex G (G4), une structure difficilement prise en charge par l’ADN pol. De plus, la région subtélomérique possède un transcrit non codant (TERRA, pour
telomeric repeat-containing RNA
) capable de former des hybrides ARN:ADN (ou
R-loop
), une source additionnelle de structures secondaires. Mais le complexe
shelterin
peut lui-même être un obstacle pour l’ADN polymérase, s’il ne se dissocie pas de l’ADN pour permettre la réplication de la
t-loop
.
Ainsi, l’ensemble du complexe nucléoprotéique que sont les télomères participe au maintien de l’intégrité génomique, à la régulation de l’activité du complexe de la télomérase et,
in fine
, à celle de la prolifération cellulaire.
|
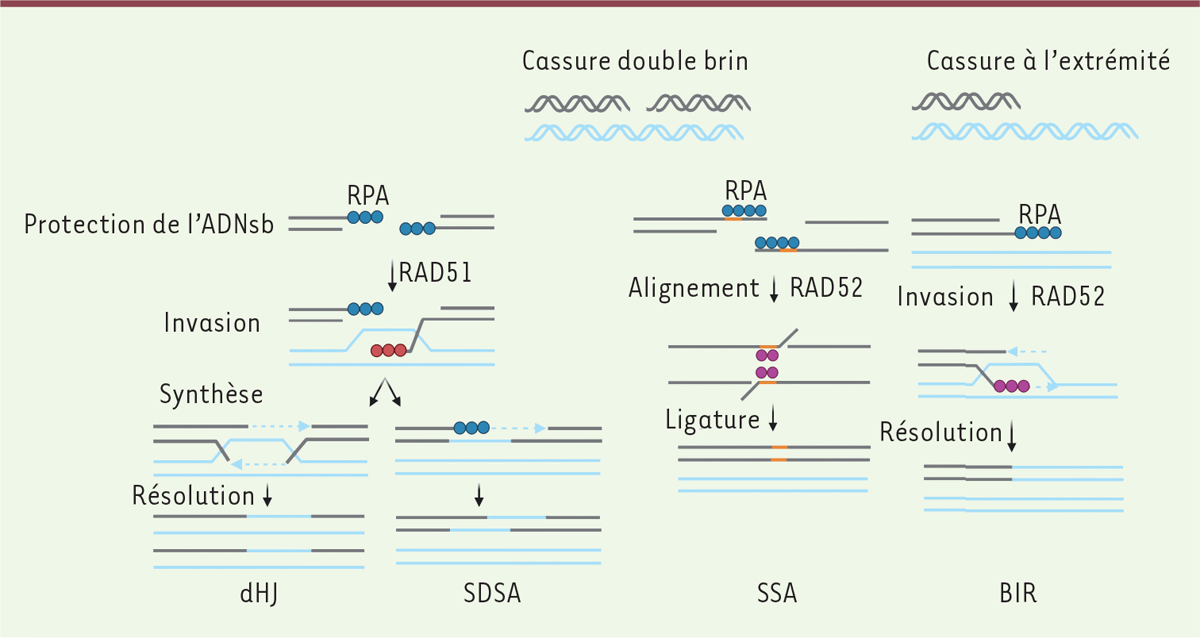
La réparation de l’ADN télomérique
Les structures secondaires présentées par les télomères et qui ne sont pas réparées, causent une augmentation de stress réplicatif provoquant des cassures double brin. La cellule répare l’ADN endommagé en utilisant la voie de la recombinaison homologue (RH), une voie sans erreur, puisqu’elle recopie l’information perdue en utilisant comme modèle les séquences homologues de la chromatide sœur. La RH est divisée en quatre voies : la dHJ (
double-holliday junction
), la SDSA (
synthesis-dependent strand annealing
), la SSA (
single-stranded annealing
) et la BIR (
break-induced replication
)
(
Figure 2
)
. La dHJ et la SDSA sont les voies classiques. Elles sont responsables de la conversion des gènes. Lors d’une CDB, l’ADNdb est digéré dans le sens 5’-3’ et génère un long ADNsb en 3’ qui « envahit » l’ADNdb de la chromatide sœur au niveau de la région homologue, grâce à une protéine de recombinaison, RAD51. L’ADN perdu est copié pour donner deux chromatides identiques. La dHJ diffère de la SDSA car elle est utilisée lors de la méiose pour permettre le réarrangement génétique par enjambement : il y échange de locus entre les chromatides, événement accompli lors de la reproduction sexuée. En l’absence de longues séquences homologues et de la protéine RAD51, la cellule utilise une voie de sauvetage, la SSA. La réparation par la voie SSA ne requiert pas de région homologue primaire entre deux chromatides. Elle utilise la résection de l’ADN jusqu’à l’obtention de séquences complémentaires entre les deux brins cassés, qui s’apparient grâce à la protéine RAD52. En revanche, la SSA est sujette à la perte d’information puisque les nucléotides perdus ne sont pas recopiés. Enfin, la voie BIR est utilisée seulement lorsqu’une extrémité de la cassure est présente dans le chromosome, comme c’est le cas d’un télomère libre. La première étape de la voie BIR est similaire à celle de la SDSA ; cependant, cette voie BIR n’est pas restreinte à la recombinaison affectant les chromatides sœurs et il peut y avoir une recombinaison avec un autre chromosome proche. De plus, l’invasion de l’ADNsb peut dépendre de RAD51; cependant, dans un contexte de télomère, elle repose sur RAD52 [
12
]. En réponse à l’invasion et à l’appariement des brins, l’ADN est alors synthétisé jusqu’à l’extrémité du génome, ce qui distingue la BIR des autres voies de réparation.
 | Figure 2.
Voies de la recombinaison homologue.
L’ADN d’une cassure double brin est digéré de 5’ vers 3’, laissant un ADN simple brin (ADNsb) protégé par la protéine RPA. En présence de longues séquences homologues, la protéine RAD51 s’associe à l’ADNsb et « envahit » la région homologue de la chromatide sœur. L’ADN perdu est synthétisé et résolu par un enjambement lors de la dHJ ou par un déplacement de brin lors de la SDSA. En absence de longues séquences homologues primaires, l’ADN est digéré jusqu’à l’obtention de micro-homologies entre les brins complémentaires, liées par la protéine RAD52. Les brèches sont remplies et l’ADN en excès digéré, résultant d’une réparation menant à une délétion de l’ADN, la SSA. Lors d’une cassure à une extrémité, la voie de BIR répare l’ADN à la suite de la digestion de l’ADNdb de 5’ vers 3’ ; l’ADNsb est protégé par RPA. La protéine RAD52 déplace RPA et s’associe à l’ADNsb. RAD52 « envahit » l’ADNsb en 3’ dans une région homologue d’une chromatide sœur ou d’un chromosome proche. La réparation s’achève en synthétisant l’entièreté du chromosome « envahi ». dHJ :
double-Holliday Junction
; SDSA :
Synthesis-Dependent Stand Annealing
; SSA :
Single-Stranded Annealing
; BIR :
Break-Induced Replication
.
|
|
La réplication des Herpèsvirus et l’utilisation du télosome
Lors de l’infection, le génome des Herpèsvirus est circularisé au niveau du noyau de la cellule grâce à des protéines cellulaires, dont le complexe ADN ligase IV/XCCR4 (
X-ray repair cross-complementing protein 4
) [
13
]. Une fois le génome circularisé, la réplication débute par l’association d’OBP (
origin-binding proteins
) à l’origine de réplication (OriR) du génome viral. Un complexe hélicase/ADN polymérase permet ensuite la synthèse de l’ADN du brin intérieur, de façon circulaire, un type de synthèse nommé RCA (
rolling circle amplification
). La nouvelle séquence synthétisée est linéaire et peut être répliquée à son tour de façon discontinue. La RCA s’effectue plusieurs fois. Il en résulte la formation de plusieurs copies du génome qui fusionnent ensemble et forment de longs concatémères. Les génomes des Herpèsvirus étant riches en séquences répétées, des structures secondaires peuvent être présentes et, par conséquent, activer la recombinaison homologue afin de continuer la réplication. Certains Herpèsvirus augmentent ainsi la RH lors de leur réplication, comme HSV-1 [
14
,
15
] et un a-Herpèsvirus aviaire, le virus de la maladie de Marek (ou MDV) [
16
]. Le génome de ce dernier virus présente à ses extrémités des séquences télomériques répétées (TMR) et code UL12, une exonucléase virale, ainsi qu’UL29, homologue à RAD52, qui lie l’ADNsb [
16
]. Ces deux protéines sont essentielles à la réplication du virus en permettant des événements de
single-stranded annealing
. Le génome des HHV-6A/B possède également des TMR à chaque extrémité. Récemment, il a été montré que la protéine U70 du HHV-6A, orthologue d’UL12, augmente la fréquence d’événements de SSA, sans toutefois que cela soit comparable aux niveaux atteints avec l’UL12 [
17
]. L’effet de l’infection, ainsi que celui de la diminution de l’expression d’U70 sur la SSA, n’ont toutefois pas été examinés dans cette étude. Notre équipe a récemment montré que, lors de l’infection par les HHV-6A/B, la protéine du complexe
shelterin
, TRF2, est recrutée au niveau des séquences TMR virales [
18
]. Il est concevable que la présence de TRF2 au niveau des TMR des HHV-6A/B protège ces derniers de la DDR, mais aussi rétablisse l’arrêt de la fourche de réplication qui résulte de la présence des structures secondaires. À noter qu’un virus HHV-6A dont les TMR ont été éliminées se réplique beaucoup plus rapidement et génère plus de virions qu’un virus sauvage [
19
]. Comme pour le HHV-6A, TRF2 est également importante pour l’EBV : en se liant à l’OriR du génome de l’EBV, elle engage le complexe de pré-réplication/réplication [
20
]. Puisque certaines protéines du complexe
shelterin
jouent un rôle dans le recrutement de protéines impliquées dans la réplication et la réparation de l’ADN, il n’est pas surprenant de retrouver les génomes herpétiques près des télomères.
Les télomères, site de l’intégration virale
Outre le fait qu’ils sont les sites d’intégration du virus lors de l’infection, les télomères sont également la niche de latence pour les HHV-6A/B et le MDV. Chez le poulet, le MDV infecte les lymphocytes B, qui constituent la niche de la phase lytique du virus et ce qui conduit à la génération de lymphomes ; il infecte ensuite les lymphocytes T CD4
+
dans lesquels il devient latent, en intégrant son génome dans les télomères de ces cellules [
21
]. L’intégration du MDV n’est pas spécifique d’un chromosome : le génome du MDV a été détecté dans 15 chromosomes différents par des techniques de FISH (
fluorescent in situ hybridization
)
2,
et de PFGE (
pulse-field gel electrophoresis
)
3
, mais sa localisation est toujours télomérique.
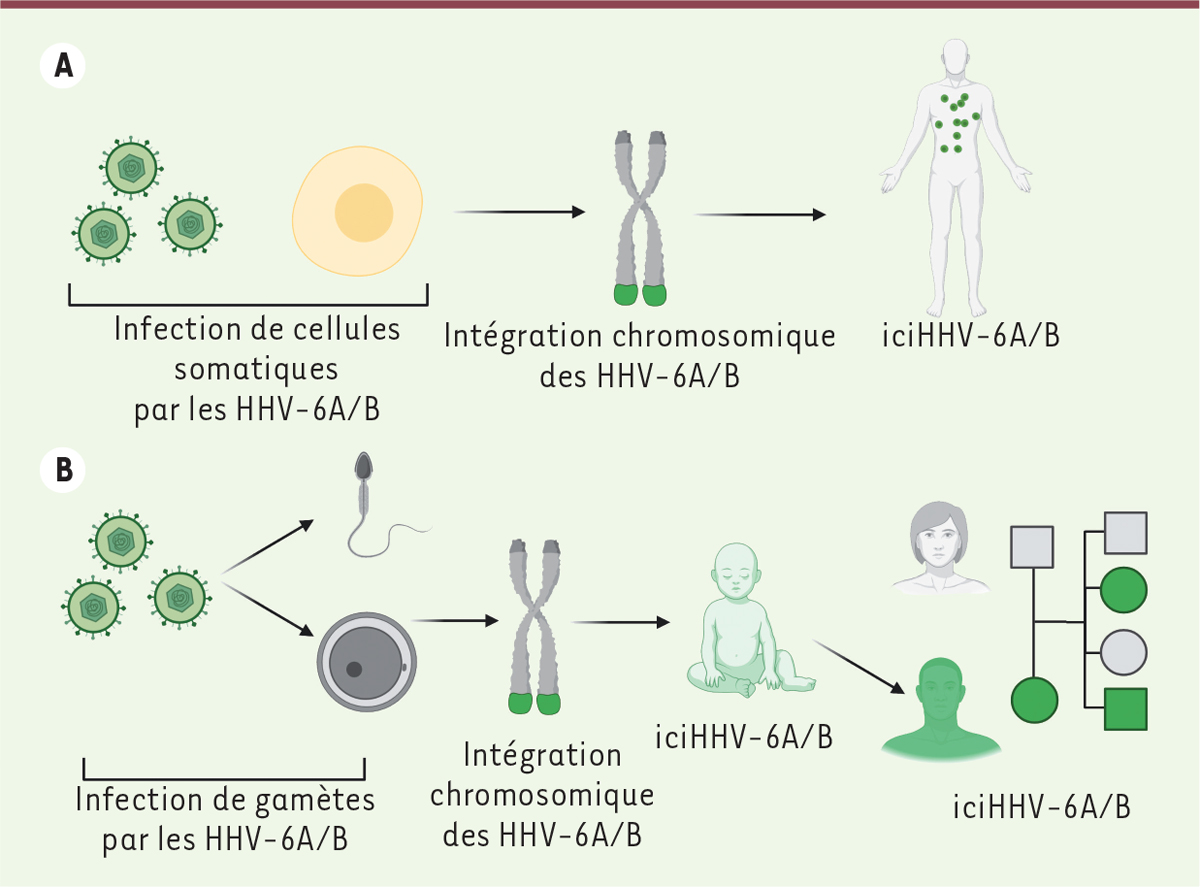
Chez l’homme, l’intégration des HHV-6A/B peut survenir à la suite de l’infection des cellules somatiques ou germinales,
via
les récepteurs cellulaires CD46, GP96 (
glycoprotein 96
) ou CD134, pour HHV-6B [
22
–
24
]. Le modèle hypothétique proposé suggère que la fécondation d’une cellule reproductrice ayant intégré le génome des HHV-6A/B sera à l’origine d’un embryon dont l’enfant aura une copie du génome intégré dans chaque cellule de son corps. Les études montrent que le virus intégré sera transmis à 50 % de la descendance sur un mode mendélien (cette condition est connue sous le terme
inherited chromosomally-integrated HHV-6A/B
[iciHHV-6A/B])
(
Figure 3
)
. À l’échelle mondiale, la prévalence des HHV-6A/B hérités est de 1,1 % ; la plus forte prévalence d’iciHHV-6A/B se situe en Écosse, avec un pourcentage d’intégration pouvant atteindre jusqu’à 4 % de la population [
25
,
26
]. Dans l’ensemble de l’Europe, la prévalence des iciHHV-6A/B se situe entre 0,5 et 3 % [
27
]. Aux États-Unis et au Canada, elle est respectivement de 1 à 1,5 % et de 0,6 % [
28
]. La répartition entre HHV-6A et HHV-6B intégrés est d’environ 40 % pour HHV-6A et 60 % pour HHV-6B.
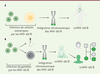 | Figure 3.
Modèles de l’intégration chromosomique des Herpèsvirus humains 6A/B. A.
Intégration des HHV-6A/B suivant l’infection de cellules somatiques. L’individu possède quelques cellules avec une copie du génome de HHV-6A/B.
B.
Intégration des HHV-6A/B dans une cellule germinale fécondée après son infection. La cellule fécondée mène à la naissance d’un enfant avec une copie du génome de HHV-6A/B dans toutes les cellules. Cet individu transmet la forme intégrée de HHV-6A/B à 50 % de sa descendance. ciHHV-6A/B :
chromosomally integrated HHV-6A/B
; iciHHV-6A/B :
inherited chromosomally integrated HHV-6A/B
.
|
Comme pour le MDV chez le poulet, la forme intégrée et héritée iciHHV-6A/B représenterait un mode de latence du virus chez l’homme. En effet, la forme épisomale du génome, qui est indicative d’un état de latence conventionnel pour les virus, n’a pas été détectée pour les HHV-6A/B. Bien que l’intégration partielle de génomes ait été observée avec d’autres Herpèsvirus, celle-ci est en général le résultat d’une exposition à des génomes viraux endommagés, dont seuls des fragments sont intégrés. Toutefois, l’EBV est capable d’intégrer son génome linéaire complet dans les chromosomes humains [
29
]. L’infection par l’EBV est généralement asymptomatique chez le jeune enfant. Les manifestations symptomatiques de l’EBV apparaissent ensuite, à l’adolescence ou à l’âge adulte. Elles sont associées à la mononucléose infectieuse et au développement de tumeurs : lymphome de Burkitt, lymphome non-Hodgkinien et carcinome nasopharyngé. Bien que l’EBV possède des protéines qui lui confèrent ce pouvoir oncogénique, l’intégration de son génome pourrait être un facteur additionnel conduisant au développent de ces cancers. En effet, l’étude la plus récente concernant l’intégration de l’EBV a identifié dans les cellules de lignées infectées par l’EBV (cellules Raji, issues de cellules d’un lymphome de Burkitt, et C666-1, issues de cellules d’un carcinome nasopharyngé), 909 sites uniques d’insertion [
30
]. Ces insertions sont une source d’instabilités génomique pouvant être responsable d’une accélération de la tumorigenèse des cellules. À ce jour, la réactivation de la forme intégrée d’EBV n’a cependant toujours pas été documentée [
31
]. Puisque l’EBV semble ne tirer aucun avantage de l’intégration de son génome, cet événement pourrait être la résultante de la présence de facteurs cellulaires à un moment précis de l’infection, conduisant le virus à s’intégrer dans différents sites sur divers chromosomes. Les conséquences de l’intégration d’EBV restent encore peu comprises.
D’autres types de virus, comme les AAV (
adeno-associated virus
) s’intègrent spécifiquement dans un chromosome, où ils demeurent latents (0,1 % des AAV s’intègrent dans le chromosome 19 [19q13.4]) [
32
]. Le virus de l’immunodéficience humaine (VIH), comme tous les rétrovirus, requiert l’intégration de son génome pour effectuer son cycle infectieux. Au contraire de ces deux derniers virus, dont la réplication nécessite l’action de l’ADN pol cellulaire quand ils sont sous leur forme intégrée, il a été suggéré que les génomes du MDV et des HHV-6A/B devaient être excisés des télomères pour qu’ils soient circularisés afin de débuter la RCA. Plusieurs études ont confirmé cette hypothèse dans des modèles cellulaires, renforçant l’idée que l’intégration chromosomique est un modèle alternatif de latence chez les HHV-6A/B [
33
,
34
]. Des 83 génomes d’Herpèsvirus séquencés à ce jour, 17 possèdent des séquences qui s’apparentent aux télomères, mais leur intégration n’a pas été étudiée [
21
]. Chez les b-Herpèsvirus, l’Herpèsvirus humain 7 (HHV-7) possède des TMR aux extrémités de son génome, mais les études n’ont, jusqu’à présent, identifié aucun patient avec une charge virale élevée, compatible avec une intégration. Davantage de criblages restent donc à être effectués.
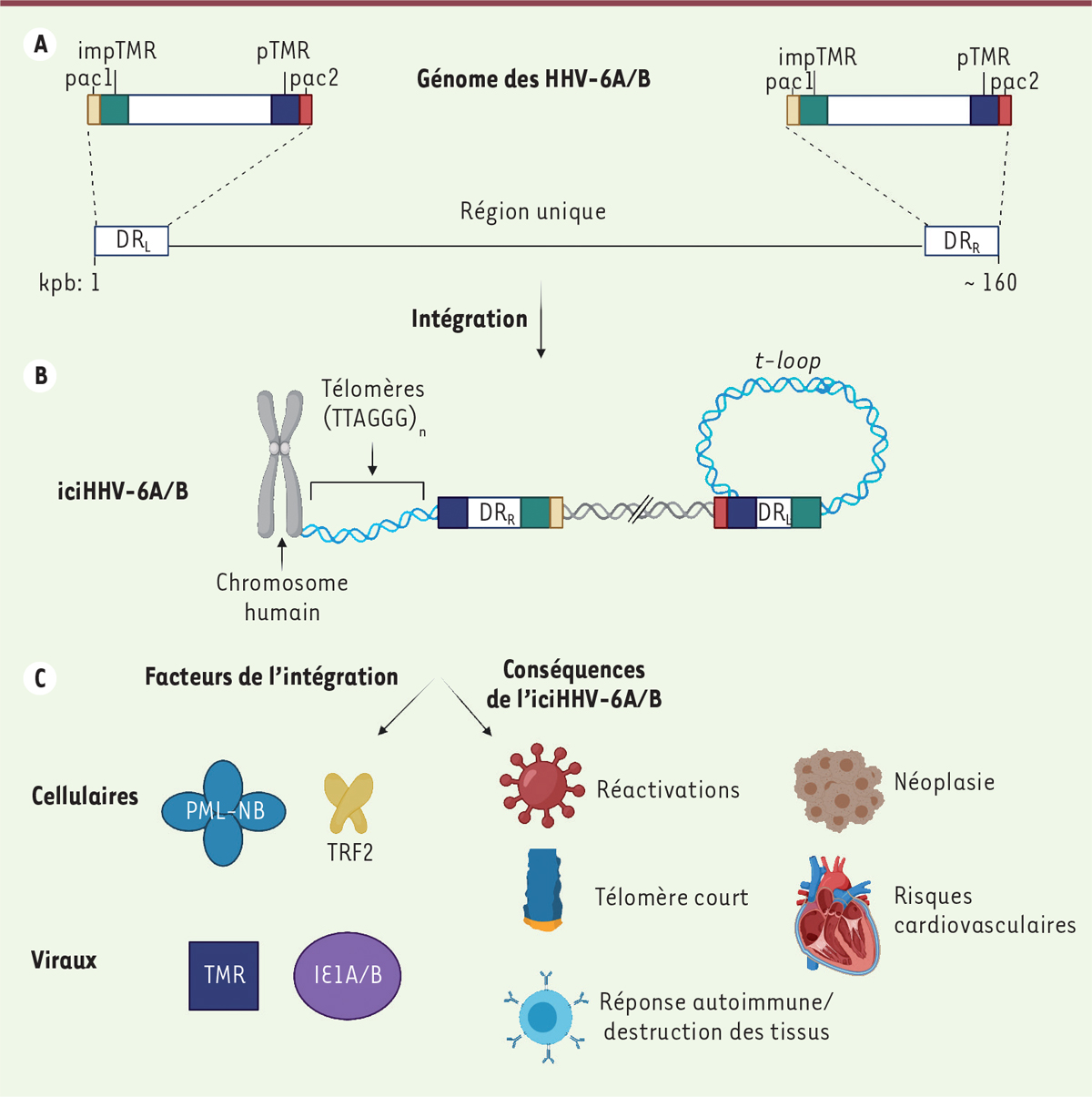
Mécanismes d’intégration aux télomères
Les mécanismes liés à l’intégration des virus MDV et HHV-6A/B demeurent encore incompris
(
Figure 4A
,
B
)
. En générant des mutants du virus MDV dont les TMR ont été retirées du génome viral (∆TMR), il a été montré que celles-ci étaient essentielles pour l’intégration du virus, et donc pour l’établissement de sa latence [
35
]. Un virus HHV-6A∆TMR a également été produit et ses capacités d’intégration ont été, comme pour le MDV, altérées [
19
]. Ces données suggèrent que le mécanisme d’intégration serait relié à la recombinaison homologue avec les télomères de la cellule hôte. Puisque le MDV possède des protéines qui s’apparentent à celles impliquées dans la SSA, UL12 et UL29, il pourrait ainsi tirer avantage de cette voie cellulaire de recombinaison pour s’intégrer. Pour les HHV-6A/B, une étude a proposé que les protéines U70, orthologue d’UL12 de MDV, et U41, homologue d’ICP8 de HSV-1, ne sont pas responsables des événements conduisant à l’intégration de ces virus [
17
]. L’importance d’U41 et d’U70 a néanmoins été évaluée dans cette étude en utilisant des shARN (
small hairpin RNA
), dont la capacité d’inhibition de l’expression des protéines virales n’était que partielle. D’autres approches sont certainement nécessaires pour évaluer sans équivoque le rôle de ces protéines virales dans l’intégration des HHV-6A/B, par exemple à l’aide de virus défectifs. Il avait été anticipé que la protéine U94, spécifiques des HHV-6A/B, et qui possède des activités d’exonucléase et d’hélicase, puisse jouer un rôle dans l’intégration virale [
36
]. Or, un mutant de HHV-6A n’exprimant pas U94 s’intègre tout aussi efficacement qu’un virus sauvage, indiquant que cette protéine n’est pas requise pour l’intégration du virus [
37
].
 | Figure 4.
Formats des génomes HHV-6A/B intégrés et mécanismes et maladies possibles associés. A.
Schéma générique du génome des HHV-6A/B. La région unique contenant les gènes viraux est flanquée par des répétitions directes (DR) à gauche (DR
L
) et à droite (DR
R
). Les DR sont composées, de 5’ vers 3’, d’une séquence pac1, de séquences télomériques imparfaites (impTMR), d’une région codante du DR, de répétions télomérique parfaites (TMR) et d’une séquence pac2. Les séquences pac1 et pac2 servent à l’empaquetage et l’encapsidation d’une molécule de génome HHV-6A/B dans un virion. Le génome et les éléments ne sont pas dessinés à l’échelle.
B.
Forme intégrée du génome HHV-6A/B dans la région sub-télomérique d’un chromosome à partir du DR
R
. La séquence pac2 du DR
R
est perdue, ainsi que la séquence pac1 du DR
L
. La région impTMR du DR
L
est synthétisée par la télomérase et forme une
t-loop
pour protéger le chromosome. Le génome et les éléments ne sont pas dessinés à l’échelle.
C.
Possibles facteurs menant à l’intégration des HHV-6A/B et conséquences cliniques associées. La protéine cellulaire TRF2 et les PML-NB aident à l’intégration des HHV-6A/B. Du point de vue viral, les TMR sont essentiels pour l’intégration d’HHV-6A et les protéines IE1A/B pourraient être impliquées. Les formes iciHHV-6A/B peuvent mener à des réactivations et aux maladies associées à celles-ci. À long terme, l’iciHHV-6A/B peut mener au raccourcissement prématuré du télomère intégré, à l’activation d’une réponse immunitaire auto-immune à la suite de l’expression continue de protéines virales, et ainsi mener à la destruction d’un tissu, à une instabilité génomique provoquant l’apparition de cellules néoplasiques et à un risque plus élevé de développer des maladies cardiovasculaires. PML-NB :
promyelocytic leukemia protein-nuclear bodies
; iciHHV-6A/B :
inherited chromosomally integrated HHV-6A/B
.
|
En plus d’avoir démontré l’association de TRF2 aux TMR des HHV-6A/B, nous avons souligné l’importance de cette protéine dans l’intégration de ces virus [
18
]. Récemment Timashev
et al
. [
6
] ont montré que TRF2 était importante pour le transfert du génome de l’ADNsb à l’ADNdb, mais ce rôle semble dépendre de la présence de partenaires d’interaction avec la protéine. Des études complémentaires sont donc nécessaires pour définir le rôle de TRF2 dans la recombinaison. PML (
promyelocytic leukemia protein
) est une autre protéine cellulaire qui affecte l’intégration des HHV-6A/B dans le génome de l’hôte. PML forme des corps nucléaires, les PML-NB (
PML nuclear bodies
), qui sont retrouvés dans les télomères endommagés et permettent leur réparation par des protéines impliquées dans la recombinaison homologue. La protéine précoce immédiate 1 (IE1) des HHV-6A/B s’associe à ces PML-NB, ce qui favorise son association aux télomères et, par conséquent, l’intégration du virus [
38
].
|
Conséquences de l’intégration des HHV-6A/B sur la biologie cellulaire et sur la biologie des télomères
Comprendre l’intégration des HHV-6A/B dans le génome de l’hôte et ses impacts cliniques est un sujet important pour la communauté scientifique
(
Figure 4C
)
. L’insertion d’un génome de 160 kpb dans une région importante pour l’homéostasie cellulaire et du génome ne peut en effet être sans conséquence pour l’hôte. Nous avons vu que l’intégration virale pourrait être un moyen utilisé par les HHV-6A/B pour être latents. La présence de séquences homologues aux extrémités du génome viral serait propice à la recombinaison homologue et mènerait ainsi à l’excision du génome complet des HHV-6A/B. Excisé, le génome peut se circulariser, se répliquer et produire de nouveaux virions. Cette possibilité a été montrée
in vitro
en stimulant des cellules de donneurs, porteuses d’iciHHV-6A/B, avec des inhibiteurs d’histones déacétylases (HDAC) ou avec le TPA (12-
O
-tétradécanoylphorbol-13-acétate) qui favorisent l’excision du virus.
In vivo,
un nourrisson porteur de la forme intégrée du virus iciHHV-6A, et atteint de déficit immunitaire combiné sévère lié à l’X (SCID), a vu son iciHHV-6A réactivé et a présenté un syndrome d’activation macrophagique [
39
]. L’utilisation d’antiviraux a rétabli momentanément l’état de santé de l’enfant. La guérison du déficit immunitaire du nourrisson a été obtenue par une greffe de cellules souches hématopoïétiques (CSH) à l’âge de sept mois. Mais, malgré l’utilisation prophylactique d’antiviraux, de hauts taux d’ADN et d’ARN viraux du HHV-6A ont été détectés, suggérant un autre épisode de réactivation de la forme iciHHV-6A à la suite de la greffe. La reconstruction du système immunitaire par la greffe a néanmoins permis d’abaisser les titres viraux d’HHV-6A. La forme iciHHV-6A/B peut également mener à la réactivation des HHV-6A/B à l’occasion de greffes de CSH ou de greffes d’organes solides. Cela a été le cas récemment chez un enfant ayant subi une greffe de foie [
40
] ; des études moléculaires ont déterminé que c’était le greffon qui était porteur de l’iciHHV-6B et qui était à l’origine de la réactivation virale. Un autre cas de réactivation de la forme iciHHV-6A présente dans le greffon a également été observé lors d’une greffe de rein chez un homme de 69 ans [
41
]. Ces réactivations ont été la source de graves complications cliniques et ont montré l’importance de dépister les greffons infectés
a priori
afin d’éviter d’infecter les receveurs et de réactiver les virus. L’excision du génome des HHV-6A/B des télomères à des fins prophylactiques signifierait que le télomère du chromosome de la cellule deviendrait plus court. Mais les télomères ayant intégré le génome viral sont déjà généralement plus courts que ceux des autres chromosomes de la cellule [
33
]. Notons également que, cliniquement, un raccourcissement de télomères est associé au développement de maladies cardiovasculaires [
42
,
43
]. Les patients présentant une forme iciHHV-6A/B du virus sont trois fois plus à risque de souffrir d’angine de poitrine que les sujets non porteurs, de mêmes âges [
28
]. Ces atteintes cardiovasculaires sont documentées : un enfant a succombé à une insuffisance cardiaque [
44
], et certains patients ont été atteints de myocardite [
45
]. Les télomères de certains chromosomes, tels que 6p, 12p, 17q, 16q, 21p, 3p-q, 9p-q, sont propices à être plus courts et certains d’entre eux ont été associés à ces maladies. L’intégration des HHV-6A/B a été détectée dans les chromosomes 9q, 10q, 13q, 17q,18q, 19q et 22q. Dans les populations nord-américaines et européennes, l’intégration des HHV-6A/B se produit dans différents chromosomes, mais elle est essentiellement observée dans les télomères du chromosome 17p. Dans la population asiatique, l’intégration des HHV-6A/B est retrouvée principalement dans le chromosome 22q [
46
–
48
]. Que la surreprésentation des virus intégrés concerne les chromosomes 17p (en Europe et en Amérique du Nord) et 22q (en Asie) soit due à des événements d’intégration ancestraux sélectionnés au cours de l’évolution ou à l’action de facteurs cellulaires ou viraux favorisant l’intégration dans certains chromosomes, reste une question non résolue, de même que le mécanisme précis de l’intégration. La présence de génomes viraux intégrés pourrait représenter une source additionnelle d’instabilité génomique.
Enfin, les iciHHV-6A/B pourraient affecter les transcrits d’ARN non codant TERRA, dont la modification est liée aux raccourcissements des télomères [
49
,
50
]. La transcription des TERRA peut produire des ARN d’une longueur de 9 kpb, en fonction de la longueur moyenne des télomères, et ainsi permettre la transcription de gènes viraux situés près du site d’intégration. Cela permettrait la traduction des protéines virales qui pourraient influencer la réactivation du virus ou initier une réponse immunitaire [
51
]. L’expression des protéines virales IE1 et U100 a été détectée dans plusieurs tissus de sujets porteurs d’iciHHV-6A/B et les sujets porteurs d’iciHHV-6A/B développent une importante réponse immunitaire humorale contre IE1, suggérant que l’expression spontanée de protéines virales à partir du génome intégré pourrait mener à la destruction des tissus. La pré-éclampsie et les avortements spontanés, deux maladies causées par la destruction des tissus par le système immunitaire, ont été récemment associés aux iciHHV-6A/B [
52
,
53
].
|
La particularité qu’ont les HHV-6A/B d’intégrer leur génome dans les télomères des chromosomes de leur hôte fait de ces virus des virus uniques parmi les Herpèsvirus humains. Le sujet est évidemment complexe. Bien que plusieurs mécanismes moléculaires de cette intégration soient désormais mieux compris, il n’en demeure pas moins que nous ne commençons qu’à effleurer la compréhension des possibles conséquences biologiques de l’intégration de ces virus. Considérant une prévalence d’environ 1 %, l’étude des sujets porteurs d’iciHHV-6A/B requiert un accès à des banques de tissus et à des données médicales d’envergure. On peut espérer que, du fait des avancées de la bio-informatique, des analyses de données massives (
Big Data
) et du partage de données et d’échantillons entre chercheurs au niveau international, les impacts biologiques de l’intégration des HHV-6A/B pourront être identifiés à moyen terme.
|
Les auteurs déclarent n’avoir aucun lien d’intérêt concernant les données publiées dans cet article.
|
Footnotes |
1.
Readhead
B
,
Haure-Mirande
JV
,
Funk
CC
,
et al.
Multiscale Analysis of Independent Alzheimer’s Cohorts Finds Disruption of Molecular, Genetic, and Clinical Networks by Human Herpesvirus.
.
Neuron.
2018;
;
99
:
:64.
–
82.e7
.
2.
Nacheva
EP
,
Ward
KN
,
Brazma
D
,
et al.
Human herpesvirus 6 integrates within telomeric regions as evidenced by five different chromosomal sites.
.
J Med Virol.
2008;
;
80
:
:1952.
–
1958
.
3.
Moyzis
RK
,
Buckingham
JM
,
Cram
LS
,
et al.
A highly conserved repetitive DNA sequence, (TTAGGG)(n), present at the telomeres of human chromosomes.
.
Proc Natl Acad Sci USA.
1988;
;
85
:
:6622.
–
6626
.
4.
Griffith
JD
,
Comeau
L
,
Rosenfield
S
,
et al.
Mammalian telomeres end in a large duplex loop.
.
Cell.
1999;
;
97
:
:503.
–
514
.
5.
de Lange
T.
.
Shelterin-Mediated Telomere Protection.
.
Annu Rev Genet.
2018;
;
52
:
:223.
–
247
.
6.
Timashev
LA
,
Lange
T
De
.
Characterization of t-loop formation by TRF2.
.
Nucleus.
2020;
;
11
:
:164.
–
77
.
7.
Shay
JW
,
Wright
WE
.
Telomeres and telomerase: three decades of progress.
.
Nat Rev Genet.
2019;
;
20
:
:299.
–
309
.
8.
Capper
R
,
Britt-Compton
B
,
Tankimanova
M
,
et al.
The nature of telomere fusion and a definition of the critical telomere length in human cells.
.
Genes Dev.
2007;
;
21
:
:2495.
–
2508
.
9.
Takai
H
,
Smogorzewska
A
,
de Lange
T
.
DNA damage foci at dysfunctional telomeres.
.
Curr Biol.
2003;
;
13
:
:1549.
–
1556
.
10.
O’Sullivan
RJ
,
Karlseder
J
.
Telomeres: Protecting chromosomes against genome instability.
.
Nat Rev Mol Cell Biol.
2010;
;
11
:
:171.
–
181
.
11.
Doksani
Y
,
Wu
JY
,
de Lange
T
,
et al.
Super-resolution fluorescence imaging of telomeres reveals TRF2-dependent T-loop formation.
.
Cell.
2013;
;
155
:
:345.
.
12.
Kramara
J
,
Osia
B
,
Malkova
A
.
Break-Induced Replication: The Where, The Why, and The How.
.
Trends Genet.
2018;
;
34
:
:518.
–
531
.
13.
Muylaert
I
,
Elias
P
.
Knockdown of DNA ligase IV/XRCC4 by RNA interference inhibits herpes simplex virus type I DNA replication.
.
J Biol Chem.
2007;
;
282
:
:10865.
–
10872
.
14.
Schumacher
AJ
,
Mohni
KN
,
Kan
Y
,
et al.
The HSV-1 Exonuclease, UL12, Stimulates Recombination by a Single Strand Annealing Mechanism.
.
PLoS Pathog.
2012;
;
8
:
:e1002862.
.
15.
Reuven
NB
,
Willcox
S
,
Griffith
JD
,
et al.
Catalysis of strand exchange by the HSV-1 UL12 and ICP8 proteins: Potent ICP8 recombinase activity is revealed upon resection of dsDNA substrate by nuclease.
.
J Mol Biol.
2004;
;
342
:
:57.
–
71
.
16.
Previdelli
RL
,
Bertzbach
LD
,
Wight
DJ
,
et al.
The role of Marek’s disease virus UL12 and UL29 in DNA recombination and the virus lifecycle.
.
Viruses.
2019
;
:11.
.
17.
Darren
J
,
Wight
J
,
Sanyal
A
,
et al.
Viral Proteins U41 and U70 of Human Herpesvirus 6A Are Dispensable for Telomere Integration.
.
Viruses.
2018;
;
10
:
:656.
.
18.
Gilbert-Girard
S
,
Gravel
A
,
Collin
V
,
et al.
Role for the shelterin protein TRF2 in human herpesvirus 6A/B chromosomal integration.
.
PLoS Pathog.
2020;
;
16
:
:e1008496.
.
19.
Wallaschek
N
,
Sanyal
A
,
Pirzer
F
,
et al.
The Telomeric Repeats of Human Herpesvirus 6A (HHV-6A) Are Required for Efficient Virus Integration.
.
PLoS Pathog.
2016;
;
12
:
:1.
–
15
.
20.
Atanasiu
C
,
Deng
Z
,
Wiedmer
A
,
et al.
ORC binding to TRF2 stimulates OriP replication.
.
EMBO Rep.
2006;
;
7
:
:716.
–
721
.
21.
Osterrieder
N
,
Wallaschek
N
,
Kaufer
BB
.
Herpesvirus genome integration into telomeric repeats of host cell chromosomes.
.
Annu Rev Virol.
2014;
;
1
:
:215.
–
235
.
22.
Santoro
F
,
Kennedy
PE
,
Locatelli
G
,
et al.
CD46 is a cellular receptor for human herpesvirus 6.
.
Cell.
1999;
;
99
:
:817.
–
827
.
23.
Ma
J
,
Jia
J
,
Jiang
X
,
et al.
gp96 Is Critical for both Human Herpesvirus 6A (HHV-6A) and HHV-6B Infections.
.
J Virol.
2020;
;
94
:
:e00311.
–
20
.
24.
Tang
H
,
Serada
S
,
Kawabata
A
,
et al.
CD134 is a cellular receptor specific for human herpesvirus-6B entry.
.
Proc Natl Acad Sci USA.
2013;
;
110
:
:9096.
–
9099
.
25.
Hoe
NL
,
Tuke
PW
,
Tedder
RS
,
et al.
The prevalence of chromosomally integrated human herpesvirus 6 genomes in the blood of UK blood donors.
.
J Med Virol.
2007;
;
79
:
:45.
–
51
.
26.
Clark
DA
.
Clinical and laboratory features of human herpesvirus 6 chromosomal integration.
.
Clin Microbiol Infect.
2016;
;
22
:
:333.
–
339
.
27.
Godet
AN
,
Soignon
G
,
Koubi
H
,
et al.
Presence of HHV-6 genome in spermatozoa in a context of couples with low fertility: What type of infection?
.
Andrologia.
2015;
;
47
:
:531.
–
535
.
28.
Gravel
A
,
Dubuc
I
,
Morissette
G
,
et al.
Inherited chromosomally integrated human herpesvirus 6 as a predisposing risk factor for the development of angina pectoris.
.
Proc Natl Acad Sci USA.
2015;
;
112
:
:8058.
–
8063
.
29.
Matsuo
T
,
Heller
M
,
Petti
L
,
et al.
Persistence of the entire epstein-barr virus genome integrated into human lymphocyte DNA.
.
Science.
1984;
;
226
:
:1322.
–
1325
.
30.
Xiao
K
,
Yu
Z
,
Li
X
,
et al.
Genome-wide analysis of Epstein-Barr Virus (EBV) integration and strain in C666–1 and Raji cells.
.
J Cancer.
2016;
;
7
:
:214.
–
224
.
31.
Bernasconi
M
,
Berger
C
,
Sigrist
JA
,
et al.
Quantitative profiling of housekeeping and Epstein-Barr virus gene transcription in Burkitt lymphoma cell lines using an oligonucleotide microarray.
.
Virol J.
2006;
;
3
:
:43.
.
32.
McCarty
DM
,
Young
SM
,
Samulski
RJ
.
Integration of adeno-associated virus (AAV) and recombinant AAV vectors.
.
Annu Rev Genet.
2004;
;
38
:
:819.
–
845
.
33.
Huang
Y
,
Hidalgo-Bravo
A
,
Zhang
E
,
et al.
Human telomeres that carry an integrated copy of human herpesvirus 6 are often short and unstable, facilitating release of the viral genome from the chromosome.
.
Nucleic Acids Res.
2014;
;
42
:
:315.
–
327
.
34.
Arbuckle
JH
,
Pantry
SN
,
Medveczky
MM
,
et al.
Mapping the telomere integrated genome of human herpesvirus 6A and 6B.
.
Virology.
2013;
;
442
:
:3.
–
11
.
35.
Kaufer
BB
,
Jarosinski
KW
,
Osterrieder
N
.
Herpesvirus telomeric repeats facilitate genomic integration into host telomeres and mobilization of viral DNA during reactivation.
.
J Exp Med.
2011;
;
208
:
:605.
–
615
.
36.
Trempe
F
,
Gravel
A
,
Dubuc
I
,
et al.
Characterization of human herpesvirus 6A/B U94 as ATPase, helicase, exonuclease and DNA-binding proteins.
.
Nucleic Acids Res.
2015;
;
4444
:
:1.
–
15
.
37.
Wallaschek
N
,
Gravel
A
,
Flamand
L
,
et al.
The putative U94 integrase is dispensable for human herpesvirus 6 (HHV-6) chromosomal integration.
.
J Gen Viroly.
2016;
;
97
:
:1899.
–
1903
.
38.
Collin
V
,
Gravel
A
,
Kaufer
BB
,
et al.
The promyelocytic leukemia protein facilitates human herpesvirus 6B chromosomal integration, immediate-early 1 protein multiSUMOylation and its localization at telomeres.
.
PLoS Pathog.
2020;
;
16
:
:e1008683.
.
39.
Endo
A
,
Watanabe
K
,
Ohye
T
,
et al.
Molecular and virological evidence of viral activation from chromosomally integrated human herpesvirus 6A in a patient with X-linked severe combined immunodeficiency.
.
Clinl Infect Dis.
2014;
;
59
:
:545.
–
548
.
40.
Bonnafous
P
,
Phan
TL
,
Himes
R
,
et al.
Evaluation of liver failure in a pediatric transplant recipient of a liver allograft with inherited chromosomally integrated HHV-6B.
.
J Med Virol.
2020;
;
92
:
:241.
–
50
.
41.
Petit
V
,
Bonnafous
P
,
Fages
V
,
et al.
Donor-to-recipient transmission and reactivation in a kidney transplant recipient of an inherited chromosomally integrated HHV-6A: Evidence and outcomes.
.
Am J Transplant.
2020;
;
20
:
:3667.
–
72
.
42.
Chang
ACY
,
Chang
ACH
,
Kirillova
A
,
et al.
Telomere shortening is a hallmark of genetic cardiomyopathies.
.
Proc Natl Acad Sci U S A.
2018;
;
115
:
:9276.
–
9281
.
43.
Saliques
S
,
Zeller
M
,
Lorin
J
,
et al.
Telomere length and cardiovascular disease.
.
Arch Cardiovasc Dis.
2010;
;
103
:
:454.
–
459
.
44.
Das
BB
.
A neonate with acute heart failure: Chromosomally integrated human herpesvirus 6-associated dilated cardiomyopathy.
.
J Pediatr.
2015;
;
167
:
:188.
–
92.e1
.
45.
Kühl
U
,
Lassner
D
,
Wallaschek
N
,
et al.
Chromosomally integrated human herpesvirus 6 in heart failure: Prevalence and treatment.
.
Eur J Heart Fail.
2015;
;
17
:
:9.
–
19
.
46.
Tweedy
J
,
Spyrou
MA
,
Pearson
M
,
et al.
Complete genome sequence of germline chromosomally integrated human herpesvirus 6A and analyses integration sites define a new human endogenous virus with potential to reactivate as an emerging infection.
.
Viruses.
2016;
;
8
:
:19.
.
47.
Mouammine
A
,
Gravel
A
,
Dubuc
I
,
et al.
Rs73185306 C/T Is Not a Predisposing Risk Factor for Inherited Chromosomally Integrated Human Herpesvirus 6A/B.
.
J Infect Dis.
2020;
;
221
:
:878.
–
81
.
48.
Tweedy
J
,
Spyrou
MA
,
Hubacek
P
,
et al.
Analyses of germline, chromosomally integrated human herpesvirus 6A and B genomes indicate emergent infection and new inflammatory mediators.
.
J Gen Virol.
2015;
;
96
:
:370.
–
389
.
49.
Kreilmeier
T
,
Mejri
D
,
Hauck
M
,
et al.
Telomere transcripts target telomerase in human cancer cells.
.
Genes.
2016;
;
7
:
:46.
.
50.
Wang
C
,
Zhao
L
,
Lu
S
.
Role of TERRA in the regulation of telomere length.
.
International J Biol Sci.
2015;
;
11
:
:316.
–
323
.
51.
Peddu
V
,
Dubuc
I
,
Gravel
A
,
et al.
Inherited Chromosomally Integrated Human Herpesvirus 6 Demonstrates Tissue-Specific RNA Expression In Vivo That Correlates with an Increased Antibody Immune Response.
.
J Virol.
2019;
;
94
:
:e01418.
–
e01419
.
52.
Gaccioli
F
,
Lager
S
,
Goffau
MC
de
,
et al.
Fetal inheritance of chromosomally integrated human herpesvirus 6 predisposes the mother to pre-eclampsia.
.
Nat Microbiol.
2020;
;
5
:
:901.
–
8
.
53.
Miura
H
,
Kawamura
Y
,
Ohye
T
,
et al.
Inherited Chromosomally Integrated Human Herpesvirus 6 Is a Risk Factor for Spontaneous Abortion.
.
J Infect Dis.
2021;
;
223
:
:1717.
–
23
.
|