Les tissus et les cellules d’un organisme sont continuellement soumis à des forces mécaniques. Par exemple, les muscles et les neurones sont étirés pendant les mouvements corporels, le battement du cœur ou la respiration. Plus généralement, les cellules sondent constamment les propriétés mécaniques de leur microenvironnement, notamment à l’aide de structures adhésives et du cytosquelette. De plus, la formation et la stabilité de ces structures dépendent justement de propriétés mécaniques, comme la rigidité du milieu environnant ou les forces exercées sur les cellules. Cette mécanosensibilité cellulaire joue un rôle clé dans la vie de la cellule car elle contrôle sa migration, mais également sa prolifération ou sa mort, en particulier au cours du développement [ 1 ], et elle est impliquée dans diverses maladies, notamment le cancer [ 2 ].
Un nombre croissant d’assemblages macromoléculaires de la cellule sont aujourd’hui reconnus comme étant contrôlés par les signaux mécaniques provenant de l’environnement extracellulaire, du cytosquelette et de la membrane plasmique. Historiquement, les premières structures mécanosensibles décrites furent celles d’adhérence à la matrice extracellulaire dépendantes des protéines de type intégrine [ 1 ]. Sont ensuite venues s’y ajouter les structures d’adhérence entre cellules impliquant les cadhérines (cohésion des tissus épithéliaux), les kinétochores (division cellulaire), les cavéoles (signalisation intracellulaire), ou encore le noyau cellulaire (expression génique). Grâce à des manipulations mécaniques in vitro de protéines purifiées, une compréhension de la mécanosensibilité à l’échelle moléculaire a émergé. Plusieurs techniques innovantes ont été utilisées pour produire et mesurer des forces sur les protéines in vitro, et même dans les cellules, en particulier pour les protéines des structures d’adhérence et les protéines du cytosquelette [ 1 , 3 ]. Des techniques telles que la microscopie à force atomique, la microfluidique, ou les pinces optiques ou magnétiques ont permis de montrer que les forces exercées directement sur les protéines stabilisent ou déstabilisent leurs interactions, ou contrôlent leur état d’activation [ 1 ]. Ces études ont permis de montrer que la mécanosensibilité cellulaire repose sur la déformation et la réorganisation des protéines. Ainsi, des forces de traction appliquées par des pinces magnétiques à la taline purifiée, une protéine qui relie les intégrines au cytosquelette d’actine, déclenchent son dépliement, révélant ainsi des sites cachés de liaison pour une autre protéine interagissant avec l’actine [ 4 ].
On a supposé qu’un stress mécanique externe appliqué aux cellules était directement et instantanément transmis aux protéines au sein des structures mécanosensibles, comme les structures d’adhérence et le cytosquelette. Cependant, les techniques de résolution moléculaire utilisées jusqu’à présent pour étudier les processus biomécaniques étaient restreintes à l’analyse de la surface des cellules vivantes et ne permettaient pas de sonder les réponses mécaniques des protéines dans les structures macromoléculaires à l’intérieur des cellules ou confinées à l’interface de la cellule avec son environnement extracellulaire [ 1 , 3 ]. Il a donc fallu développer une méthode permettant d’appliquer des forces externes aux cellules tout en capturant la réponse mécanique des protéines individuelles au sein de structures cellulaires mécanosensibles.
Au cours de la dernière décennie, les techniques de microscopie de super-résolution (prix Nobel de chimie en 2014) [ 13 ] ( → ) et de traçage de protéines uniques ont révolutionné l’imagerie en sciences du vivant en permettant de résoudre les structures cellulaires à l’échelle nanométrique et en permettant d’enregistrer les mouvements des protéines dans les cellules [ 5 ]. La résolution spatiale d’un instrument optique tel qu’un microscope, c’est-à-dire la distance minimale à laquelle deux objets peuvent être distingués, est dictée par la diffraction de la lumière, qui se produit lorsque l’onde lumineuse rencontre un obstacle qui modifie sa propagation. En passant par l’ouverture d’un instrument optique, la lumière subit ainsi une diffraction résultant de l’interférence des ondes émises à cet endroit. Un objet ponctuel donne donc une image floue, appelée tache d’Airy. Si deux objets sont trop proches l’un de l’autre, les taches de diffraction se chevauchent, et il devient impossible de discerner ces objets. Dans le cas d’un microscope conventionnel, la résolution spatiale est de l’ordre de 250 nm. Dans le cas des techniques de microscopie de super-résolution, les résolutions varient de quelques dizaines de nanomètres a quelques nanomètres [ 5 ], ce qui permet d’étudier l’architecture et la dynamique des structures cellulaires à l’échelle des protéines (2-10 nm). Grâce à la microscopie de super-résolution, des études ont ainsi 1) montré que les protéines sont organisées dans l’espace en nano-domaines fonctionnels distincts, 2) découvert de nouvelles organisations protéiques, et 3) reconsidéré les assemblages macromoléculaires en termes dynamiques, notamment dans les axones des neurones, les synapses, les structures subcellulaires protrusives propulsées par l’actine, et les structures d’adhérence dépendantes des cadhérines et des intégrines [ 2 , 6 – 11 ].
(→) Voir le Repère Nobel de M. Moreau et P. Cochard, m/s n° 12, décembre 2014, page 1169
En combinant des étirements cellulaires sur des substrats élastiques avec les techniques de microscopie de super-résolution ( Figure 1A ) et de traçage de protéines uniques ( Figure 1B ), nous avons pu, pour la première fois, analyser la réponse mécanique de protéines individuelles [ 12 ]. Pour cela, en collaboration avec le groupe dirigé par Pierre Nassoy (physicien à l’institut d’optique d’Aquitaine), nous avons inventé un nouveau dispositif d’étirement cellulaire compatible avec les exigences optiques requises pour effectuer des acquisitions de super-résolution et de traçage de protéines uniques. Ce dispositif est par ailleurs compatible avec les principales familles de la microscopie de super-résolution. Celles fondées sur la détection de fluorophores individuels, i.e. le PALM ( photoactivation localization microscopy ), le STORM ( stochastic optical reconstruction microscopy ), le PAINT ( points accumulation for imaging in nanoscale topography ), et celles fondées sur la réduction de la taille de la tache d’Airy mentionnée plus haut, i.e. le STED ( stimulated emission depletion ), le RESOLFT ( reversible saturable opticaL fluorescence transitions ) [ 5 ]. L’application de forces mécaniques sur les cellules tout en utilisant ces techniques innovantes devait permettre de compléter les études précédentes visant à comprendre l’organisation dynamique des protéines au sein des cellules adhérant à des substrats passifs (par exemple le verre) [ 2 , 9 – 11 ]. Nous avons ainsi pu suivre la réponse immédiate de protéines individuelles aux forces d’étirement au sein de structures mécanosensibles dans leur contexte cellulaire [ 12 ], une avancée importante par rapport aux études in vitro sur la mécanosensibilité de protéines purifiées [ 4 ].
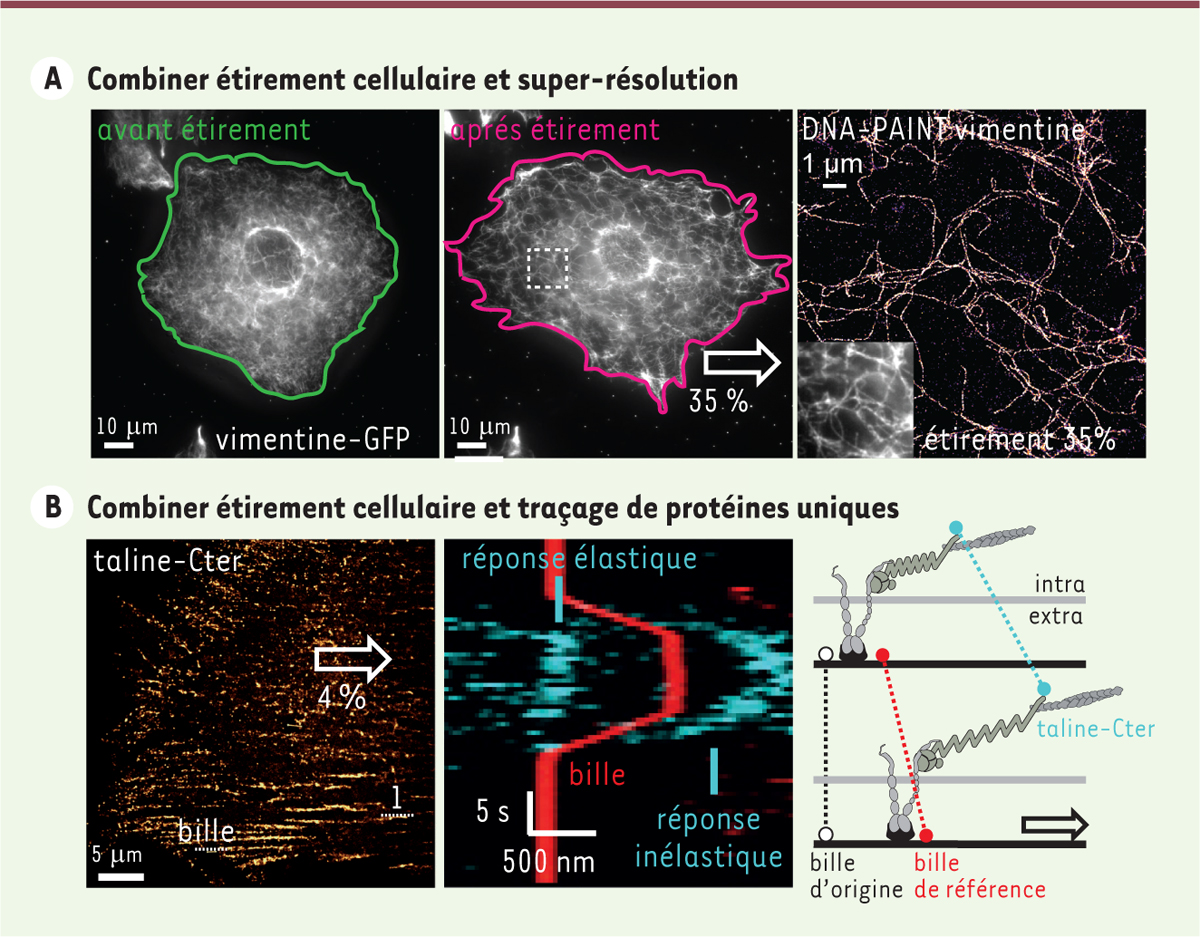
| Figure 1.
A. Combiner étirement cellulaire et microscopie de super-résolution.
Une cellule exprimant la vimentine, une protéine composant les filaments intermédiaires du cytosquelette, imagée par microscopie en fluorescence conventionnelle, avant (en vert, à gauche) et après (magenta, au milieu) un étirement cellulaire de forte amplitude (35 %), puis avec la microscopie de super-résolution (DNA-PAINT, à droite). L’image super-résolue de la région limitée par le trait en pointillé (au milieu) montre les filaments de vimentine après l’étirement cellulaire. On peut apprécier le gain de détail et de résolution spatiale dans cette image par rapport à l’image de la même zone obtenue par la microscopie conventionnelle en fluorescence (en insert, à droite), ce qui permet d’étudier les changements d’organisation des protéines du cytosquelette à l’échelle nanométrique.
B. Combiner étirement cellulaire et traçage de protéines uniques.
Réponse inélastique de la taline soumise à des forces d’étirement correspondant au dépliement de la protéine. Traçage de protéines uniques (taline) dans une cellule pendant un étirement cellulaire d’amplitude modérée (4 %, à gauche). Kymographe représentant horizontalement l’espace le long de la ligne 1, et verticalement, le temps permettant de décrire la réponse dynamique de la taline (en cyan) lors d’un cycle d’étirement du substrat suivi au moyen de billes adsorbées (en rouge). La taline peut présenter des réponses élastiques durant lesquelles elle suit le déplacement du substrat (forme en trapèze), ou des réponses inélastiques (forme parabolique) durant lesquelles elle continue son mouvement alors même que le substrat n’est plus étiré. Schéma illustrant ce dernier type de réponse avec la déformation de la taline (taline-Cter, en cyan) induite par l’étirement du substrat (points blancs et rouges), auquel la cellule est ancrée par les intégrines (points noirs).
|
Nous avons montré que lors d’un étirement cellulaire, les intégrines suivent le déplacement élastique du substrat. D’autres protéines connectées aux intégrines et au cytosquelette d’actine, comme la taline, ont un comportement inélastique car elles continuent à être déformées alors même que le substrat n’est plus étiré [ 12 ] ( Figure 1B ). Ces déformations moléculaires différées et amplifiées par rapport à la contrainte mécanique initiale sont déclenchées par un remodelage transitoire et actif de l’appareil contractile de la cellule, composé de filaments d’actine et de moteurs moléculaires du type myosine. Ces déformations protéiques, qui révèlent des sites cachés d’interaction moléculaire, permettent le recrutement de nouvelles protéines au sein des structures d’adhérence, par exemple la vinculine et la zyxine [ 12 ]. De plus, nous avons pu montrer que certaines réponses mécaniques ne se produisent que dans les structures d’adhérence précoces, et dépendent donc de l’état de maturation des structures mécanosensibles [ 12 ]. Ainsi, cette méthode permet d’analyser à l’échelle moléculaire, dans les cellules, à la fois la déformation des protéines et leur réorganisation à la suite de stimulus mécaniques externes.
Les méthodes conventionnelles de micro-rhéologie procèdent en moyennant l’ensemble des hétérogénéités, des inélasticités et des fluctuations temporelles à l’échelle moléculaire. En combinant la microscopie de super-résolution et le traçage de protéines uniques avec l’étirement des cellules, notre approche ouvre la voie à la nano-rhéologie de la cellule [ 12 ]. Nous avons découvert que la déformation des protéines des structures d’adhérence dépendantes des intégrines n’est pas nécessairement déclenchée par la transmission directe des forces mécaniques externes, mais peut résulter d’une réponse active et transitoire du cytosquelette d’actine, qui amplifie les stimulus mécaniques faibles pour déformer (taline) ou recruter (vinculine, zyxine) des protéines. Nos résultats indiquent que les événements initiaux de la mécanosensibilité cellulaire doivent se produire en quelques secondes, dans la fenêtre temporelle de cette amplification active locale. L’ensemble de ces résultats expérimentaux vient étayer des arguments théoriques fondés sur un modèle physique de la réponse mécanique de la cellule [ 12 ]. Enfin, cette nouvelle technique devrait également permettre d’analyser, à l’échelle moléculaire, le fonctionnement d’autres structures mécanosensibles de la cellule, depuis la membrane plasmique jusqu’au noyau de la cellule.