| |
| Med Sci (Paris). 38(1): 12–14. doi: 10.1051/medsci/2021237.Un long ARN non codant régule l’activité de la phénylalanine hydroxylase, l’enzyme responsable de la phénylcétonurie Arnaud Wiedemann,1,2 Chunru Lin,3 Abderrahim Oussalah,2,4 Bernard Namour,2,4 Élise Jeannesson,2,4 Jean-Louis Guéant,2,4 and François Feillet1,2* 1Centre de référence des maladies métaboliques, Service de pédiatrie, CHU Nancy 2Inserm U1256, Nutrition-génétique et exposition aux risques environnementaux (NGERE)
,
9 avenue de la forêt de Haye
,
CS 50184
,
54505Vandoeuvre-les-Nancy
,
France 3Department of Molecular and Cellular Oncology, The University of Texas MD Anderson Cancer Center
,
Houston
,
TX77030
,
États-Unis 4Laboratoire de biochimie et de biologie moléculaire, CHU Nancy MeSH keywords: Bioptérine, Humains, Phenylalanine 4-monooxygenase, Phénylcétonuries, ARN long non codant, génétique |
L’histoire de la phénylcétonurie (PCU) débute en 1934 [
1
] et, depuis sa description, les différents mécanismes qui sous-tendent la pathogenèse de cette maladie sont progressivement découverts, la rendant de plus en plus complexe. Après la découverte de l’enzyme responsable de cette maladie, la phénylalanine hydroxylase (PAH) [
2
], les déficits en son cofacteur (tétrahydrobioptérine ou BH4) ont été décrits dans les années 1970 [
3
] chez des patients atteints de ce que l’on a appelé à l’époque une hyperphénylalaninémie maligne, et, en 2017, la responsabilité de DNAJC12 (
DnaJ heat shock protein family (Hsp40) member C1
2), une molécule chaperon, a été mise en évidence [
4
].
La découverte de cette molécule chaperon semblait signer la fin de la compréhension des mécanismes moléculaires de la phénylcétonurie. Les études de génétique moléculaire réalisées chez les patients atteints de PCU retrouvaient en effet, dans 99 % [
5
,
6
] des cas, des mutations du gène
PAH
. Les quelques patients restants étaient, quant à eux, porteurs soit d’un déficit de synthèse ou de recyclage du cofacteur de l’enzyme, le BH4, soit de la molécule chaperon DNAJC12 [
7
]. Mais en août 2021, un article publié par Li
et al
. [
8
] de l’
Anderson Institute
, à Houston (États-Unis), a ouvert une nouvelle voie en montrant qu’un long ARN non codant (ARNlnc), appelé Pair chez la souris et Hulc pour son homologue chez l’homme, interagit avec la PAH et régule son activité.
|
Un long ARN non codant est impliqué dans la régulation de la phénylalaninémie
Actuellement, il est estimé que seuls 2 % du génome codent des protéines [
9
], mais une importante portion de l’ADN est cependant transcrite en ARN. La fonction de ces ARN non-codants n’est pas encore totalement élucidée [
10
] : ils peuvent en effet interagir avec des protéines, des peptides, des séquences d’ADN, des ARN messagers, voire avec des petites molécules [
10
]. Ces interactions influencent essentiellement le contrôle épigénétique de l’expression des gènes, leur transcription et la régulation des protéines qu’ils codent, mais aussi, et en particulier, la régulation de la réponse immunitaire.
L’équipe de Li
et al.
avait remarqué qu’un long ARN non codant (ARNlnc), appelé Pair, était particulièrement exprimé au niveau du foie et, pour comprendre sa fonction, ces auteurs ont créé une souris déficiente pour le gène codant cet ARNlnc. De façon tout à fait inattendue, les auteurs ont observé que cette souris a développé un phénotype comparable à celui que développent des souris dans des modèles murins de phénylcétonurie, à savoir, une hypopigmentation et un tableau neurologique sévère (retard de développement, épilepsie, microcéphalie). Ils ont ensuite pu mettre en relation ce phénotype particulier avec une hyperphénylalaninémie semblable à celle observée dans les autres modèles murins de PCU. Constatant que les taux plasmatiques des autres acides aminés n’étaient pas altérés par la déplétion de
Pair
, ils ont alors envisagé que le tableau clinique et biologique qu’ils observaient était bien en rapport avec une baisse d’activité de la phénylalanine hydroxylase (PAH) due au déficit de cet ARNlnc. En parallèle, les auteurs ont montré que cet ARNlnc avait un homologue chez l’homme, appelé
Hulc
.
|
Un long ARN non codant est impliqué dans la régulation de la phénylalaninémie
Des études de
pulldown
1
et d’immunoprécipitation réalisées sur des foies de souris ont montré que l’ARNlnc
Pair
s’associait à la PAH. Une analyse de tissus hépatiques humains provenant de deux donneurs sains a par ailleurs révélé que la PAH humaine était également associée à l’ARNlnc
HULC
(l’homologue de
PAIR
), et que sa présence était indispensable au bon fonctionnement de la PAH. Une analyse approfondie a ensuite montré que l’ARNlnc
Hulc/Pair
s’associe au domaine N-terminal de régulation de la PAH. Cette association stabiliserait probablement la conformation ouverte de la PAH, facilitant ainsi la catalyse de la Phe en Tyr par l’enzyme. Dans le tissu cérébral des souris
Pair
-/-
, une diminution de quantité de tyrosine hydroxylase a également été observée. Il n’y a pas eu d’études sur la tryptophane hydroxylase, mais ce résultat pose la question de savoir si cet ARNlnc agit uniquement sur la PAH ou s’il agit également sur les deux autres hydroxylases dont le BH4 est aussi le cofacteur, et pour lesquelles la DNAJC12 est également une protéine chaperon. Si cela était le cas, les déficits en
Pair
/
Hulc
entraîneraient donc un déficit global des trois hydroxylases, et non pas simplement de la PAH, ce qui pourrait expliquer le phénotype extrêmement sévère des souris
Pair
-/-
.
|
Un analogue de synthèse de
Pair
restore l’activité de la PAH (
Figure 1
)
Un analogue de l’ARNlnc
Pair
/
Hulc
a été synthétisé (
lncRNA mimics
).
In vivo,
administré aux souris
Pair
-/-
qui sont dépourvues de l’ARNlnc
Pair
, cet analogue a permis de restaurer l’activité de la PAH et de normaliser les taux plasmatiques de Phe.
In vitro,
des souches de bactéries transformées pour exprimer différents variants défectifs du gène codant la PAH identifiés chez les patients ont été cultivées en l’absence ou en présence de cet analogue de synthèse, et son interaction avec l’enzyme produite et son activité ont été examinées. Dans ces conditions, le
lncRNA mimics
a permis de restaurer l’activité enzymatique de la PAH chez certaines souches bactériennes exprimant un gène
PAH
présentant des mutations touchant le site d’interaction entre l’enzyme et l’ARNlnc. Le
lncRNA mimics
n’a par contre eu aucun effet sur d’autres souches portant des mutations de l’enzyme situées hors de ce site d’interaction. Le
lncRNA mimics
a été particulièrement efficace sur une souche porteuse de la mutation p.R408W (arginine en position 408 remplacée par un tryptophane) de la PAH, une mutation très sévère (à l’origine d’une activité résiduelle de la PAH inférieure à 2 % de l’activité normale [
11
]. Cette mutation est la plus fréquente dans les pays de l’est européen. Elle représente en effet 76 % des variants retrouvés chez les patients atteints de PCU en Lettonie, 63 % en Russie [
12
] et 57 % en Pologne [
13
]. Cette découverte apporte donc un espoir thérapeutique considérable pour ces patients chez lesquels le seul traitement actuel repose sur un régime alimentaire restrictif à vie, et dont on sait à quel point la poursuite et le suivi est difficile à l’âge adulte [
14
].
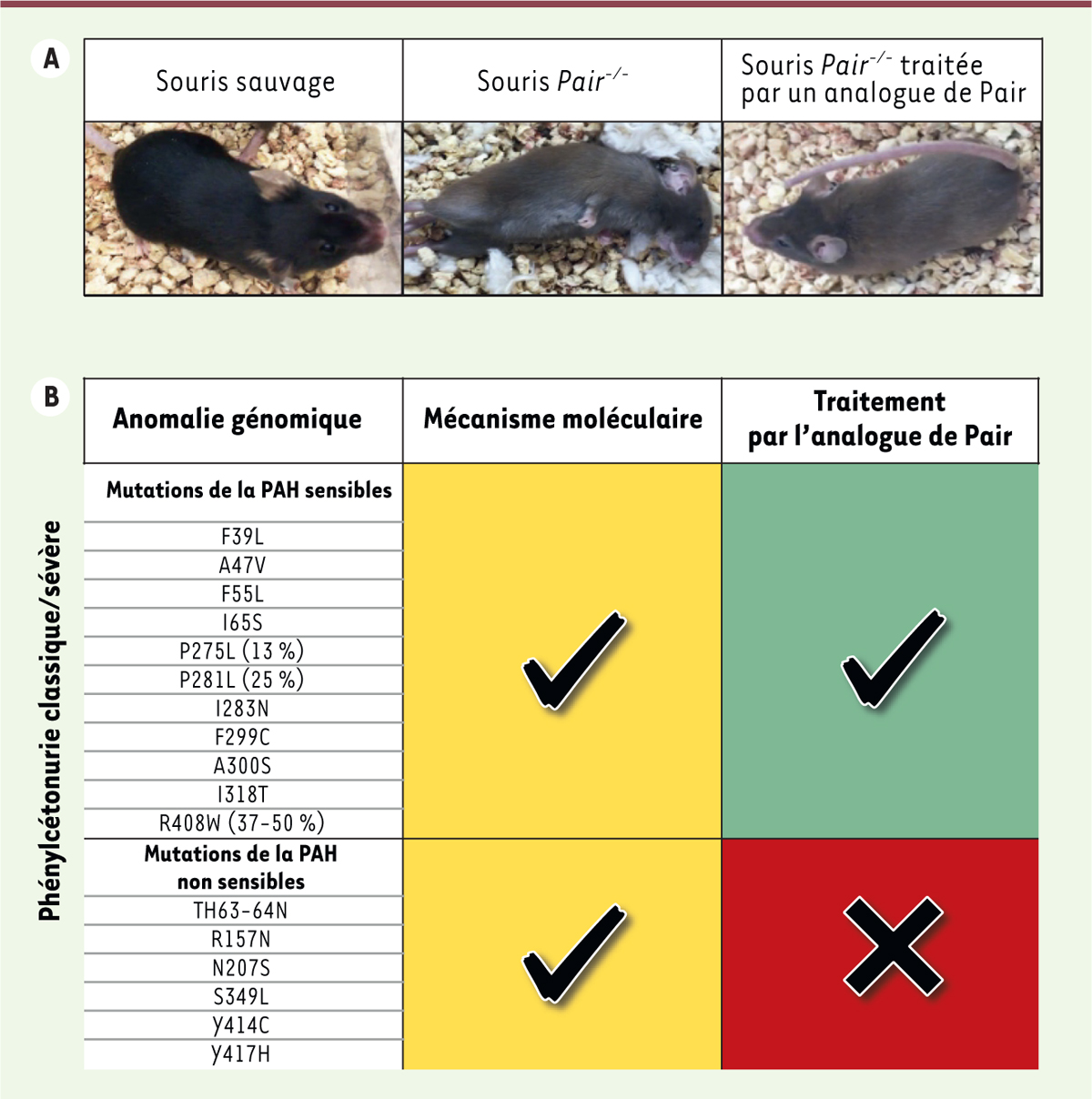
 | Figure 1.
Un long ARN non codant (ARNlnc) agit comme cible thérapeutique pour une maladie génétique humaine.
L’ARNlnc Hulc et son homologue chez la souris (Pair) modulent l’activité enzymatique de la phénylalanine hydroxylase (PAH), l’enzyme responsable de phénylcétonurie (PCU). L’administration d’un analogue ARNlnc permet de traiter la souris Pair-/-
(A)
. Dans des modèles cellulaires porteurs de mutations responsables de PCU, et pour lesquelles les mécanismes moléculaires impliqués sont connus, l’anologue ARNlnc permet de restaurer l’activité enzymatique de la PAH
(B)
. La réponse à ce traitement n’est toutefois possible que lorsque la mutation de l’enzyme modifie son interaction avec
Hulc
(mutations sensibles). Cet analogue ARNlnc pourrait ainsi constituer une option thérapeutique prometteuse pour les patients atteints de PCU présentant des mutations de
Hulc
ou des mutations de la PAH sensibles à ce traitement.
|
|
Plusieurs points à éclaircir
L’origine génétique de la grande majorité, sinon de la totalité, des patients atteints de PCU est désormais bien établie. Il est donc peu probable que certains d’entre eux présentent une maladie qui aurait pour origine uniquement un variant du gène
Hulc
. Par contre, il est très probable que ces variants influent sur l’activité de l’enzyme PAH, ce qui pourrait expliquer, au moins en partie, l’absence de corrélation entre génotype et phénotype que l’on observe chez certains patients présentant une PCU [
15
]. Une étude de l’action du
lncRNA mimics
sur les principaux variants connus de la PAH nécessite cependant d’être réalisée (selon un modèle similaire à celui utilisé pour déterminer la sensibilité des patients au BH4) [
16
]. En effet, seuls 17 variants ont été testés dans le travail relaté ici, alors que 1 282 variants du gène ont été décrits [
17
]. Il est également très important d’établir si cet ARNlnc agit uniquement sur la PAH ou s’il a également un effet sur l’ensemble des trois hydroxylases (Phé, Tyr et Trp), car s’il traversait la barrière hémato-encéphalique, il pourrait avoir un impact sur la synthèse de certains neurotransmetteurs (voies de la dopamine et de la sérotonine). Cela est d’autant plus important que si un tel traitement de la PCU par
IncRNA mimics
venait à voir le jour, il faudrait s’assurer de l’absence d’effets délétères de ce type de traitement sur le statut des neurotransmetteurs.
|
La publication de Li et
al
. qui décrit pour la première fois l’action d’un ARNlnc sur une enzyme responsable d’une maladie héréditaire du métabolisme, la phénylcétonurie, est donc une avancée majeure. La PCU, qui est la maladie métabolique la plus fréquente, est connue depuis plus de 70 ans, et ce qui paraissait une maladie simple (une enzyme déficitaire qui entraîne une accumulation de son substrat en amont, la phénylalanine) a vu sa physiopathologie se complexifier au cours du temps, avec les découvertes successives de son cofacteur, le BH4, puis de sa molécule chaperon, la DNAJC12. Le modèle rendant compte de la pathogenèse de la maladie vient encore de s’enrichir avec l’implication de l’ARNlnc
Hulc
dans la régulation de l’activité de l’enzyme. Mendes de Oliveira écrivait récemment dans le
New Engl J Med
que les maladies monogéniques sont cliniquement plus variables que ne le suggérait leur description initiale [
18
]. Au vu de cette nouvelle découverte, et si l’on prend en compte le fait que seuls 2 % du génome codent des protéines [
9
], on peut se demander si les maladies monogéniques « existeront » encore dans le futur.
|
Les auteurs déclarent n’avoir aucun lien d’intérêt concernant les données publiées dans cet article.
|
Footnotes |
1.
Folling
A.
Excretion of phenylpyruvic acid in urine as a metabolicnomaly in connection with imbecility.
.
Nord Med Tidskr.
1934;
;
8
:
:1054.
–
1059
.
2.
Jervis
GA
.
Studies on phenylpyruvic oligophrenia - The position of the metabolic error.
.
J Biol Chem.
1947;
;
169
:
:651.
–
656
.
3.
Smith
I
,
Clayton
BE
,
Wolff
OH
.
Letter: a variant of phenylketonuria.
.
Lancet.
1975;
;
1
:
:328.
–
329
.
4.
Anikster
Y
,
Haack
TB
,
Vilboux
T
,
et al.
Biallelic mutations in DNAJC12 cause hyperphenylalaninemia, dystonia, and intellectual disability.
.
Am J Hum genet.
2017;
;
100
:
:257.
–
266
.
5.
Jeannesson-Thivisol
E
,
Feillet
F
,
Chery
C
,
et al.
Genotype-phenotype associations in French patients with phenylketonuria and importance of genotype for full assessment of tetrahydrobiopterin responsiveness.
.
Orphanet J Rare Dis.
2015;
;
10
:
:158.
.
6.
Ho
G
,
Alexander
I
,
Bhattacharya
K
,
et al.
The molecular bases of phenylketonuria (PKU) in new south wales, australia: mutation profile and correlation with tetrahydrobiopterin (BH4) responsiveness.
.
JIMD Rep.
2014;
;
14
:
:55.
–
65
.
7.
Blau
N
,
Martinez
A
,
Hoffmann
GF
,
Thony
B
.
DNAJC12 deficiency: a new strategy in the diagnosis of hyperphenylalaninemias.
.
Mol Genet Metab.
2018;
;
123
:
:1.
–
5
.
8.
Li
Y
,
Tan
Z
,
Zhang
Y
,
et al.
A noncoding RNA modulator potentiates phenylalanine metabolism in mice.
.
Science.
2021;
;
373
:
:662.
–
73
.
9.
Lander
ES
,
Linton
LM
,
Birren
B
,
et al.
Initial sequencing and analysis of the human genome.
.
Nature.
2001;
;
409
:
:860.
–
921
.
10.
Kazimierczyk
M
,
Kasprowicz
MK
,
Kasprzyk
ME
,
Wrzesinski
J
.
Human long noncoding RNA interactome : detection, characterization and function.
.
Int J Mol Sci.
2020;
;
21
:
:1027.
.
11.
Zurfluh
MR
,
Zschocke
J
,
Lindner
M
,
et al.
Molecular genetics of tetrahydrobiopterin-responsive phenylalanine hydroxylase deficiency.
.
Hum Mutat.
2008;
;
29
:
:167.
–
175
.
12.
Sueoka
H
,
Moshinetsky
A
,
Nagao
M
,
Chiba
S
.
Mutation screening of phenylketonuria in the Far East of Russia.
.
J Hum Genet.
1999;
;
44
:
:368.
–
371
.
13.
Zygulska
M
,
Eigel
A
,
Dworniczak
B
,
et al.
Phenylketonuria in Poland : 66 % of PKU alleles are caused by three mutations.
.
Hum Genet.
1991;
;
88
:
:91.
–
94
.
14.
MacDonald
A
,
Gokmen-Ozel
H
,
van Rijn
M
,
Burgard
P
.
The reality of dietary compliance in the management of phenylketonuria.
.
J Inherit Metab Dis.
2010;
;
33
:
:665.
–
670
.
15.
Lindner
M
,
Haas
D
,
Mayatepek
E
,
et al.
Tetrahydrobiopterin responsiveness in phenylketonuria differs between patients with the same genotype.
.
Mol Genet Metab.
2001;
;
73
:
:104.
–
106
.
16.
Danecka
MK
,
Woidy
M
,
Zschocke
J
,
et al.
Mapping the functional landscape of frequent phenylalanine hydroxylase (PAH) genotypes promotes personalised medicine in phenylketonuria.
.
J Med Genet.
2015;
;
52
:
:175.
–
185
.
18.
Mendes de Oliveira
E
,
Keogh
JM
,
Talbot
F
,
et al.
Obesity-associated GNAS mutations and the melanocortin pathway.
.
N Engl J Med.
2021;
;
385
:
:1581.
–
92
.
|