| |
| Med Sci (Paris). 37(11): 981–983. doi: 10.1051/medsci/2021137.
Étude
in silico
de la communication intercellulaire avec ICELLNET
Lucile Massenet-Regad,1,2a Floriane Noël,1b and Vassili Soumelis1,3c 1Université de Paris, Inserm U976
,
75006Paris
,
France 2Université Paris-Saclay
,
91190Saint Aubin
,
France 3AP-HP, Hôpital Saint-Louis, Département d’immunologie-histocompatibilité
,
1 avenue Claude Vellefaux
,
75010Paris
,
France MeSH keywords: Communication cellulaire, Simulation numérique |
Comment les cellules communiquent-elles ?
Les cellules sont des systèmes dynamiques qui intègrent de nombreux signaux et échangent constamment de l’information avec les cellules qui les entourent. Ces échanges d’informations entre cellules leur permettent de s’organiser pour construire des tissus, des organes, et de se coordonner pour maintenir un état d’équilibre ou répondre à une situation menaçante. Pour communiquer, une cellule « émettrice » transmet des signaux d’information à une cellule « réceptrice ». Les informations émises sont généralement codées sous la forme de molécules solubles (
e.g
., cytokines, hormones) ou membranaires (
e.g
., molécules du complexe majeur d’histocompatibilité, points de contrôle immunitaire). Ces molécules se lient ensuite à leurs récepteurs spécifiques sur la cellule cible (formant des paires ligand-récepteur), ce qui peut déclencher une réponse cellulaire
(
Figure 1A
)
.
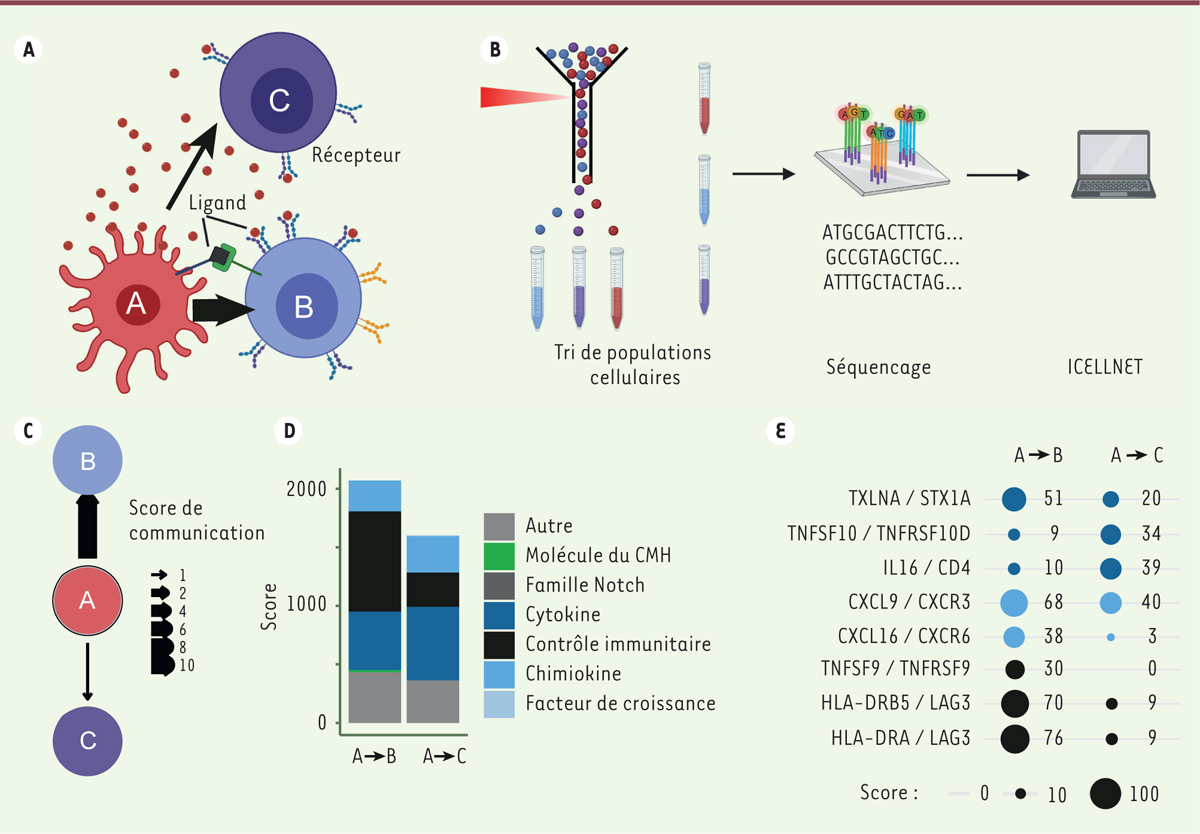
 | Figure 1.
Principe d’étude de la communication intercellulaire et utilisation d’ICELLNET. A.
Schéma créé par BioRender.com illustrant le contexte biologique d’une cellule dendritique (cellule A) communiquant avec deux lymphocytes T dans des états biologiques différents (cellules B et C).
B.
Schéma créé par BioRender.com des étapes expérimentales nécessaires à l’obtention des profils transcriptomiques de chaque population cellulaire pour appliquer ICELLNET.
C.
Schéma représentant le score de communication global entre les cellules A et B, et A et C, obtenu avec ICELLNET.
D.
Diagramme représentant la contribution de chaque famille de molécules aux scores de communication obtenus avec ICELLNET. CMH : complexe majeur d’histocompatibilité.
E.
Graphique représentant les interactions ligand-récepteur qui contribuent le plus à la différence de communication observée entre les cellules A et B d’une part, et les cellules A et C d’autre part. TXLNA : α-taxiline ; STX1A : syntaxine-1A ; TNFSF9, TNFSF10 : membres de la superfamille du facteur de nécrose tumorale ; TNFRSF9, TNFRSF10D : membres de la superfamille du récepteur du facteur de nécrose tumorale ; IL-16 : interleukine 16 ; CD4 : cluster de différenciation 4 ; CXCL9, CXCL16 : chimiokines 9 et 16 (ligands des récepteurs CXCR3 et CXCR6, respectivement) ; CXCR3, CXCR6 : récepteurs des chimiokines 4 (CXCR3-B), 9, 10, 11 (CXCR3-A et -B) et 16 (CXCR6) ; HLA-DRA, HLA-DRB5 : chaîne α et chaîne β5 des antigènes d’histocompatibilité de classe II HLA-DR ; LAG3 : gène d’activation lymphocytaire 3.
|
Plusieurs cellules communiquant les unes avec les autres peuvent ainsi former des réseaux de communication intercellulaire. Un dysfonctionnement des mécanismes de communication entre cellules peut conduire à des perturbations importantes de processus biologiques et à des maladies (cancer, maladies auto-immunes, diabète, etc.). |
Comment étudier la communication intercellulaire ?
Les approches classiques d’étude de la communication intercellulaire se focalisent le plus souvent sur une ou un petit nombre d’interactions spécifiques [
1
]. Comment peut-on analyser une grande quantité d’informations sur un grand nombre de cellules ?
Les très nombreux profils cellulaires d’expression de gènes désormais disponibles grâce aux études transcriptomiques permettent d’envisager la reconstruction des réseaux de communication intercellulaire en se fondant sur la co-expression de gènes codant des paires ligand-récepteur spécifiques des cellules émettrices et réceptrices [
2
]. Plusieurs outils bioinformatiques ont été développés pour réaliser ce type d’analyse, principalement à partir de jeux de données transcriptomiques à l’échelle de la cellule unique (
single cell RNAseq
) [
2
–
6
]. Ces outils permettent d’inférer les réseaux de communication entre cellules et de proposer des hypothèses intéressantes sur le rôle de certaines populations cellulaires ou de décrypter des mécanismes moléculaires [
3
]. Néanmoins, l’intégration globale de la communication intercellulaire et l’interprétation biologique de ces voies de communication restent des enjeux importants.
|
Comment fonctionne ICELLNET ?
Nous avons développé ICELLNET, une méthode bioinformatique (package R) pour étudier la communication intercellulaire de manière globale à partir de profils transcriptomiques de populations cellulaires
(
Figure 1B
)
[
7
]. ICELLNET intègre notamment une base de données originale qui rassemble les gènes codant les différentes sous-unités des ligands et récepteurs impliqués dans de nombreuses interactions décrites dans la littérature scientifique. Cette base de données rassemble actuellement plus de 700 interactions classées par familles de molécules, et contient toutes les interactions connues pour les cytokines (247 interactions), chimiokines (112 interactions) et les molécules de contrôle immunitaire (
immune checkpoints
) (59 interactions) avec leurs récepteurs respectifs.
Pour étudier la communication entre deux types de cellules, ICELLNET calcule un score de communication à partir des profils transcriptomiques et donc de l’expression des gènes codant les ligands et récepteurs inclus dans la base de données. Chaque profil transcriptomique correspond aux gènes exprimés par un type de cellule dans un contexte biologique défini. Ainsi, le score de communication peut être calculé entre deux types différents de cellules provenant d’un même tissu, ou bien entre types cellulaires provenant de deux jeux de données différents, comme, par exemple, un même type cellulaire dans différents états d’activation. ICELLNET met également à disposition 31 profils transcriptomiques de référence, permettant de reconstruire les voies de communication d’une cellule d’intérêt avec ces cellules « types », à partir de son seul profil transcriptomique. Cela peut notamment permettre de comparer les différences de communication pour un même type cellulaire dans plusieurs états d’activation ou pour un même type de cellules provenant de tissus différents.
ICELLNET offre différentes méthodes de visualisation complémentaires et à différents niveaux de granularité et d’information pour faciliter l’analyse et l’interprétation biologique des résultats : 1) une représentation des scores de communication globaux entre les différents types de cellules sous la forme d’un réseau ; 2) l’analyse de la contribution de chaque famille de molécules (classées selon leur structure moléculaire) au score de communication ; 3) l’identification et la représentation d’interactions spécifiques ligand-récepteur ayant les scores de communication les plus élevés, ou bien les scores les plus différents entre différentes conditions biologiques
(
Figure 1C
–
E
)
.
|
Quels sont les mécanismes de communication mis en évidence par ICELLNET dans différents contextes biologiques ?
Cet outil d’analyse de la communication intercellulaire peut être employé dans différents domaines biologiques. Nous avons ainsi étudié la communication de sous-types de fibroblastes associés aux tumeurs avec les autres composants cellulaires du microenvironnement tumoral dans une forme particulière de cancer du sein [
7
], ainsi que la communication de cellules dendritiques dans différents états d’activation
in vitro
. Certaines prédictions d’ICELLNET ont ensuite été validées expérimentalement, comme, par exemple, le rôle de l’interleukine 10 dans le contrôle de la communication des cellules dendritiques humaines [
7
].
Plus récemment, l’utilisation d’ICELLNET nous a permis de mettre en évidence un dysfonctionnement de la communication entre certaines cellules présentatrices d’antigènes et les lymphocytes T chez des patients atteints d’une forme sévère de l’infection par le virus SARS-CoV-2 (
severe acute respiratory syndrome-coronavirus-2
) par rapport à des formes plus modérées de la maladie [
8
].
L’importance de la communication entre cellules dans tous les processus biologiques, en physiologie et en pathologie, ainsi que la disponibilité d’un grand nombre de profils transcriptionnels de différents types cellulaires soulignent l’importance du développement d’outils bioinformatiques dédiés à l’analyse globale de la communication intercellulaire, comme ICELLNET. Ce nouvel outil, conçu pour être utilisé par des bioinformaticiens mais aussi par des biologistes non formés à la programmation, offre de nombreuses possibilités de méta-analyses à partir de données publiques, encore largement sous-exploitées. Décrypter la communication intercellulaire permettra de mieux comprendre les mécanismes conduisant au développement et à la progression de certaines maladies, et pourrait conduire à l’identification de nouvelles cibles thérapeutiques. |
Les auteurs déclarent n’avoir aucun lien d’intérêt concernant les données publiées dans cet article.
|
Ces travaux ont bénéficié d’un financement de l’Inserm. Floriane Noël est soutenue par le laboratoire d’excellence INFLAMEX, ANR-10-LABX-17, et Lucile Massenet-Regad a bénéficié d’une allocation doctorale de la Ligue nationale contre le cancer.
|
1.
Kulbe
H
,
Chakravarty
P
,
Leinster
DA
,
et al.
A dynamic inflammatory cytokine network in the human ovarian cancer microenvironment.
.
Cancer Res.
2012;
;
72
:
:66.
–
75
.
2.
Armingol
E
,
Officer
A
,
Harismendy
O
,
et al.
Deciphering cell-cell interactions and communication from gene expression.
.
Nat Rev Genet.
2021;
;
22
:
:71.
–
88
.
3.
Vento-Tormo
R
,
Efremova
M
,
Botting
RA
,
et al.
Single-cell reconstruction of the early maternal–fetal interface in humans.
.
Nature.
2018;
;
563
:
:347.
–
353
.
4.
Efremova
M
,
Vento-Tormo
M
,
Teichmann
SA
,
et al.
Cell PhoneDB: inferring cell–cell communication from combined expression of multi-subunit ligand–receptor complexes.
.
Nat Protoc.
2020;
;
15
:
:1484.
–
506
.
5.
Jin
S
,
Guerrero-Juarez
CF
,
Zhang
L
,
et al.
Inference and analysis of cell-cell communication using Cell Chat.
.
Nat Commun.
2021;
;
12
:
:1088.
.
6.
Browaeys
R
,
Saelens
W
,
Saeys
Y
.
NicheNet: modeling intercellular communication by linking ligands to target genes.
.
Nat Methods.
2020;
;
17
:
:159.
–
62
.
7.
Noël
F
,
Massenet-Regad
L
,
Carmi-Levy
I
,
et al.
Dissection of intercellular communication using the transcriptome-based framework ICELLNET.
.
Nat Commun.
2021;
;
12
:
:1089.
.
8.
Saichi
M
,
Ladjemi
MZ
,
Korniotis
S
,
et al.
Single-cell RNA sequencing of blood antigen-presenting cells in severe COVID-19 reveals multi-process defects in antiviral immunity.
.
Nat Cell Biol.
2021;
;
23
:
:538.
–
51
.
|