| |
| Med Sci (Paris). 37(10): 895–901. doi: 10.1051/medsci/2021088.Exposome chimique et approches « non ciblées » Un changement de paradigme pour évaluer l’exposition des populations aux contaminants chimiques Arthur David,1* Jade Chaker,1* Luc Multigner,1 and Vincent Bessonneau1* 1Univ Rennes, Inserm, École des hautes études en santé publique (EHESP), Institut de recherche en santé, environnement et travail (Irset) - UMR_S 1085
,
15 avenue du Professeur Léon Bernard
,
35043Rennes
,
France |
Vignette (Photo © Inserm/Esprit sorcier).
De nombreux travaux, tels que les études d’association génétique sur « génome entier » (ou
genome-wide association studies
, GWAS), ont été menés à grande échelle depuis les années 2000, dans le cadre du programme « génome humain », afin d’étudier la contribution de déterminants génétiques dans la survenue de maladies chroniques (par exemple, les cancers ou les maladies cardio-vasculaires) ou dans les anomalies de la reproduction, qui représentent aujourd’hui des causes majeures de morbidité et, pour certaines d’entre elles, de mortalité à l’échelle mondiale [
1
,
2
]. Si ces approches de type GWAS constituent une avancée considérable pour identifier des facteurs de risque de maladies, il est maintenant reconnu que le génome et ses altérations ne peuvent à lui seul les expliquer. Il ne peut également expliquer seul les variations temporelles de l’incidence ou de la prévalence de certaines maladies chroniques que l’on observe depuis ces cinquante dernières années [
1
,
3
-
5
]. C’est dans ce contexte que le concept d’exposome a été développé en 2005 par Christopher Wild, afin de souligner l’importance d’une meilleure prise en compte des facteurs non génétiques dans la survenue des maladies [
6
], en complément des informations données par l’analyse du génome. Ce concept d’exposome renvoie à l’ensemble des facteurs environnementaux auxquels l’homme est exposé, depuis sa conception jusqu’à sa mort, ainsi qu’à l’interaction entre les caractéristiques propres de l’individu (son génotype et son phénotype) et son environnement [
6
–
8
]. Ces facteurs environnementaux sont nombreux, divers dans leur nature (physiques, chimiques, comportementaux) ; ils proviennent de multiples sources, en grande partie issues des activités humaines (alimentation, mode de vie, activités professionnelles, pollution des milieux, etc.) et varient au cours du temps
(
Figure 1
)
. En raison de la nature extrêmement variée et dynamique de ces facteurs d’exposition, le développement d’outils opérationnels fiables et robustes permettant d’évaluer l’exposome dans sa dimension holistique et ainsi d’étudier les associations possibles de cet exposome avec des évènements de santé est donc particulièrement complexe.
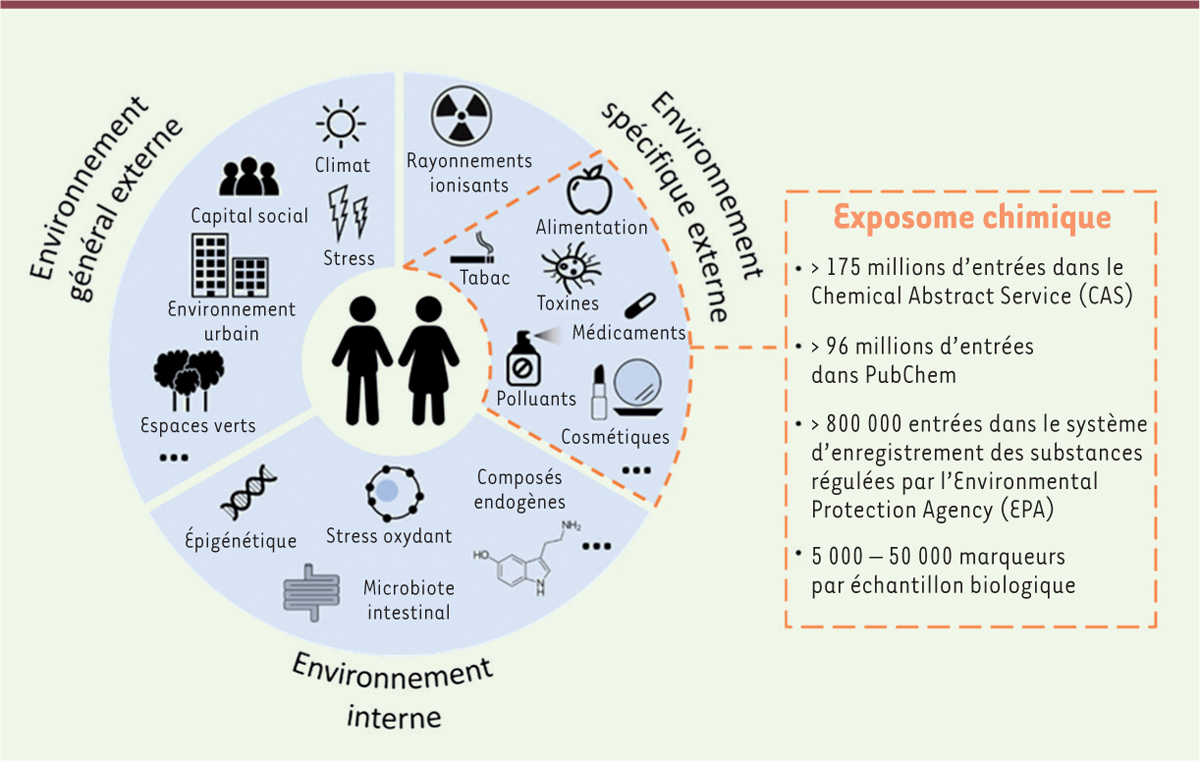
 | Figure 1.
Les différentes composantes de l’exposome, telles que décrites par Christopher Wild en 2012 [
7
], incluant l’exposome général externe, l’exposome spécifique externe et l’exposome interne.
L’exposome chimique comprend l’ensemble des composés chimiques, qu’ils soient d’origine anthropique (xénobiotiques) ou naturelle (toxines) en provenance de multiples sources. L’exposome chimique interne comprend les molécules exogènes (dont les xénobiotiques et les molécules d’origine naturelle) présentes chez l’homme, ainsi que leurs produits de transformation.
|
|
Une révolution méthodologique et technique pour décrypter l’« exposome chimique interne »
Historiquement, l’étude des effets de l’exposition aux composés chimiques, tels que les xénobiotiques comme le bisphénol A
1,
, sur la santé humaine
via
les études de cohortes en épidémiologie, ont reposé sur l’évaluation de la toxicité de molécules uniques (par opposition aux mélanges). L’un des principaux verrous qu’il est nécessaire de lever pour étudier les liens complexes entre expositions chimiques et évènements de santé, est celui de la non-exhaustivité des descripteurs actuellement disponibles (les caractéristiques intrinsèques des composés chimiques, qu’elles soient physico-chimiques ou toxicologiques) pour caractériser l’ensemble de l’ « exposome chimique interne », intégrant les molécules exogènes environnementales, dont les xénobiotiques et leurs produits de transformation, et les composés endogènes, dont ceux provenant du métabolisme, le métabolome
2
. Ce déficit de descripteurs est encore plus significatif si l’on s’intéresse à des contaminants récemment identifiés, appelés contaminants émergents, pour lesquels très peu de données d’exposition ou de toxicité sont disponibles.
Pour faire face à la complexité que représente cette myriade de composés chimiques éventuellement présents dans un échantillon, des approches innovantes reposant sur des méthodes holistiques ont émergé. La révolution technique utilisant de nouveaux équipements d’analyse par spectrométrie de masse à haute résolution (SMHR) et produisant des données en grand nombre a été concomitante du développement d’outils bio-informatiques performants permettant de traiter ces nombreuses données (algorithmes de déconvolution, extraction des signaux combinés à des statistiques multivariées, annotation des signaux obtenus par SMHR). Ces outils, qui reposent sur la définition de profils de spectrométrie de masse, permettent désormais la détection simultanée, dans les matrices biologiques examinées, de plusieurs dizaines de milliers de signaux chimiques (des molécules d’origine exogène et des composés endogènes ainsi que leurs métabolites) [
9
]. Lorsque ces nouvelles approches sont appliquées à des échantillons biologiques (sang, urine), elles apportent, en plus des données d’exposition interne (exposome chimique interne), des informations sur les réponses biologiques liées au stress induit par l’exposition externe (marqué par un changement du profil des composés endogènes à la suite d’une perturbation de l’homéostasie) [
10
]. Ces informations peuvent ainsi potentiellement renseigner sur les fonctions biologiques qui ont été affectées par l’exposition. Elles ouvrent donc de nouvelles perspectives en termes de toxicologie prédictive et d’identification de biomarqueurs d’effets précoces. La mise en place de ces nouvelles méthodes holistiques constitue une réponse opérationnelle à l’application du concept d’exposome chimique. Ces méthodes représentent ainsi une excellente opportunité de faire face à la diversité des xénobiotiques auxquels l’homme est (et sera) exposé, et pour lesquels il n’existe encore que très peu d’informations tant au niveau de leur présence que de leur toxicité [
9
]. L’implémentation de ces méthodes non ciblées, reposant sur des méthodologies de « omiques » pour le traitement des données et leur interprétation biologique, ainsi que leur mise en œuvre dans des études épidémiologiques à visée étiologique à grande échelle, ouvrent ainsi un champ majeur de recherche en santé environnementale.
|
L’exposome et les fenêtres de vulnérabilité
Un défi important pour une mesure exhaustive des expositions chimiques et l’étude de leurs implications potentielles dans la survenue de maladie chroniques, est la prise en compte de leurs variabilités dans le temps. Il n’existe, à ce jour, aucune méthode qui permette un suivi longitudinal des expositions chimiques au cours du temps, depuis la conception de l’individu jusqu’à sa mort. Certaines périodes de la vie, dites de vulnérabilité (périodes
in utero
, néonatale et de la petite enfance, adolescence, ménopause et andropause), peuvent néanmoins s’avérer critiques pour le développement de l’individu. Elles constituent donc des périodes privilégiées au cours des études. Ainsi, la période de vie prénatale est désormais considérée comme une fenêtre de sensibilité majeure aux conséquences potentiellement délétères d’expositions à des xénobiotiques, certaines pouvant se manifester à un âge ultérieur de la vie [
11
]. Ce concept est à l’origine de la notion de la DOHaD (ou origine développementale de la santé et des maladies, en anglais,
developmental origins of health and diseases
) (
→
), qui trouve ses racines dans l’origine fœtale des maladies se déclarant à l’âge adulte, un concept avancé par le médecin et épidémiologiste britannique David Barker
3
. De ce point de vue, un point de convergence évident existe entre le concept d’exposome et celui de la DOHaD ; ils pourraient donc s’enrichir mutuellement, et ainsi nous permettre de mieux comprendre l’impact de l’environnement prénatal sur notre état de santé à l’âge adulte. Toutefois, l’établissement des liens entre exposition prénatale et évènements de santé à la naissance ou à l’âge adulte reste très difficile à établir en raison des multiples facteurs qui peuvent intervenir et de leurs possibles interactions.
(→) Voir le numéro thématique Origine développementale de la santé et des maladies (DOHaD), environnement et épigénétique,
m/s
n° 1, janvier 2016
Le choix des matrices biologiques sur lesquelles les nouvelles méthodes d’analyse reposant sur la SMHR pourraient être appliquées afin de caractériser les expositions chimiques durant les premiers âges de la vie reste encore à être précisé. L’analyse chimique de prélèvements de fluides et de tissus biologiques maternels (urines, sang veineux périphérique, cheveux, etc.) ou fœtaux (sang de cordon, etc.) est habituellement réalisée pour estimer l’exposition fœtale aux polluants [
12
]. Des prélèvements complémentaires d’organes, de tissus ou de fluides apparaissent également importants à examiner afin que l’analyse reflète au mieux l’exposition réelle du fœtus [
13
]. En cela, le recours au placenta, qui reste peu exploré, apparaît potentiellement pertinent lorsqu’il s’agit d’étudier et de caractériser l’exposome chimique de l’embryon et du fœtus au cours de la grossesse.
|
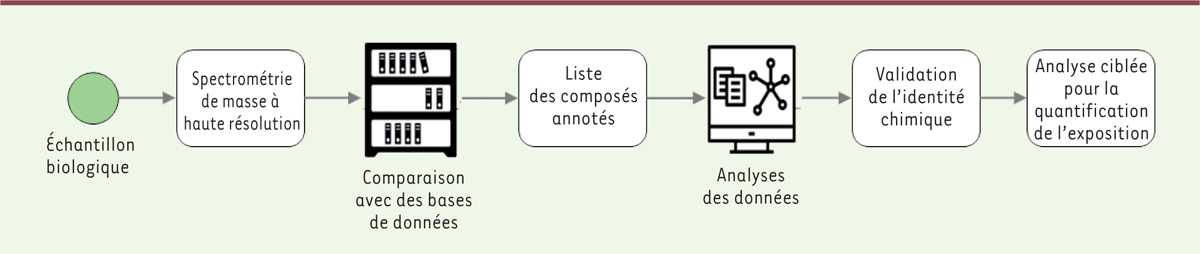
Les différentes étapes associées à une approche analytique non ciblée reposant sur la SMHR
L’utilisation des approches reposant sur la SMHR pour caractériser l’exposome chimique se traduit de manière opérationnelle par l’application d’une suite d’étapes
(
Figure 2
)
comprenant : 1) la préparation et l’analyse des échantillons ; 2) l’extraction bio-informatique des marqueurs obtenus ; et 3) leur annotation.
 | Figure 2.
Les différentes étapes impliquées dans l’application d’approches analytiques non ciblées reposant sur la spectrométrie de masse à haute résolution (SMHR).
|
La prise en compte de la toxicocinétique
4,
des xénobiotiques qui seront analysés est un élément important pour le choix de la matrice biologique sur laquelle ces approches seront déployées. Ce choix est en partie dicté par la recherche de certains xénobiotiques, qui peuvent être métabolisés et présents dans les fluides biologiques ou les tissus (comme l’urine pour les composés chimiques non persistants ayant une courte demi-vie), sous la forme de produits de biotransformation (métabolites de phase I et/ou II)
5
[
14
]. La disponibilité des échantillons, mais aussi l’expertise du laboratoire effectuant l’analyse, peuvent aussi avoir une incidence sur le choix de la matrice qui sera utilisée (l’échantillon pouvant ne pas être disponible ou le laboratoire ne pas avoir la compétence pour le traiter). Les échantillons biologiques qui seront sélectionnés seront préparés selon des méthodes pour lesquelles un compromis devra être trouvé entre non-sélectivité de la technique (afin de conserver le maximum d’exhaustivité) et traitement de l’échantillon (souvent nécessaire pour éliminer certains composés endogènes qui sont présents à de fortes concentrations et qui peuvent induire des interférences).
L’analyse des échantillons par SMHR, qui est habituellement couplée à une première étape permettant de séparer les constituants de l’extrait selon des critères biophysiques (méthode séparative, telle que la chromatographie), permet de détecter un ensemble de composés qui, à ce stade, ne seront pas identifiés. Il s’agira en effet de profils ou d’empreintes chimiques brutes indiquant les différentes masses des molécules sous forme native (
i.e.
l’ion moléculaire), de leurs isotopes, leurs potentiels adduits formés par la présence d’autres ions
6,
, ou encore de leurs fragments présents dans l’échantillon. À ces différentes masses sont associés des temps de rétention chromatographique et des abondances ou des intensités bien définis. La SMHR permet d’obtenir, avec une grande précision, la masse des composés détectés, avec une incertitude de l’ordre de 5 à 10 parties par million (ppm ; soit de 5x10
-4
à 1x10
-3
d’erreur à une masse de 100 unités de masse
7
) selon le type d’appareil utilisé. Les données brutes recueillies seront ensuite traitées par des logiciels de bio-informatique afin de transformer l’ensemble des spectres de masse obtenus en une matrice correspondant à l’ensemble des marqueurs détectés. Chacun de ces marqueurs se définit par une masse exacte (obtenue par spectrométrie) et un temps de rétention (mesuré à l’étape de la chromatographie), qui sont associés à une abondance dans l’échantillon analysé.
Les valeurs de masses exactes, interprétées par les logiciels de bio-informatique, peuvent alors être associées à des formules chimiques brutes connues, et comparées à des valeurs de référence intégrées dans des bases de données. Ces dernières peuvent être généralistes, telles que
Exposome Explorer
[
15
] ou
Human Metabolome Databas
e [
16
], ou spécifiques de la matrice utilisée, telles que
Serum Metabolome
[
17
]. Des outils permettent d’automatiser cette étape d’identification par comparaison, comme les logiciels
xMSannotator
[
18
] ou
MS-DIAL
[
19
]. Certains permettent également d’effectuer des liens entre différentes masses pour reconstituer le profil isotopique des composés détectés (notamment un profil distinctif, dans le cas de composés bromés ou chlorés) [
20
], ou pour créer des réseaux de composés
a priori
liés biologiquement entre eux (composés impliqués dans les mêmes processus biologiques, ou issus d’un même composé parental, etc.). Il est ainsi possible d’obtenir une liste de composés qui seront annotés automatiquement. Une étape de vérification (ou « curation ») manuelle des données sera toutefois nécessaire, afin de confirmer les annotations fournies automatiquement, et ainsi obtenir une identification formelle. Cette dernière étape consiste à resserrer le faisceau d’indices autour des candidats qui ont été annotés afin d’aboutir à une identification précise de la molécule. Ces indices reposent notamment sur le profil de fragmentation de la molécule, lorsque celle-ci a été analysée par spectrométrie de masse en tandem (MS/MS), profil qui peut être comparé à un profil de fragmentation de molécules connues, obtenu expérimentalement, ou à un profil qui a été prédit
in silico
.
Le temps de rétention (spécifique du système de chromatographie utilisé), comparé à une valeur obtenue en utilisant le même système avec un composé standard, permettra de confirmer l’identité chimique du composé d’intérêt.
Une grille d’évaluation indiquant la confiance accordée à une annotation, proposée par Emma Schymanski
et al.
en 2014 [
21
], permet d’attribuer une note comprise entre 5 (confiance faible, masse exacte uniquement) et 1 (confiance maximale, identification formelle) à chaque annotation, afin d’harmoniser la communication entre laboratoires.
Une étude non ciblée peut donc permettre d’identifier des composés d’intérêt (distinguant deux groupes, par exemple). Cependant, ces approches ne permettent pas d’obtenir de quantification absolue des composés détectés. Des méthodes ciblées seront développées à cette fin, sur la base des identifications d’intérêt obtenues avec les approches non ciblées. Ces approches non ciblées peuvent ainsi être déployées à des fins exploratoires afin de fournir une évaluation non biaisée de l’exposition aux contaminants chimiques organiques. |
Les défis analytiques et méthodologiques à surmonter
Les approches non ciblées constituent une opportunité permettant de mieux caractériser les expositions aux xénobiotiques et de mieux comprendre les effets qui leur sont associés. Toutefois, il est essentiel de considérer et de dépasser certaines limites analytiques et méthodologiques afin de pouvoir pleinement bénéficier des avantages de ces approches. Avant de pouvoir caractériser l’exposome chimique à large spectre à l’aide de ces approches, il convient en effet de considérer : 1) la nécessité de les associer à une large gamme de méthodes complémentaires (chromatographie liquide, gazeuse ou ionique), afin de couvrir la diversité des contaminants chimiques susceptibles d’être présents dans l’environnement et qui possèdent des propriétés physico-chimiques extrêmement diverses (il existe plus de 96 millions de molécules référencées dans la base PubChem
8
) ; 2) les problèmes de sensibilité analytique liés au fait que de nombreux contaminants et métabolites d’intérêt ne sont présents qu’à l’état de traces (i.e. de l’ordre du ng/mL ou inférieur) en comparaison à certains métabolites endogènes présents à des concentration de l’ordre du mg/mL ; 3) le manque d’outils permettant une annotation (attribution d’une identité moléculaire à un signal chimique) automatisée des ensembles de données obtenus (actuellement, seuls 10 % de ces ensembles de données peuvent être annotés), et le manque de bases de données spécifiques à l’exposome chimique ; 4) le manque d’harmonisation et de standardisation des traitements des données obtenues par ces méthodes non ciblées, ce qui permettrait une comparaison des données entre laboratoires ; et 5) le besoin de changer d’échelle pour étudier l’exposome chimique à l’échelle populationnelle : la plupart des études restent actuellement au stade de « preuve de concept ».
La mise en place de plateformes analytiques de SMHR à grande échelle, comme le
Hercules Exposome Research Center
(Université Emory, aux États-Unis), ou le
National Phenome Centre
, soutenu par le MRC (
Medical Research Council
) et le NIHR (
National Institute for Health Research)
, au Royaume-Uni, possédant une taille suffisante pour couvrir la profondeur et l’étendue de l’exposome chimique et une puissance analytique adaptée pour les analyses de cohortes, constitue assurément une avancée pour surmonter les défis analytiques et méthodologiques qui se posent pour l’analyse de l’exposome. Certaines initiatives européennes sont actuellement en cours de construction pour mettre en place des réseaux de laboratoires qui pourraient constituer, à terme, une infrastructure de recherche sur l’exposome chimique capable de répondre aux besoins des études de cohortes.
|
Les premières applications internationales à grande échelle
Il n’existe, à ce jour, aucune application concrète et opérationnelle à grande échelle des approches d’analyses non ciblées. Toutefois, quelques premières initiatives de recherche sont apparues en Europe au début des années 2010
(
Tableau I
)
. Le programme européen FP7 (2012-2017) a ainsi soutenu trois projets reposant sur des mesures ciblées de l’exposome chimique, visant à améliorer les connaissances de l’impact des expositions environnementales sur la santé des populations.
Tableau I.
| Projets européens (FP7) (2012-2017) |
Nom des projets
But
|
Fonds (millions d’euros) |
| HELIX |
The Human Early-Life Exposome
Nouveaux outils pour intégrer expositions environnementales au début de la vie et santé des enfants à travers l’Europe
|
11,2 |
| EXPOsOMICs |
Enhanced exposure assessment and omic profiling
Pour les expositions environnementales hautement prioritaires en Europe
|
11,6 |
| HEALS |
Health and Environment-wide Associations based on Large population Surveys |
15 |
|
|
Projets NIEHS (États-Unis)
|
Nom des projets But
|
Fonds (millions de dollars)
|
| CHEAR (2015-2019) |
The Children’s Health Exposure Analysis Resource
Recherche sur les influences de l’environnement sur la santé des enfants
|
34 |
| HHEAR (2019-2024) |
Human Health Exposure Analysis Resource
Cartographie de l’exposome humain
|
35 |
|
|
Projets européens (H2020) (2017-2022)
|
Nom des projets But
|
Fonds (millions d’euros)
|
| HBM4EU |
The European Human Biomonitoring Initiative
Générer des connaissances pour une information claire sur les produits chimiques et pour protéger la santé humaine
|
74 |
|
|
The European Human Exposome Network
(9 projets, 24 pays) (2020-2025)
|
|
106 |
| EXPANSE |
Exposome powered tools for healthy living in urban settings
|
|
| EQUAL-LIFE |
Early environmental quality and life-course mental health effects
|
|
| LONGITOOLS |
Dynamic longitudinal exposome trajectories in cardiovascular and metabolic non-communicable diseases
|
|
| ATHLETE |
Advancing tools for human early life-course exposome research and translation
|
|
| EXIMIOUS |
Mapping exposure-induced immune effects: connecting the exposome and the immunome
|
|
| HEDIMED |
Human exposomic determinants of immune mediated diseases
|
|
| HEAP |
Human exposome assessment platform
|
|
| REMEDIA |
Impact of exposome on the course of lung diseases
|
|
| EPHOR |
Exposome project for health and occupational research
|
|
Initiatives de recherche internationales pour l’étude des liens entre l’exposome chimique et la santé
. NIEHS :
National Institute of Environmental Health Sciences.
|
Le projet HELIX [
22
] s’est intéressé à l’étude, au sein de six cohortes prospectives de naissance, déjà établies (représentant 32 000 couples mère-enfant et 1 200 mesures de marqueurs d’exposition), des liens possibles entre santé, croissance et développement de l’enfant et imprégnation au cours des premières années de vie par des polluants persistants (polychlorobiphényles [PCB], dichlorodiphényldichloroéthylène [DDE], hexachlorobenzène [HCB], polybromodiphényléthers [PBDE] et substances perfluorées [PFAS]), à des polluants non-persistants (phtalates, phénols et pesticides organochlorés), et à des métaux.
Les projets EXPOsOMICs [
23
] et HEALS ont mesuré une centaine de biomarqueurs d’exposition chez plus de 3 000 participants provenant de 14 régions européennes, dont des nouveau-nés, des enfants et des adultes, afin d’évaluer les liens entre les expositions environnementales au cours de différentes périodes de vulnérabilité et la survenue d’évènements de santé (maladies cardiovasculaires, maladies respiratoires, développement neurologique anormal, obésité, diabète de type 2, etc.).
Bien que ces études s’intéressent à des mélanges complexes, le nombre de substances identifiées est encore très loin de celui qui permettrait une description exhaustive de l’exposome chimique, voire de celui que les plateformes analytiques utilisant la SMHR à large échelle pourraient mesurer. Ces études s’intéressent également aux relations entre les mesures d’expositions externes (mesures environnementales) et l’exposome chimique interne (mesures d’imprégnation) afin de fournir de nouveaux outils pour l’évaluation de ces expositions.
À partir de 2017, le soutien européen de la recherche sur l’exposome chimique s’est poursuivi avec le projet HBM4EU (
European Human Biomonitoring Initiative
). Celui-ci visait à harmoniser les activités de biosurveillance (mesure d’imprégnation dans les matrices biologiques) menées dans 28 pays partenaires, afin d’améliorer l’évaluation de l’exposition aux substances chimiques en Europe et, notamment, permettre une meilleure compréhension des effets sur la santé des mélanges complexes de substances chimiques. Ce projet est l’un des premiers à avoir inclus l’analyse non ciblée de matrices biologiques par SMHR pour une caractérisation exhaustive, sans
a priori
, des expositions environnementales.
En 2020, L’Europe a annoncé la création d’un réseau de recherche,
the European Human Exposome Network
, qui financera 9 projets impliquant 126 groupes de recherche issus de 24 pays. Ce réseau vise à poursuivre le développement de nouveaux outils pour une meilleure caractérisation de l’exposome, en particulier l’exposome chimique, ainsi qu’à leurs utilisations à large échelle pour améliorer les connaissances de l’impact des expositions environnementales sur la santé des populations.
Aux États-Unis, le
National Institute of Environmental Health Sciences
(NIEHS) a lancé en 2015 une première initiative de grande ampleur pour la mise en place d’une infrastructure de recherche (CHEAR pour
Children’s Health Exposure Analysis Resource
) permettant l’étude des liens entre l’exposome chimique et la santé des enfants [
24
]. Cette infrastructure regroupait des laboratoires spécialisés dans l’analyse chimique (ciblées et non ciblées) et l’analyse des données massives pour la caractérisation de l’exposome chimique. L’objectif de ce programme était de permettre à la communauté scientifique dont les recherches sont centrées sur la santé des enfants, d’avoir accès, sans frais, à une infrastructure de pointe pour la caractérisation à large échelle de l’exposome chimique. Il a permis l’évaluation de l’exposome chimique chez plus de 50 000 enfants, inclus dans plus de 30 études s’intéressant aux liens entre les expositions environnementales au cours des premières années de vie et la survenue d’évènements de santé chez les enfants (tels que l’asthme, le diabète, l’autisme, l’obésité, etc.). Une nouvelle infrastructure (HHEAR pour
Human Health Exposure Analysis Resource
), similaire à CHEAR, a été établie en 2019 pour étudier les effets de l’exposome chimique au cours de différentes fenêtres de vulnérabilité sur la santé de la population adulte.
Ces premières applications à grande échelle ont montré que l’analyse non ciblée par spectrométrie de masse à haute résolution (SMHR) pourrait constituer, dans un avenir proche, une approche robuste pour la détection de plusieurs milliers de substances chimiques à partir d’un échantillon biologique unique, et ainsi permettre de révéler de nouvelles expositions à des xénobiotiques. Cette nouvelle approche offre une opportunité unique d’identifier de nouveaux facteurs de risques environnementaux pour les maladies chroniques. Cependant, des efforts de recherche supplémentaires sont nécessaires, tant en ce qui concerne l’aspect analytique que l’aspect bio-informatique, pour couvrir la profondeur et l’étendue de l’exposome chimique et faciliter l’annotation et l’interprétation des données massives ainsi produites. |
Les auteurs déclarent n’avoir aucun lien d’intérêt concernant les données publiées dans cet article.
|
Footnotes |
1.
Cui
Y
,
Balshaw
DM
,
Kwok
RK
,
et al.
The exposome: embracing the complexity for discovery in environmental health.
.
Environ Health Perspect.
2016;
;
124
:
:A137.
–
A140
.
2.
Bodmer
W
,
Bonilla
C
.
Common and rare variants in multifactorial susceptibility to common diseases.
.
Nat Genet.
2008;
;
40
:
:695.
–
701
.
3.
Manolio
TA
,
Collins
FS
,
Cox
NJ
et al.
Finding the missing heritability of complex diseases.
.
Nature.
2009;
;
461
:
:747.
–
753
.
4.
Rappaport
SM
Genetic factors are not the major causes of chronic diseases.
.
PLoS One.
2016;
;
11
:
:e0154387.
.
5.
Vermeulen
R
,
Schymanski
EL
,
Barabasi
AL
,
Miller
GW
.
The exposome and health: where chemistry meets biology.
.
Science.
2020;
;
367
:
:392.
–
6
.
6.
Wild
CP
.
Complementing the genome with an exposome: the outstanding challenge of environmental exposure measurement in molecular epidemiology.
.
Cancer Epidemiol Biomarkers Prev.
2005;
;
14
:
:1847.
–
1850
.
7.
Wild
CP
The exposome: from concept to utility.
.
Int J Epidemiol.
2012;
;
41
:
:24.
–
32
.
8.
Rappaport
SM
,
Barupal
DK
,
Wishart
D
et al.
The blood exposome and its role in discovering causes of disease.
.
Environ Health Perspect.
2014;
;
122
:
:769.
–
774
.
9.
Dennis
KK
,
Marder
E
,
Balshaw
DM
et al.
Biomonitoring in the era of the exposome.
.
Environ Health Perspect.
2017;
;
125
:
:502.
–
510
.
10.
Bessonneau
V
,
Gerona
RR
,
Trowbridge
J
, et al.
Gaussian graphical modeling of the serum exposome and metabolome reveals interactions between environmental chemicals and endogenous metabolites.
.
Sci Rep.
2021;
;
11
:
:7607.
–
22
.
11.
Barouki
R
,
Gluckman
PD
,
Grandjean
P
et al.
Developmental origins of non-communicable disease: implications for research and public health.
.
Environ Health.
2012;
;
11
:
:42.
.
12.
Chevrier
C
,
Limon
G
,
Monfort
C
et al.
Urinary biomarkers of prenatal atrazine exposure and adverse birth outcomes in the PELAGIE birth cohort.
.
Environ Health Perspect.
2011;
;
119
:
:1034.
–
1041
.
13.
Barr
DB
,
Bishop
A
,
Needham
LL
Concentrations of xenobiotic chemicals in the maternal-fetal unit.
.
Reprod Toxicol.
2007;
;
23
:
:260.
–
266
.
14.
David
A
,
Chaker
J
,
Léger
T
, et al.
Acetaminophen metabolism revisited using non-targeted analyses: Implications for human biomonitoring.
.
Environ Int.
2021;
;
149
:
:106388.
.
15.
Neveu
V
,
Moussy
A
,
Rouaix
H
et al.
Exposome-explorer: a manually-curated database on biomarkers of exposure to dietary and environmental factors.
.
Nucleic Acids Res.
2017;
;
45
:
:D979.
–
D984
.
16.
Wishart
DS
,
Tzur
D
,
Knox
C
et al.
hmdb: the human metabolome database.
.
Nucleic Acids Res.
2007;
;
35
:
:D521.
–
D526
.
17.
Psychogios
N
,
Hau
DD
,
Peng
J
et al.
The human serum metabolome.
.
PLoS One.
2011;
;
6
:
:e16957.
.
18.
Uppal
K
,
Walker
DI
,
Jones
DP
xMSannotator: an R package for network-based annotation of high-resolution metabolomics data.
.
Anal Chem.
2017;
;
89
:
:1063.
–
1067
.
19.
Tsugawa
H
,
Cajka
T
,
Kind
T
et al.
MS-DIAL: data-independent MS/MS deconvolution for comprehensive metabolome analysis.
.
Nat Methods.
2015;
;
12
:
:523.
–
526
.
20.
Chaker
J
,
Gilles
E
,
Léger
T
, et al.
From metabolomics to HRMS-based exposomics: adapting peak picking and developing scoring for MS1 suspect screening.
.
Anal Chem.
2021;
;
93
:
:1792.
–
800
.
21.
Schymanski
EL
,
Jeon
J
,
Gulde
R
et al.
Identifying small molecules via high resolution mass spectrometry: communicating confidence.
.
Environ Sci Technol.
2014;
;
48
:
:2097.
–
2098
.
22.
Vrijheid
M
,
Slama
R
,
Robinson
O
et al.
The human early-life exposome (helix): project rationale and design.
.
Environ Health Perspect.
2014;
;
12
:
:535.
–
544
.
23.
Vineis
P
,
Chadeau-Hyam
M
,
Gmuender
H
et al.
The exposome in practice: design of the EXPOsOMICS project.
.
Int J Hyg Environ Health.
2017;
;
220
:
:142.
–
151
.
24.
Balshaw
DM
,
Collman
GW
,
Gray
KA
,
Thompson
CL
The children’s health exposure analysis resource (chear): enabling research into the environmental influences on children’s health outcomes.
.
Curr Opin Pediatr.
2017;
;
29
:
:385.
–
389
.
|