| |
| Med Sci (Paris). 37(5): 461–467. doi: 10.1051/medsci/2021062.Les principaux outils biologiques appliqués au dépistage néonatal État des lieux et perspectives d’avenir David Cheillan1,2* 1Service de biochimie et biologie moléculaire - Centre de biologie Est, Hospices Civils de Lyon
,
69500Bron
,
France 2Commission de biologie - Centre national de coordination du dépistage néonatal
,
69500Bron
,
France |
Vignette (© CNCDN).
Le dépistage néonatal (DNN) est un programme de santé publique visant à rechercher de nombreuses maladies chez tous les nouveau-nés (NN) avant l’apparition des signes cliniques, afin de leur proposer un traitement et une prise en charge adaptée. Depuis les années 1960 et le succès de la mise en place du dépistage de la phénylcétonurie chez les nouveau-nés [
1
], l’essor du DNN n’a cessé de croître. On dénote ainsi aujourd’hui près d’une centaine de maladies dépistables dont la plupart répondent aux critères de James Maxwell Glover Wilson et Gunnar Jungner [
2
,
41
] (
→
).
(→) Voir la Synthèse de J.P. Farriaux, page 433 de ce numéro
Depuis la découverte par Guthrie d’un test simple ayant permis le dosage de la phénylalanine à partir de taches de sang prélevé au talon du nouveau-né, déposées sur un buvard, le DNN repose sur ce mode de prélèvement et sur l’utilisation de techniques biologiques permettant de déterminer différents paramètres spécifiques des maladies recherchées.
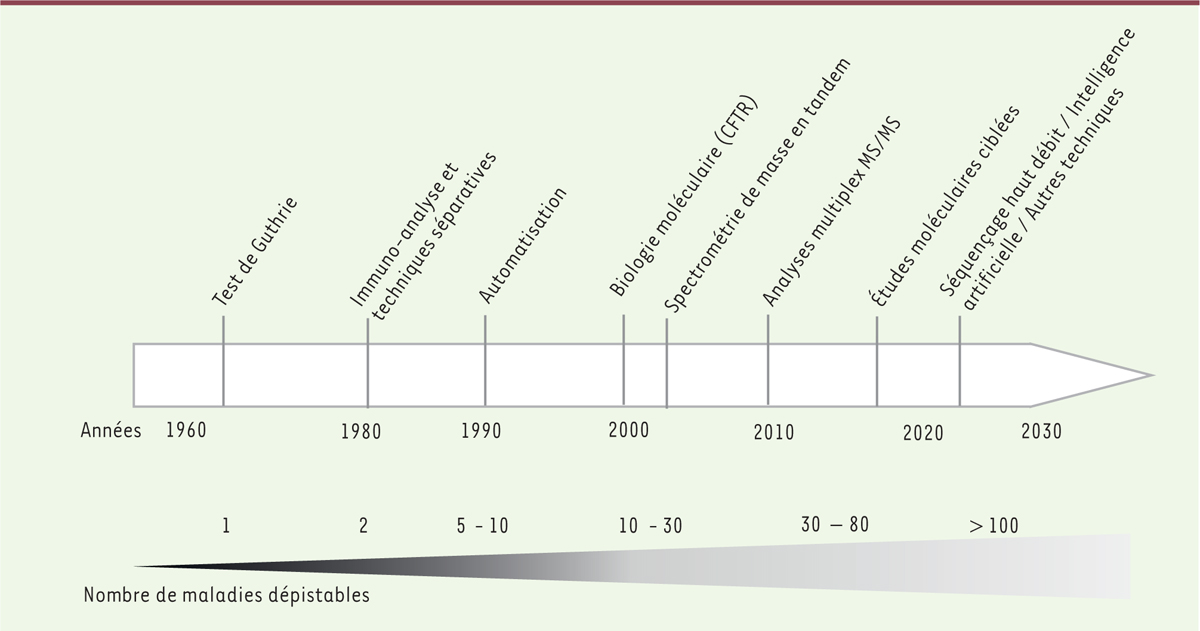
Les outils mis en œuvre dans le DNN ont suivi les évolutions techniques de la biologie médicale : développement de l’immunoanalyse et de l’automatisation dans les années 1970-1990, puis essor de la spectrométrie de masse dans les années 2000 et, enfin, révolution actuelle par le séquençage à haut débit du génome
(
Figure 1
)
. Seules quelques maladies, comme la surdité ou les cardiopathies congénitales, mettent en œuvre des dépistages non biologiques que nous n’aborderons pas dans cette Synthèse. L’objectif sera ici de présenter un panorama le plus exhaustif possible des outils biologiques appliqués au DNN, avec leurs intérêts mais aussi leurs limites, et d’envisager les perspectives d’avenir de cette thématique en perpétuelle évolution.
 | Figure 1.
Apport des évolutions techniques en fonction du temps dans le développement des programmes de dépistage néonatal.
CFTR :
cystic fibrosis transmembrane conductance regulator
.
|
|
Le prélèvement de Guthrie
Au-delà du test d’inhibition de croissance bactérienne développé en 1963 par Robert Guthrie qui permettait le dosage de la phénylalanine à partir du sang déposé sur un buvard, ce mode de prélèvement est resté le point central du tout programme de DNN et tend à se développer en biologie clinique et dans le suivi des patients [
3
,
4
].
Les principales caractéristiques de ce mode de prélèvement sont :
-
un microprélèvement de sang capillaire : une goutte de sang représentant 50 à 75 µl de sang total,
-
le support du prélèvement, de type buvard, qui bénéficie d’un marquage «
in vitro diagnostic
» validé et a donc des qualités rhéologiques spécifiques et contrôlées,
-
après séchage, à température ambiante, le prélèvement peut être conservé plusieurs semaines à température ambiante et peut être transporté par simple courrier postal (exception à la réglementation du transport des substances dangereuses de l’Organisation mondiale de la santé [OMS]),
-
la prise d’essai se fait grâce à des systèmes de poinçonnage du buvard qui permettent d’obtenir, en général, une pièce de 3 mm de diamètre contenant environ 3 µl de sang total.
À partir de ce prélèvement, il est ensuite possible de quantifier de nombreux paramètres biologiques, petites molécules, protéines, activités enzymatiques, etc., mais aussi de déterminer la présence d’agents infectieux et, enfin, de réaliser des études de génétique moléculaire. Il est néanmoins important d’en connaître les limites, comme l’incertitude du volume de sang contenu dans l’emporte-pièce utilisé pour prélever l’échantillon à partir de la tache, les effets chromatographiques possibles, l’impact de l’hématocrite, ou la nécessité de l’utilisation d’automates dédiés à ce type de test [
5
].
De nouveaux systèmes d’autoprélèvement, sous la forme de stylo ou au format type carte de crédit, sont apparus sur le marché. Ils sont constitués de microcapillaires de diamètre interne fixe (3 µl ou 10 µl) qui permettent de standardiser le volume de sang prélevé. À l’avenir, ces systèmes devraient probablement permettre de réduire les inconvénients de la prise de l’échantillon tout en conservant les qualités intrinsèques de ce prélèvement si particulier [
6
,
7
].
|
Seuils et performances d’un test de dépistage
Le DNN a pour objectif d’identifier, dans l’ensemble de la population des nouveau-nés, les individus atteints d’une maladie en minimisant les risques de faux-négatifs (ce qui repose sur la sensibilité du test) et de faux-positifs (ce qui repose sur sa spécificité). Il est donc important de déterminer des seuils d’action offrant les meilleures performances. Ces seuils sont définis selon la distribution au sein de la population des paramètres qui sont mesurés (en général entre le 99
e
percentile de la population normale et le premier percentile de la population des malades), mais aussi en fonction des méthodes de dosage utilisées. Selon les paramètres évalués, il peut parfois être important de proposer des seuils différents chez les enfants prématurés afin de prendre en considération la spécificité de cette catégorie de nouveau-nés. Les performances des seuils ainsi choisis doivent être évaluées régulièrement, en suivant différentes variables, dont la valeur prédictive positive et la valeur prédictive négative
(
Tableau I
)
.
Tableau I.
|
Nouveau-nés malades |
Nouveau-nés non malades |
|
|
|
|
|
Valeur prédictive positive
VPP = VP / (VP + FP)
|
|
Test positif
|
Vrais positifs (VP) |
Faux positifs (FP) |
Probabilité d’être malade quand le test est positif
|
|
|
Test de dépistage
|
|
|
|
|
|
Test négatif
|
Faux négatifs (FN) |
Vrais négatifs (VN) |
Valeur prédictive négative
VPN = VN / (VN + FN)
|
|
|
|
|
Probabilité d’être non malade quand le test est négatif
|
|
|
|
Sensibilité
VP / (VP + FN)
|
Spécificité
VN / (VN + FP)
|
|
|
|
Pourcentage de malades identifiés
|
Pourcentage de non malades identifiés
|
|
Performances d’un test de dépistage : déterminations de la sensibilité, de la spécificité, et des valeurs prédictives positive et négative du test.
|
|
Les techniques biochimiques classiques Les programmes de DNN ont suivi les évolutions techniques de la biologie médicale et, naturellement, des méthodes de quantification de paramètres biologiques à partir de sang déposé sur le buvard se sont développées en utilisant la colorimétrie, la fluorimétrie, l’immunoanalyse ou les méthodes séparatives. La colorimétrie et la fluorimétrie
Pendant de très nombreuses années, la fluorimétrie a été la méthode de choix pour évaluer les taux de phénylalanine dans le sang. Ce dosage reposait sur la méthode de Richard Mc Caman et Eli Robins [
8
], fondée sur la formation, en présence de ninhydrine et d’un dipeptide (la leucyl-alanine), d’un composé fluorescent. Cette méthode n’est pratiquement plus utilisée. Elle a été remplacée par la spectrométrie de masse en tandem (MS/MS) sur laquelle nous reviendrons.
Dans les pays qui réalisent un DNN de la galactosémie congénitale ou du déficit en glucose-6-phosphate deshydrogénase (G6PD), la fluorimétrie reste néanmoins utilisée pour la mesure du galactose total ou de l’activité de la G6PD en utilisant le principe de la fluorescence émise par le NADP (nicotinamide adénine dinucléotide phosphate) réduit, qui dépend de son état d’oxydation. La méthode de dosage colorimétrique n’est pratiquement plus utilisée en raison de sa faible sensibilité analytique, mais elle peut être utile pour la mesure de l’activité de la biotinidase
1
.
L’immunoanalyse
Les méthodes utilisant l’immunoanalyse ont été développées lorsque des maladies endocriniennes ont été introduites dans les programmes de DNN. Il s’agissait de l’hypothyroïdie congénitale (avec la mesure de la thyréostimuline, TSH), de l’hyperplasie congénitale des surrénales (avec évaluation de la 17-hydroxyprogestérone) et de la mucoviscidose (par mesure de la trypsine immunoréactive). Ces techniques, très utilisées en biologie clinique, ont été adaptées à l’utilisation du sang déposé sur buvard avec des automates dédiés. En raison de la faible prise d’essai (quelques µl de sang total), les techniques déployées nécessitent une grande sensibilité analytique. La méthode de révélation est ainsi très souvent fondée sur de la fluorimétrie en temps résolu, avec l’utilisation de terres rares (ou lanthanides) comme l’europium. Cette technique est systématiquement utilisée à travers le monde dans le cadre du DNN, pour les paramètres hormonologiques [
9
].
Isoélectrofocalisation, chromatographie liquide haute performance et électrophorèse capillaire
Les méthodes séparatives classiquement utilisées en biologie clinique sont assez peu appliquées au DNN. La principale application de ces méthodes est le dépistage de la drépanocytose, afin de rechercher l’hémoglobine anormale HbS, résultant d’une mutation du gène codant la b-globine, l’une des chaînes de l’hémoglobine (a2b2). Historiquement, l’isoélectrofocalisation (IEF) de l’hémoglobine, qui permet de mettre en évidence une différence de point isoélectrique entre la protéine mutée et la protéine normale, a été largement employée, mais actuellement l’électrophorèse capillaire et la chromatographie en phase liquide de haute performance (CLHP ou en anglais HPLC pour
high performance liquid chromatography
) sont les méthodes les plus courantes, plusieurs fabricants ayant développé des solutions techniques adaptées à l’utilisation du sang déposé sur buvard. Ces techniques ont toutes des performances analytiques comparables et peuvent être utilisées indifféremment [
10
].
La spectrométrie de masse
La spectrométrie de masse (MS) est une technique permettant de séparer les molécules contenues dans une matrice complexe selon leur masse et leur charge, après des étapes d’ionisation et de fragmentation. En biologie clinique, cet outil, couplé très souvent à la chromatographie en phase liquide, a permis de considérablement améliorer les techniques séparatives. Dans le cadre du DNN, c’est surtout la spectrométrie de masse en tandem (MS/MS) qui s’est développée dans les années 1990, avec la mise au point et l’identification de profils spécifiques des acylcarnitines
2
pour la recherche des anomalies de la b-oxydation mitochondriale des acides gras et, plus largement, pour le dépistage de nombreuses maladies héréditaires du métabolisme (MHM) [
11
,
12
]. Les appareils utilisés pour cette application sont, assez classiquement, de type « triple quadripôles avec une source
electrospray
». Le principal intérêt de cette méthode est de pouvoir réaliser des analyses en multiplex fondées sur la réalisation de profils ou la quantification simultanée de nombreuses molécules (mode MRM –
multi-reaction monitoring
) à partir d’un seul échantillon prélevé par un emporte-pièce de 3 mm à partir de la tache de sang, sans nécessiter une séparation préalable, et donc en quelques minutes. Grâce à ces avancées, la MS/MS est devenu un outil central pour le DNN.
L’application de la spectrométrie de masse pour le dépistage des maladies héréditaires du métabolisme Les premières avancées de cette méthode dans le DNN des MHM ont été rendues possibles grâce à la réalisation de profils d’acylcarnitines. Ces molécules sont en effet idéales pour une analyse en MS/MS. Toutes les acylcarnitines produisent, après fragmentation, un ion de 85 Daltons (Da) qui permet très facilement d’identifier et de quantifier toutes les espèces moléculaires de cette famille. Lors d’un déficit de la b-oxydation mitochondriale des acides gras, les espèces d’acylcarnitines, en amont du blocage enzymatique, s’accumulent. Un déficit énergétique apparaît alors, entraînant les symptômes de la maladie. Le plus fréquent de ces déficits est la carence en acyl-CoA deshydrogénase des acides gras à chaîne moyenne (MCAD) qui est à l’origine d’une accumulation d’octanoylcarnitine (ou C8) caractéristique de cette maladie.
Rapidement, les possibilités d’analyses multiplex offertes par la MS/MS ont été exploitées en ajoutant la quantification de plusieurs acides aminés, par la perte neutre d’un ion de masse de 102 Da après fragmentation. Les méthodes développées permettaient ainsi, en plus des anomalies de la b-oxydation mitochondriale, le dépistage de nombreuses maladies du métabolisme intermédiaire, amino-acidopathies, et aciduries organiques. Les kits disponibles permettent désormais de dépister plus d’une trentaine de MHM [
13
]. Initialement, les techniques nécessitaient une étape de dérivation (avec l’introduction d’un groupe butyl, ou butylation) mais les évolutions techniques des appareils ont rendu possible le passage des échantillons directement après extraction, sans manipulation préalable de ces derniers, avec l’ajout de standards internes deutérés
,
réduisant ainsi de façon importante les temps de préparation des séries d’échantillons. Enfin, les industriels ont amélioré leurs kits en offrant la possibilité de quantifier des molécules additionnelles pour accroître les performances du DNN de certaines maladies, comme, par exemple, l’ajout de succinyl acétone pour le DNN de la tyrosinémie de type I. Actuellement, bien que la liste des MHM dépistées varie d’un pays à l’autre, il est indéniable que le dépistage de ces maladies par MS/MS est au cœur des programmes de DNN à travers le monde [
14
,
15
].
L’extension des possibilités de dépistage aux maladies de surcharge lysosomale
Les maladies de surcharge lysosomale (MSL) sont un groupe de plus de 70 maladies héréditaires affectant le métabolisme du lysosome. Une grande majorité de ces maladies apparaissent dès la naissance et peuvent toucher la plupart des organes. Dès la fin des années 1990, des possibilités thérapeutiques sont apparues, principalement fondées sur le principe de l’enzymothérapie substitutive, complétée, par la suite, par des approches de réduction de substrat et, enfin, de thérapie génique [
16
]. Le diagnostic des MSL a longtemps reposé sur des mesures d’activités enzymatiques réalisées sur des leucocytes ou des lymphocytes, complétées par la quantification ou l’identification de biomarqueurs quand ceux-ci étaient disponibles. Comme dans le cas des autres classes de MHM, la MS/MS a fortement contribué à améliorer l’offre diagnostique pour ces maladies [
17
]. Les nombreux travaux de l’équipe de Michael Gelb (à Seattle, États-Unis) ont ainsi permis d’adapter les mesures d’activités enzymatiques lysosomales à la MS/MS réalisée à partir du sang déposé sur buvard, en utilisant des substrats spécifiques modifiés [
18
]. Une des grandes avancées de cette approche a été la possibilité de réaliser des analyses en multiplex, comme pour les maladies du métabolisme intermédiaire, et de combiner activité enzymatique et biomarqueurs quand cela s’avère nécessaire [
19
,
20
]. Devant ces possibilités, de nombreux pays ont mis en place des programmes de DNN des MSL, notamment pour la maladie de Pompe
3,
, la mucopolysaccharidose de type I (maladie de Hurler)
4,
ou la maladie de Fabry
5
[
21
]. Néanmoins, le DNN de ces maladies reste controversé, principalement en raison de problèmes éthiques liés à l’hétérogénéité du retentissement symptomatique de ces maladies sur les patients, et aux incertitudes sur les bénéfices à long terme des traitements actuellement disponibles [
22
,
23
].
Les autres possibilités offertes au DNN par la spectrométrie de masse
La place centrale prise par la spectrométrie de masse dans le DNN et sa grande polyvalence ont permis le développement de nombreux tests à partir du sang déposé sur buvard. Le dosage de C26:0-lysophosphatidylcholine (C26-LysoPC) par MS/MS permet le dépistage de l’adrénoleucodystrophie liée à l’X (ALD-X), une maladie peroxysomale qu’il est possible de traiter
6
. Le taux de C26-LysoPC peut facilement être évalué avec les acylcarnitines ou d’autres biomarqueurs, et quelques pays ont d’ores et déjà initié des programmes pilotes de DNN de l’ALD-X [
24
,
25
]. Le DNN de la drépanocytose profite également des possibilités offertes par cette technique, que ce soit la MS/MS elle-même, ou d’autres techniques, comme le MALDI-TOF, ou spectrométrie de masse par analyse de temps de vol (
Matrix-Analyzed Laser Desorption Ionisation – Time of Flight
) nouvellement déployée en microbiologie [
26
,
27
].
|
Les techniques de génétique moléculaire
Pendant de nombreuses années, les outils de génétique moléculaire ont été assez peu utilisés pour le DNN en raison de l’efficacité des techniques biochimiques qui étaient disponibles, mais aussi à cause d’une réglementation plus contraignante pour l’utilisation de ces outils d’analyse génétique. Néanmoins, depuis 2002, des tests génétiques sont utilisés dans l’organigramme du DNN de la mucoviscidose. Avec l’essor des nouvelles techniques de séquençage à haut débit (
next generation sequencing
, NGS), il est fort probable que ces techniques se développeront de façon importante à l’avenir.
Les contraintes liées à la règlementation
En France, l’utilisation des tests génétiques est très encadrée par la loi, notamment par le décret N° 2008-321 du 4 avril 2008 relatif à l’examen des caractéristiques génétiques d’une personne ou à son identification par empreintes génétiques à des fins médicales [
28
]. Dans le cadre du DNN, plusieurs points de cette réglementation posent des questions :
-
L’absence d’un prescripteur clairement identifié.
-
La difficulté de recueillir un consentement éclairé,
a priori
, pour des analyses génétiques qui ne seront pas systématiquement réalisées.
-
La nécessité pour les enfants mineurs, d’obtenir l’accord des deux parents pour réaliser un test génétique.
-
La complexité du rendu du résultat qui nécessite une consultation médicale dédiée à l’information.
Bien que des réponses aient pu être apportées à certaines de ces questions à la faveur de la mise en place du DNN de la mucoviscidose qui repose sur des analyses génétiques, cette réglementation reste peu adaptée pour l’utilisation des tests génétiques en population générale. Il est probable que des évolutions seront proposées dans le cadre des prochaines révisions des lois de bioéthique. Les techniques de génétique moléculaire actuellement utilisées L’amplification spécifique d’allèles
L’amplification spécifique d’allèles consiste à utiliser des amorces spécifiques de variants de gènes d’intérêt, afin de les amplifier par PCR (
polymerase chain reaction
) et de les révéler. Il est ainsi possible de mettre en évidence, de façon simple et rapide, la présence ou l’absence de variants pathogènes chez un individu. Cette méthode a été largement déployée pour le DNN de la mucoviscidose pour rechercher et identifier les variants fréquents du gène
CFTR
(
cystic fibrosis transmembrane conductance regulator
) responsables de la maladie. En France, c’est cette approche qui est actuellement utilisée [
29
].
La PCR quantitative La PCR quantitative, ou PCR en temps réel, est une technique classique qui permet d’évaluer la quantité initiale d’un fragment d’ADN ou d’une séquence dans un échantillon. Bien que peu utilisée dans l’univers du DNN, cette approche s’est révélée pertinente, très récemment, pour le dépistage de deux maladies : les déficits immunitaires combinés sévères (DICS, ou SCID en anglais) et l’amyotrophie spinale.
Dans le cas des déficits immunitaires combinés sévères, la technique utilisée permet la quantification des TREC (
T-cell receptor excision circles
) à partir du sang déposé sur le buvard. Les TREC sont les séquences d’ADN qui sont excisées lors des réarrangements que subit le récepteur des lymphocytes T (TCR) au cours de la thymopoïèse néonatale. Chez les nouveau-nés, les TREC qui sont produits peuvent être détectés dans les lymphocytes T circulants du sang périphérique. Chez les patients atteints de déficits immunitaires combinés sévères, l’absence ou un niveau bas de TREC dans ces cellules est un signe clinique pathognomonique de ces déficits [
30
].
Pour l’amyotrophie spinale, le dépistage peut être réalisé par la mise en évidence par PCR quantitative de la délétion de l’exon 7 du gène
SMN1
(
survival of motor neuron 1
), à l’origine de la maladie. La technique de PCR quantitative peut également être multiplexée avec la quantification des TREC vu précédemment [
31
].
Ces avancées technologiques, associées à la mise sur le marché de nouveaux traitements innovants, ont accéléré la mise en place de programmes de DNN pour ces deux maladies dans quelques pays. Il est vraisemblable que de nombreux autres pays les intègreront rapidement dans leur programme de santé publique [
32
,
33
].
|
Les nouveaux outils de séquençage à haut débit La dernière décennie a vu s’implanter une nouvelle technique de séquençage à haut débit dite de nouvelle génération (NGS) qui, couplée au progrès de la bio-informatique, a révolutionné la génétique moléculaire. Celle-ci est devenue un outil incontournable dans les laboratoires de biologie médicale, que ce soit pour la recherche de groupes de gènes d’intérêt, ou pour l’analyse d’exomes. De nombreux pays ont en effet mis en place des plateformes de séquençage à très haut débit avec pour objectif d’intégrer l’étude du génome entier dans la démarche diagnostique.
Rapidement, les acteurs du DNN se sont néanmoins posé la question des possibilités techniques et de l’utilité d’intégrer le séquençage haut débit dans les programmes et, au-delà de l’évaluation de la faisabilité technique, des projets de recherche ont permis d’avancer dans cette réflexion [
34
]. Le consensus actuel tend à limiter son utilisation en raison de la performance des biomarqueurs utilisés et des conséquences des découvertes génétiques incidentes qui sont inévitables. Néanmoins, bien que des équipes proposent d’utiliser cet outil en test de seconde ligne en intégrant, sous forme de panel, tous les gènes impliqués dans les maladies dépistées, sa place dans le DNN reste à définir [
35
-
37
].
|
Comme les autres domaines d’activités de la biologie clinique, le DNN suit les évolutions techniques et plusieurs secteurs devraient connaître des développements majeurs. Sur le plan pré-analytique, l’automatisation de la préparation des échantillons devrait se développer. Des techniques d’extraction directe des molécules d’intérêt à partir du buvard existent déjà pour la MS/MS et l’important développement de son automatisation va très probablement se poursuivre [
38
]. Au niveau analytique, au-delà de la place importante que devrait prendre le séquençage à haut débit, les nouvelles techniques de miniaturisation, telles que la microfluidique sur puce, devraient apporter des capacités d’analyse complémentaires à la MS/MS, notamment pour les mesures des activités enzymatiques. Un système récemment commercialisé (Seekers
®
) permet en effet de mesurer simultanément quatre activités enzymatiques lysosomales et d’évaluer l’activité de la biotinidase ou de la galactotransférase. Ce nouvel outil rapide et simple semble prometteur. Il est en cours d’évaluation dans le cadre du DNN [
39
].
L’augmentation du nombre de maladies dépistées engendre une quantité importante de données à interpréter. Les nouveaux outils post-analytiques utilisant l’intelligence artificielle apporteront une aide cruciale aux laboratoires de biologie médicale. Le CLIR (
Collaborative Laboratory Integrated Reports
), un outil spécifique du DNN utilisant la MS/MS et développé par la Mayo Clinic (Rochester, États-Unis), est déjà utilisé par certains États américains et par la Norvège [
40
]. D’autres initiatives pourraient voir le jour afin d’améliorer les valeurs prédictives positives des biomarqueurs utilisés.
|
Depuis la mise en place du dépistage néonatal (DNN) dans les années 1960, les évolutions technologiques ont toujours été le moteur de son développement. L’innovation du mode de prélèvement du sang déposé sur buvard a permis d’envisager un dépistage en population générale. L’immunoanalyse a démontré la pertinence de cette approche et l’apport de la spectrométrie de masse en tandem (MS/MS) a été majeur pour étendre de façon importante le dépistage à de nombreuses maladies. Globalement, nous pouvons dire qu’avec ces outils, tous les programmes de DNN ont atteint une forme de palier, malgré certaines divergences entre les pays. Néanmoins, il est très vraisemblable que l’essor extrêmement rapide des nouveaux outils moléculaires de séquençage du génome va forcer les acteurs et les décideurs à envisager un changement de paradigme et une extension du nombre de maladies génétiques dépistées. Pour autant, il faudra toujours se poser la question de savoir s’il faut tout dépister, la technologie le permettant, ou s’il faut se raisonner pour le bénéfice premier de l’enfant qui vient de naître. |
Les auteurs déclarent n’avoir aucun lien d’intérêt concernant les données publiées dans cet article.
|
Footnotes |
1.
Guthrie
R
,
Susi
A
.
A simple phenylalanine method for detecting phenylketonuria in large populations of newborn infants.
.
Pediatrics.
1963;
;
32
:
:338.
–
343
.
3.
Freeman
JD
,
Rosman
LM
,
Ratcliff
JD
,
et al.
State of the science in dried blood spots.
.
Clin Chem.
2018;
;
64
:
:656.
–
679
.
4.
Capiau
S
,
Veenhof
H
,
Koster
RA
et al.
Official international association for therapeutic drug monitoring and clinical toxicology guideline: development and validation of dried blood spot-based methods for therapeutic drug monitoring.
.
Ther Drug Monit.
2019;
;
41
:
:409.
–
430
.
5.
Moat
SJ
,
Dibden
C
,
Tetlow
L
,
et al.
Effect of blood volume on analytical bias in dried blood spots prepared for newborn screening external quality assurance.
.
Bioanalysis.
2020;
;
12
:
:99.
–
109
.
6.
Leuthold
LA
,
Heudi
O
,
Déglon
J
et al.
New microfluidic-based sampling procedure for overcoming the hematocrit problem associated with dried blood spot analysis.
.
Anal Chem.
2015;
;
87
:
:2068.
–
2071
.
7.
Sen
A
,
Gillett
M
,
Weaver
L
,
et al.
In vitro testing of the hemaPEN microsampling device for the quantification of acetaminophen in human blood.
.
Bioanalysis.
2020;
;
12
:
:1725.
–
37
.
8.
McCaman
M.
,
Robins
E
.
Fluorimetric method for the determination of phenylalanine in serum.
.
J Lab Clin Med.
1962;
;
59
:
:885.
.
9.
Millington
DS
.
The role of technology in newborn screening.
.
NC Med J.
2019;
;
80
:
:49.
–
53
.
10.
Frömmel
C
.
Newborn screening for sickle cell disease and other hemoglobinopathies: a short review on classical laboratory methods-isoelectric focusing, HPLC, and capillary electrophoresis.
.
Int J Neonatal Screen.
2018;
;
4
:
:39.
.
11.
Millington
DS
,
Kodo
N
,
Norwood
DL
,
Roe
CR
.
Tandem mass spectrometry: a new method for acylcarnitine profiling with potential for neonatal screening for inborn errors of metabolism.
.
J Inherit Metab Dis.
1990;
;
13
:
:321.
–
324
.
12.
Rashed
MS
,
Bucknall
MP
,
Little
D
et al.
Screening blood spots for inborn errors of metabolism by electrospray tandem mass spectrometry with a microplate batch process and a computer algorithm for automated flagging of abnormal profiles.
.
Clin Chem.
1997;
;
43
:
:1129.
–
1141
.
13.
Haute Autorité de Santé.
.
Évaluation a priori de l’extension du dépistage néonatal à une ou plusieurs erreurs innées du métabolisme par spectrométrie de masse en tandem.
. Volet 2.
Saint-Denis:
:
HAS;
,
2020
.
https://www.has-sante.fr/jcms/c_2866458/fr/
.
14.
Loeber
JG
,
Burgard
P
,
Cornel
MC
et al.
Newborn screening programmes in Europe. Arguments and efforts regarding harmonization. Part 1. From blood spot to screening result.
.
J Inherit Metab Dis.
2012;
;
35
:
:603.
–
611
.
15.
Martínez-Morillo
E
.
Prieto García B, Álvarez Menéndez FV. Challenges for worldwide harmonization of newborn screening programs.
.
Clin Chem.
2016;
;
62
:
:689.
–
698
.
16.
Platt
FM
,
d’Azzo
A
,
Davidson
BL
,
et al.
Lysosomal storage diseases.
.
Nat Rev Dis Primers.
2018;
;
4
:
:27.
.
17.
Piraud
M
,
Pettazzoni
M
,
Lavoie
P
et al.
Contribution of tandem mass spectrometry to the diagnosis of lysosomal storage disorders.
.
J Inherit Metab Dis.
2018;
;
41
:
:457.
–
477
.
18.
Gelb
MH
,
Lukacs
Z
,
Ranieri
E
,
Schielen
PCJI
.
Newborn screening for lysosomal storage disorders: methodologies for measurement of enzymatic activities in dried blood spots.
.
Int J Neonatal Screen.
2019;
;
5
:
:1.
.
19.
Spacil
Z
,
Tatipaka
H
,
Barcenas
M
et al.
High-throughput assay of 9 lysosomal enzymes for newborn screening.
.
Clin Chem.
2013;
;
59
:
:502.
–
511
.
20.
Burlina
AB
,
Polo
G
,
Rubert
L
et al.
Implementation of second-tier tests in newborn screening for lysosomal disorders in North eastern Italy.
.
Int J Neonatal Screen.
2019;
;
5
:
:24.
.
21.
Ames
EG
,
Fisher
R
,
Kleyn
M
,
Ahmad
A
.
Current practices for US newborn screening of pompe disease and MPSI.
.
Int J Neonatal Screen.
2020;
;
6
:
:72.
.
22.
Parini
R
,
Broomfield
A
,
Cleary
MA
et al.
International working group identifies need for newborn screening for mucopolysaccharidosis type I but states that existing hurdles must be overcome.
.
Acta Paediatr.
2018;
;
107
:
:2059.
–
2065
.
23.
Ehmann
P
,
Lantos
JD
.
Ethical issues with testing and treatment for Krabbe disease.
.
Dev Med Child Neurol.
2019;
;
61
:
:1358.
–
1361
.
24.
Lee
S
,
Clinard
K
,
Young
SP
,
et al.
Evaluation of X-linked adrenoleukodystrophy newborn screening in North Carolina.
.
JAMA Netw Open.
2020;
;
3
:
:e1920356.
.
25.
Barendsen
RW
,
Dijkstra
IME
,
Visser
WF
et al.
Adrenoleukodystrophy newborn screening in the Netherlands (SCAN study): The X-factor.
.
Front Cell Dev Biol.
2020;
;
17
:
:499.
.
26.
Daniel
Y
,
Turner
C
.
Newborn sickle cell disease screening using electrospray tandem mass spectrometry.
.
Int J Neonatal Screen.
2018;
24 ;
4
:
:35.
.
27.
Naubourg
P
,
El Osta
M
,
Rageot
D
et al.
A Multicentre pilot study of a two-tier newborn sickle cell disease screening procedure with a first tier based on a fully automated Maldi-Tof Ms platform.
.
Int J Neonatal Screen.
2019;
;
23
:
:10.
.
29.
Audrézet
MP
,
Munck
A
,
Scotet
V
et al.
Comprehensive CFTR gene analysis of the French cystic fibrosis screened newborn cohort: implications for diagnosis, genetic counseling, and mutation-specific therapy.
.
Genet Med.
2015;
;
17
:
:108.
–
116
.
30.
Thakar
MS
,
Hintermeyer
MK
,
Gries
MG
et al.
A Practical approach to newborn screening for severe combined immunodeficiency using the T cell receptor excision circle assay.
.
Front Immunol.
2017;
;
8
:
:1470.
.
31.
Taylor
JL
,
Lee
FK
,
Yazdanpanah
GK
et al.
Newborn blood spot screening test using multiplexed real-time PCR to simultaneously screen for spinal muscular atrophy and severe combined immunodeficiency.
.
Clin Chem.
2015;
;
61
:
:412.
–
419
.
32.
Boemer
F
,
Caberg
JH
,
Dideberg
V
et al.
Newborn screening for SMA in Southern Belgium.
.
Neuromuscul Disord.
2019;
;
29
:
:343.
–
349
.
33.
Audrain
MAP
,
Léger
AJC
,
Hémont
CAF
et al.
Newborn screening for severe combined immunodeficiency: analytic and clinical performance of the T cell receptor excision circle assay in France (Depistrec Study).
.
J Clin Immunol.
2018;
;
38
:
:778.
–
786
.
34.
Holm
IA
,
Agrawal
PB
,
Ceyhan-Birsoy
O
et al.
The BabySeq project: implementing genomic sequencing in newborns.
.
BMC Pediatr.
2018;
;
18
:
:225.
.
35.
Yamaguchi
T
,
Nakamura
A
,
Nakayama
K
,
et al.
Targeted next-generation sequencing for congenital hypothyroidism with positive neonatal TSH screening.
.
J Clin Endocrinol Metab.
2020;
;
105
:
:308.
.
36.
Adhikari
AN
,
Gallagher
RC
,
Wang
Y
,
et al.
The role of exome sequencing in newborn screening for inborn errors of metabolism.
.
Nat Med.
2020;
;
26
:
:1392.
–
7
.
37.
Morava
E
,
Baumgartner
M
,
Patterson
M
,
et al.
Newborn screening: to WES or not to WES, that is the question.
.
J Inherit Metab Dis.
2020;
;
43
:
:904.
–
5
.
38.
Luginbühl
M
,
Gaugler
S
.
The application of fully automated dried blood spot analysis for liquid chromatography-tandem mass spectrometry using the CAMAG DBS-MS 500 autosampler.
.
Clin Biochem.
2020;
;
82
:
:33.
–
9
.
39.
Sanders
KA
,
Gavrilov
DK
,
Oglesbee
D
,
et al.
A Comparative effectiveness study of newborn screening methods for four lysosomal storage disorders.
.
Int J Neonatal Screen.
2020;
;
6
:
:44.
.
40.
Tangeraas
T
,
Sæves
I
,
Klingenberg
C
,
et al.
Performance of expanded newborn screening in norway supported by post-analytical bioinformatics tools and rapid second-tier DNA analyses.
.
Int J Neonatal Screen.
2020;
;
6
:
:51.
.
41.
Farriaux
JP
.
Histoire et organisation du programme français de dépistage néonatal systématique (1967–2020).
.
Med Sci (Paris).
2021;
;
37
:
:433.
–
40
.
|